Окраска ветвей в дендрограмме в R
Уважаемый гений R резидент,
Я хотел бы раскрасить ветви кластера в дендрограмме, где листья не помечены.
Я нашел следующий скрипт здесь, в Stackru:
clusDendro <- as.dendrogram(Clustering)
labelColors <- c("red", "blue", "darkgreen", "darkgrey", "purple")
## function to get colorlabels
colLab <- function(n) {
if(is.leaf(n)) {
a <- attributes(n)
# clusMember - a vector designating leaf grouping
# labelColors - a vector of colors for the above grouping
labCol <- labelColors[clusMember[which(names(clusMember) == a$label)]]
attr(n, "nodePar") <- c(a$nodePar, lab.col = labCol)
}
n
}
## Graph
clusDendro <- dendrapply(clusDendro, colLab)
op <- par(mar = par("mar") + c(0,0,0,2))
plot(clusDendro,
main = "Major title",
horiz = T, type = "triangle", center = T)
par(op)
Я попытался адаптировать его к своим данным следующим образом, но безуспешно.
Gdis.UPGMA<-hclust(Gdis, method = "average", members=NULL)
k<-12
Gdiswo<-reorder.hclust(Gdis.UPGMA, Gdis, labels = FALSE)
cutg <- cutree(Gdiswo, k=k)
clusDendro <- as.dendrogram(Gdiswo)
labelColors <- c("red", "blue", "darkgreen", "darkgrey", "purple")
## function to get colorlabels
colLab <- function(n) {
if(is.leaf(n)) {
a <- attributes(n)
# cutg - a vector designating leaf grouping
# labelColors - a vector of colors for the above grouping
labCol <- labelColors[cutg[which(names(cutg) == a$label)]]
attr(n, "nodePar") <- c(a$nodePar, lab.col = labCol)
}
n
}
## Graph
clusDendro <- dendrapply(clusDendro, colLab)
op <- par(mar = par("mar") + c(0,0,0,2))
plot(clusDendro,
main = "Major title",
horiz = T, type = "triangle", center = T)
par(op)
Я подозреваю, что n вызывает проблему, но я не уверен, что я должен поставить вместо n. Поскольку сроки диссертации приближаются, я был бы очень благодарен за любой совет. Спасибо, Элизабет
2 ответа
Вам нужно установить edgePar элементы объекта дендрограммы.
В помощь для ?dendrapply Есть пример, чтобы установить цвета меток узла. Изменяя только одну строку, чтобы указать на "edgePar" и настройка col, Вы почти там:
attr(n, "edgePar") <- c(a$nodePar, list(col = mycols[i], lab.font= i%%3))
Полный модифицированный пример:
## a smallish simple dendrogram
dhc <- as.dendrogram(hc <- hclust(dist(USArrests), "ave"))
## toy example to set colored leaf labels :
local({
colLab <<- function(n) {
if(is.leaf(n)) {
a <- attributes(n)
i <<- i+1
attr(n, "edgePar") <-
c(a$nodePar, list(col = mycols[i], lab.font= i%%3))
}
n
}
mycols <- grDevices::rainbow(attr(dhc21,"members"))
i <- 0
})
dL <- dendrapply(dhc21, colLab)
plot(dL) ## --> colored labels
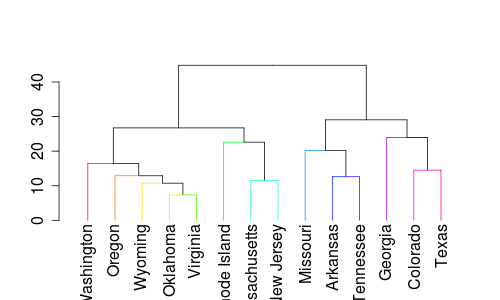
Вы можете прочитать все об этом, внимательно изучив ?dendrapply а также ?as.dendrogram
Просто для получения дополнительной информации, если вы хотите покрасить метки, измените edgePar на nodePar и используйте lab.col. Из-за узлов по умолчанию вам также нужно установить pch в NA, если вы хотите, чтобы все выглядело одинаково:
## a smallish simple dendrogram
dhc <- as.dendrogram(hc <- hclust(dist(USArrests), "ave"))
## create random colours for leaves based on a md5 hash of the leaf labels
library(digest);
dL <- dendrapply(dhc, function(n){
if(is.leaf(n)){
labelCol <- paste("#",substring(digest(attr(n,"label")),1,6), sep="");
attr(n, "edgePar") <- list(col = labelCol);
attr(n, "nodePar") <- list(pch = NA, lab.col = labelCol, lab.cex = 0.75);
}
n;
});
plot(dL); ## --> colored labels
