Rvest Web Scrape возвращает пустой символ
Я ищу, чтобы собрать некоторые данные из химической базы данных, используя R, в основном name, CAS Number, а также molecular weight теперь. Однако у меня возникли проблемы с получением rvest чтобы извлечь информацию, которую я ищу. Это код, который я до сих пор:
library(rvest)
library(magrittr)
# Read HTML code from website
# I am using this format because I ultimately hope to pull specific items from several different websites
webpage <- read_html(paste0("https://pubchem.ncbi.nlm.nih.gov/compound/", 1))
# Use CSS selectors to scrape the chemical name
chem_name_html <- webpage %>%
html_nodes(".short .breakword") %>%
html_text()
# Convert the data to text
chem_name_data <- html_text(chem_name_html)
Тем не менее, когда я пытаюсь создать name_html, R возвращает только символ (пустой). я использую SelectorGadget чтобы получить узел HTML, но я заметил, что SelectorGadget дает мне другой узел, чем то, что делает инспектор в Google Chrome. Я пробовал оба ".short .breakword" а также ".summary-title short .breakword" в этой строке кода, но ни один не дает мне то, что я ищу.
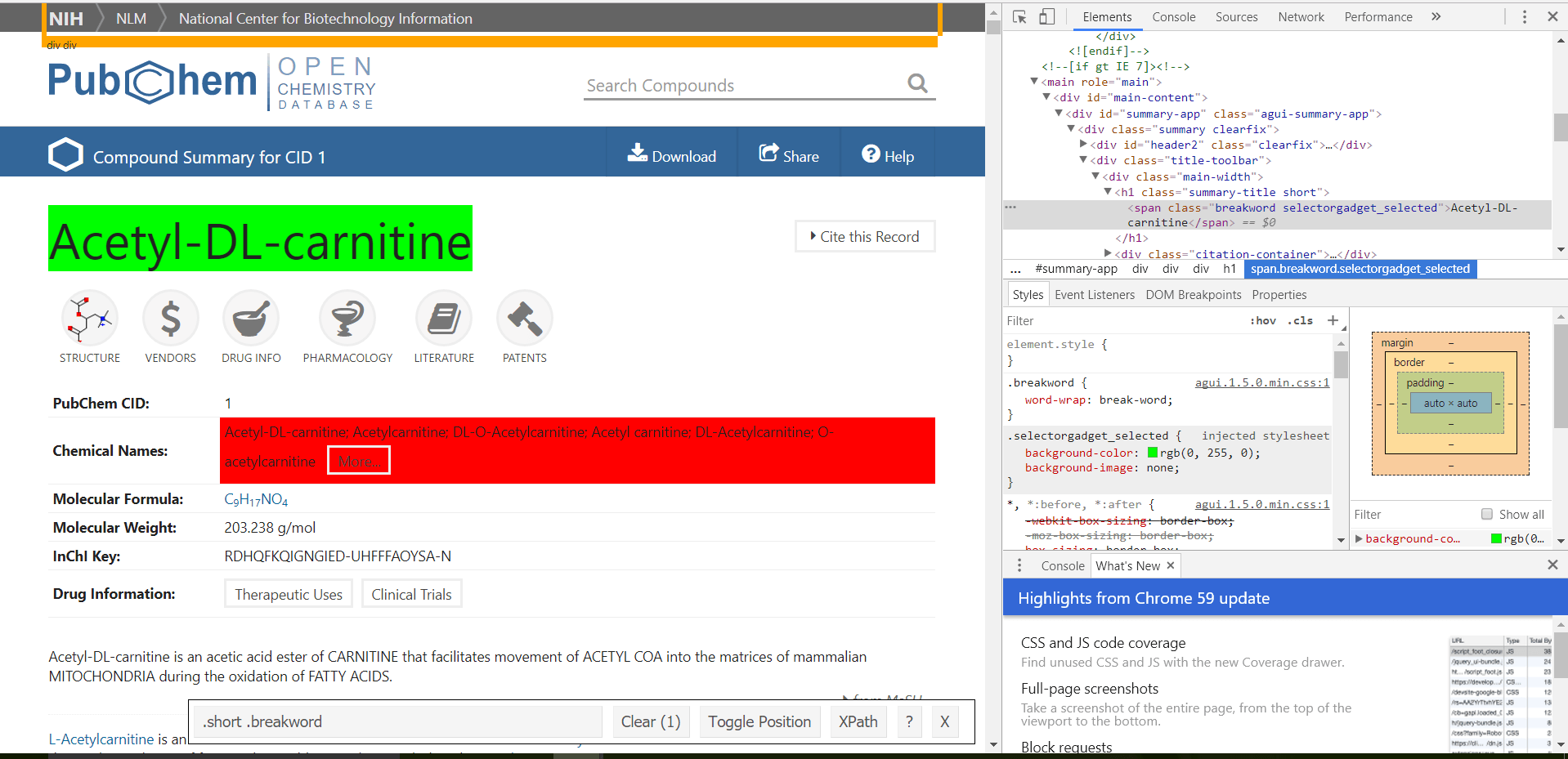
1 ответ
Я недавно столкнулся с теми же проблемами, используя rvest скрести ПабХим. Проблема в том, что информация на странице отображается с использованием javascript при прокрутке страницы вниз, поэтому rvest получает только минимальную информацию со страницы.
Есть несколько обходных путей, хотя. Самый простой способ получить нужную информацию в R - это использовать пакет R под названием webchem,
Если вы ищете имя, номер CAS и молекулярный вес, то вы можете сделать что-то вроде:
library(webchem)
chem_properties <- pc_prop(1, properties = c('IUPACName', 'MolecularWeight'))
Полный список составных свойств, которые можно извлечь с помощью этого API, можно найти здесь. К сожалению, через этот API нет свойства для получения номера CAS, но webchem дает нам еще один способ запросить это с помощью службы химического перевода.
chem_cas <- cts_convert(query = '1', from = 'CID', to = 'CAS')
Второй способ получить информацию со страницы, которая немного более надежна, но с которой не так просто работать, - это получить информацию из API JSON.
library(jsonlite)
chem_json <-
read_json(paste0("https://pubchem.ncbi.nlm.nih.gov/rest/pug_view/data/compound/", "1", "/JSON/?response_type=save$response_basename=CID_", "1"))
С помощью этой команды вы получите список списков, в котором мне пришлось написать сложный скрипт, чтобы разобрать необходимую мне информацию со страницы. Если вы знакомы с JSON, вы можете анализировать гораздо больше информации со страницы, но не все. Например, в таких разделах, как Литература, Патенты и Биомолекулярные взаимодействия и пути, информация в этих разделах не будет полностью отображаться в информации JSON.
Последний и наиболее полный способ получить всю информацию со страницы - использовать что-то вроде Scrapy или PhantomJS, чтобы отобразить полный html-вывод страницы PubChem, а затем использовать rvest поцарапать это, как вы изначально хотели. Это то, над чем я все еще работаю, так как я впервые использую веб-скребки.
Я все еще новичок в этой области, но, надеюсь, это вам немного поможет.