Вложенная Anova в python с помощью Spm1d. Невозможно распечатать статистику f и значения p
Я ищу простое решение для выполнения многофакторного анализа ANOVA в Python. Мне нужна двухфакторная вложенная ANOVA, и Python-модуль SPM1D - один из способов сделать это, однако у меня возникла проблема.
http://www.spm1d.org/doc/Stats1D/anova.html
для любого из вложенных примеров подходов никогда не печатается F-статистика или p_values, и я не могу найти какой-либо способ напечатать их или отправить их в переменную.
Чтобы пройти через ход выполнения одного из их примеров, где B вложен в A, с Y наблюдениями:
import numpy as np
from matplotlib import pyplot
import spm1d
dataset = spm1d.data.uv1d.anova2nested.SPM1D_ANOVA2NESTED_3x3()
Y,A,B = dataset.get_data()
#(1) Conduct ANOVA:
alpha = 0.05
FF = spm1d.stats.anova2nested(Y, A, B, equal_var=True)
FFi = FF.inference(0.05)
print( FFi )
#(2) Plot results:
pyplot.close('all')
FFi.plot(plot_threshold_label=True, plot_p_values=True)
pyplot.show()
Единственным указанием на статистическую значимость является то, отклонена ли гипотеза h0 или нет.
> print( FFi )
SPM{F} inference list
design : ANOVA2nested
nEffects : 2
Effects:
A z=(1x101) array df=(2, 6) h0reject=True
B z=(1x101) array df=(6, 36) h0reject=False
На самом деле этого должно быть достаточно. Однако в науке ученым нравится думать о чем-то как о более или менее значительном, что на самом деле является дерьмом... значение является бинарным. Но вот как они думают об этом, поэтому я должен подыграть, чтобы опубликовать работу.
Пример кода создает график matplotlib, и это ДОЛЖНО иметь статистику f и p_values!
#(2) Plot results:
pyplot.close('all')
FFi.plot(plot_threshold_label=True, plot_p_values=True)
pyplot.show()
 Но я не могу получить какой-либо вывод, который его печатает.
Но я не могу получить какой-либо вывод, который его печатает.
FFi.get_p_values
а также
FFi.get_f_values
произвести вывод:
<bound method SPMFiList.get_p_values <kabammi edit -- or get_f_values> of SPM{F} inference list
design : ANOVA2nested
nEffects : 2
Effects:
A z=(1x101) array df=(2, 6) h0reject=True
B z=(1x101) array df=(6, 36) h0reject=False
Так что я не знаю, что делать. Очевидно, класс FFi.plot может получить доступ к p_values (с plot_p_values), но FFi.get_p_values не может!!? Кто-нибудь может протянуть руку?
ура, К
1 ответ
Самый простой способ получить значения p - это использовать get_p_values метод, который вы упоминаете, вам просто нужно вызвать метод, добавив () к концу.
p = FFi.get_p_values()
print(p)
Это дает:
([0.016584151119287904], [])
Чтобы увидеть более подробную информацию о каждом эффекте в 2+-ходовом ANOVA, включая значения p, используйте print наряду с индивидуальной статистикой F, как это:
print( FFi[0] )
print( FFi[1] )
Первый оператор print выдаст вывод примерно так:
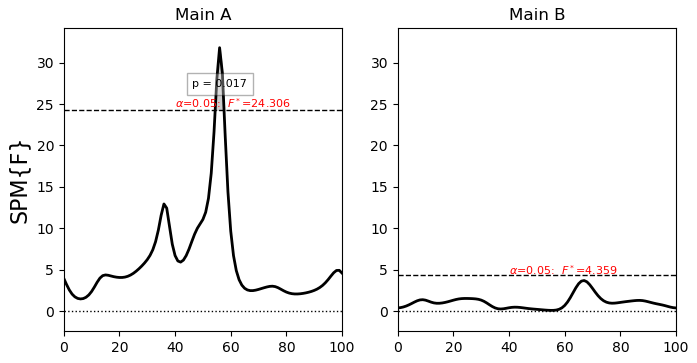
SPM{F} inference field
SPM.effect : Main A
SPM.z : (1x101) raw test stat field
SPM.df : (2, 6)
SPM.fwhm : 11.79254
SPM.resels : (1, 8.47993)
Inference:
SPM.alpha : 0.050
SPM.zstar : 24.30619
SPM.h0reject : True
SPM.p_set : 0.017
SPM.p_cluster : (0.017)
Вы можете получить значения p кластеров следующим образом:
p = [F.p for F in FFi]
который дает тот же результат, что и вызов get_p_values,
Обратите внимание, что в этом случае нет значений p для FFi[1] потому что тестовая статистика не может пересечь alpha-определенный порог (см. панель "Основная B" на рисунке выше). Если вам также необходимо сообщить значения p в этом случае, один из вариантов - просто использовать "p > alpha". Более точное значение p доступно параметрически вплоть до p = 0,5, но большие значения p не очень точны при использовании параметрических методов, поэтому, если вам нужны значения p для всех случаев, рассмотрите возможность использования непараметрической версии: spm1d.stats.nonparam.anova2nested,