Нелинейные наименьшие квадраты в R - Левенберга Марквардта для соответствия параметрам модели Хелигмана Полларда
Я пытаюсь воспроизвести бумажные решения Костаки. В этой статье сокращенная таблица смертности расширена до полной таблицы жизни с использованием модели де Хелигмана-Полларда. Модель имеет 8 параметров, которые должны быть установлены. Автор использовал модифицированный алгоритм Гаусса-Ньютона; этот алгоритм (E04FDF) является частью библиотеки компьютерных программ NAG. Не следует ли Левенбергу Марквардту дать такой же набор параметров? Что не так с моим кодом или применением алгоритма LM?
library(minpack.lm)
## Heligman-Pollard is used to expand an abridged table.
## nonlinear least squares algorithm is used to fit the parameters on nqx observed over 5 year intervals (5qx)
AGE <- c(0, 5, 10, 15, 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70)
MORTALITY <- c(0.010384069, 0.001469140, 0.001309318, 0.003814265, 0.005378395, 0.005985625, 0.006741766, 0.009325056, 0.014149626, 0.021601755, 0.034271934, 0.053836246, 0.085287751, 0.136549522, 0.215953304)
## The start parameters for de Heligman-Pollard Formula (Converged set a=0.0005893,b=0.0043836,c=0.0828424,d=0.000706,e=9.927863,f=22.197312,g=0.00004948,h=1.10003)
## I modified a random parameter "a" in order to have a start values. The converged set is listed above.
parStart <- list(a=0.0008893,b=0.0043836,c=0.0828424,d=0.000706,e=9.927863,f=22.197312,g=0.00004948,h=1.10003)
## The Heligman-Pollard Formula (HP8) = qx/px = ...8 parameter equation
HP8 <-function(parS,x)
ifelse(x==0, parS$a^((x+parS$b)^parS$c) + parS$g*parS$h^x,
parS$a^((x+parS$b)^parS$c) + parS$d*exp(-parS$e*(log(x/parS$f))^2) +
parS$g*parS$h^x)
## Define qx = HP8/(1+HP8)
qxPred <- function(parS,x) HP8(parS,x)/(1+HP8(parS,x))
## Calculate nqx predicted by HP8 model (nqxPred(parStart,x))
nqxPred <- function(parS,x)
(1 -(1-qxPred(parS,x)) * (1-qxPred(parS,x+1)) *
(1-qxPred(parS,x+2)) * (1-qxPred(parS,x+3)) *
(1-qxPred(parS,x+4)))
##Define Residual Function, the relative squared distance is minimized
ResidFun <- function(parS, Observed,x) (nqxPred(parS,x)/Observed-1)^2
## Applying the nls.lm algo.
nls.out <- nls.lm(par=parStart, fn = ResidFun, Observed = MORTALITY, x = AGE,
control = nls.lm.control(nprint=1,
ftol = .Machine$double.eps,
ptol = .Machine$double.eps,
maxfev=10000, maxiter = 500))
summary(nls.out)
## The author used a modified Gauss-Newton algorithm, this alogorithm (E04FDF) is part of the NAG library of computer programs
## Should not Levenberg Marquardt yield the same set of parameters
2 ответа
Суть в том, что @Roland абсолютно прав, это очень некорректная проблема, и вы не обязательно должны ожидать надежных ответов. Ниже я
- очистил код несколькими небольшими способами (это просто эстетично)
- изменил
ResidFunвозвращать остатки, а не квадратные остатки. (Первое верно, но это не имеет большого значения.) - исследовал результаты нескольких разных оптимизаторов. Похоже, что ответ, который вы получаете, лучше, чем перечисленные выше "конвергентные параметры", которые, как я предполагаю, являются параметрами из исходного исследования (не могли бы вы дать ссылку?).
Загрузить пакет:
library(minpack.lm)
Данные, как фрейм данных:
d <- data.frame(
AGE = seq(0,70,by=5),
MORTALITY=c(0.010384069, 0.001469140, 0.001309318, 0.003814265,
0.005378395, 0.005985625, 0.006741766, 0.009325056,
0.014149626, 0.021601755, 0.034271934, 0.053836246,
0.085287751, 0.136549522, 0.215953304))
Первый просмотр данных:
library(ggplot2)
(g1 <- ggplot(d,aes(AGE,MORTALITY))+geom_point())
g1+geom_smooth() ## with loess fit
Выбор параметров:
Предположительно это параметры из оригинальной статьи...
parConv <- c(a=0.0005893,b=0.0043836,c=0.0828424,
d=0.000706,e=9.927863,f=22.197312,g=0.00004948,h=1.10003)
Возмущенные параметры:
parStart <- parConv
parStart["a"] <- parStart["a"]+3e-4
Формулы:
HP8 <-function(parS,x)
with(as.list(parS),
ifelse(x==0, a^((x+b)^c) + g*h^x,
a^((x+b)^c) + d*exp(-e*(log(x/f))^2) + g*h^x))
## Define qx = HP8/(1+HP8)
qxPred <- function(parS,x) {
h <- HP8(parS,x)
h/(1+h)
}
## Calculate nqx predicted by HP8 model (nqxPred(parStart,x))
nqxPred <- function(parS,x)
(1 -(1-qxPred(parS,x)) * (1-qxPred(parS,x+1)) *
(1-qxPred(parS,x+2)) * (1-qxPred(parS,x+3)) *
(1-qxPred(parS,x+4)))
##Define Residual Function, the relative squared distance is minimized
ResidFun <- function(parS, Observed,x) (nqxPred(parS,x)/Observed-1)
nb это немного изменилось по сравнению с версией ОП; nls.lm хочет остатки, а не квадрат остатков.
Функция суммы квадратов для использования с другими оптимизаторами:
ssqfun <- function(parS, Observed, x) {
sum(ResidFun(parS, Observed, x)^2)
}
применение nls.lm, (Не уверен почему ftol а также ptol были понижены от sqrt(.Machine$double.eps) в .Machine$double.eps - первое, как правило, практическое ограничение точности...
nls.out <- nls.lm(par=parStart, fn = ResidFun,
Observed = d$MORTALITY, x = d$AGE,
control = nls.lm.control(nprint=0,
ftol = .Machine$double.eps,
ptol = .Machine$double.eps,
maxfev=10000, maxiter = 1000))
parNLS <- coef(nls.out)
pred0 <- nqxPred(as.list(parConv),d$AGE)
pred1 <- nqxPred(as.list(parNLS),d$AGE)
dPred <- with(d,rbind(data.frame(AGE,MORTALITY=pred0,w="conv"),
data.frame(AGE,MORTALITY=pred1,w="nls")))
g1 + geom_line(data=dPred,aes(colour=w))
Строки неразличимы, но параметры имеют некоторые большие различия:
round(cbind(parNLS,parConv),5)
## parNLS parConv
## a 1.00000 0.00059
## b 50.46708 0.00438
## c 3.56799 0.08284
## d 0.00072 0.00071
## e 6.05200 9.92786
## f 21.82347 22.19731
## g 0.00005 0.00005
## h 1.10026 1.10003
d, f, g, h близки, но a, b, c отличаются на порядки, а e на 50% отличается.
Глядя на исходные уравнения, здесь происходит то, что a^((x+b)^c) устанавливается на постоянную, потому что a приближается 1: один раз a примерно 1, b а также c по сути не имеют значения.
Давайте проверим корреляцию (нам нужно обобщенное обратное, потому что матрица очень сильно коррелирует):
obj <- nls.out
vcov <- with(obj,deviance/(length(fvec) - length(par)) *
MASS::ginv(hessian))
cmat <- round(cov2cor(vcov),1)
dimnames(cmat) <- list(letters[1:8],letters[1:8])
## a b c d e f g h
## a 1.0 0.0 0.0 0.0 0.0 0.0 -0.1 0.0
## b 0.0 1.0 -1.0 1.0 -1.0 -1.0 -0.4 -1.0
## c 0.0 -1.0 1.0 -1.0 1.0 1.0 0.4 1.0
## d 0.0 1.0 -1.0 1.0 -1.0 -1.0 -0.4 -1.0
## e 0.0 -1.0 1.0 -1.0 1.0 1.0 0.4 1.0
## f 0.0 -1.0 1.0 -1.0 1.0 1.0 0.4 1.0
## g -0.1 -0.4 0.4 -0.4 0.4 0.4 1.0 0.4
## h 0.0 -1.0 1.0 -1.0 1.0 1.0 0.4 1.0
Это на самом деле не так полезно - это просто подтверждает, что многие переменные сильно коррелированы...
library(optimx)
mvec <- c('Nelder-Mead','BFGS','CG','L-BFGS-B',
'nlm','nlminb','spg','ucminf')
opt1 <- optimx(par=parStart, fn = ssqfun,
Observed = d$MORTALITY, x = d$AGE,
itnmax=5000,
method=mvec,control=list(kkt=TRUE))
## control=list(all.methods=TRUE,kkt=TRUE)) ## Boom!
## fvalues method fns grs itns conv KKT1 KKT2 xtimes
## 2 8.988466e+307 BFGS NA NULL NULL 9999 NA NA 0
## 3 8.988466e+307 CG NA NULL NULL 9999 NA NA 0
## 4 8.988466e+307 L-BFGS-B NA NULL NULL 9999 NA NA 0
## 5 8.988466e+307 nlm NA NA NA 9999 NA NA 0
## 7 0.3400858 spg 1 NA 1 3 NA NA 0.064
## 8 0.3400858 ucminf 1 1 NULL 0 NA NA 0.032
## 1 0.06099295 Nelder-Mead 501 NA NULL 1 NA NA 0.252
## 6 0.009275733 nlminb 200 1204 145 1 NA NA 0.708
Это предупреждает о плохом масштабировании, а также находит множество разных ответов: только ucminf утверждает, что сошлись, но nlminb получает лучший ответ - и itnmax параметр, кажется, игнорируется...
opt2 <- nlminb(start=parStart, objective = ssqfun,
Observed = d$MORTALITY, x = d$AGE,
control= list(eval.max=5000,iter.max=5000))
parNLM <- opt2$par
Завершает, но с ложным предупреждением о сходимости...
round(cbind(parNLS,parConv,parNLM),5)
## parNLS parConv parNLM
## a 1.00000 0.00059 1.00000
## b 50.46708 0.00438 55.37270
## c 3.56799 0.08284 3.89162
## d 0.00072 0.00071 0.00072
## e 6.05200 9.92786 6.04416
## f 21.82347 22.19731 21.82292
## g 0.00005 0.00005 0.00005
## h 1.10026 1.10003 1.10026
sapply(list(parNLS,parConv,parNLM),
ssqfun,Observed=d$MORTALITY,x=d$AGE)
## [1] 0.006346250 0.049972367 0.006315034
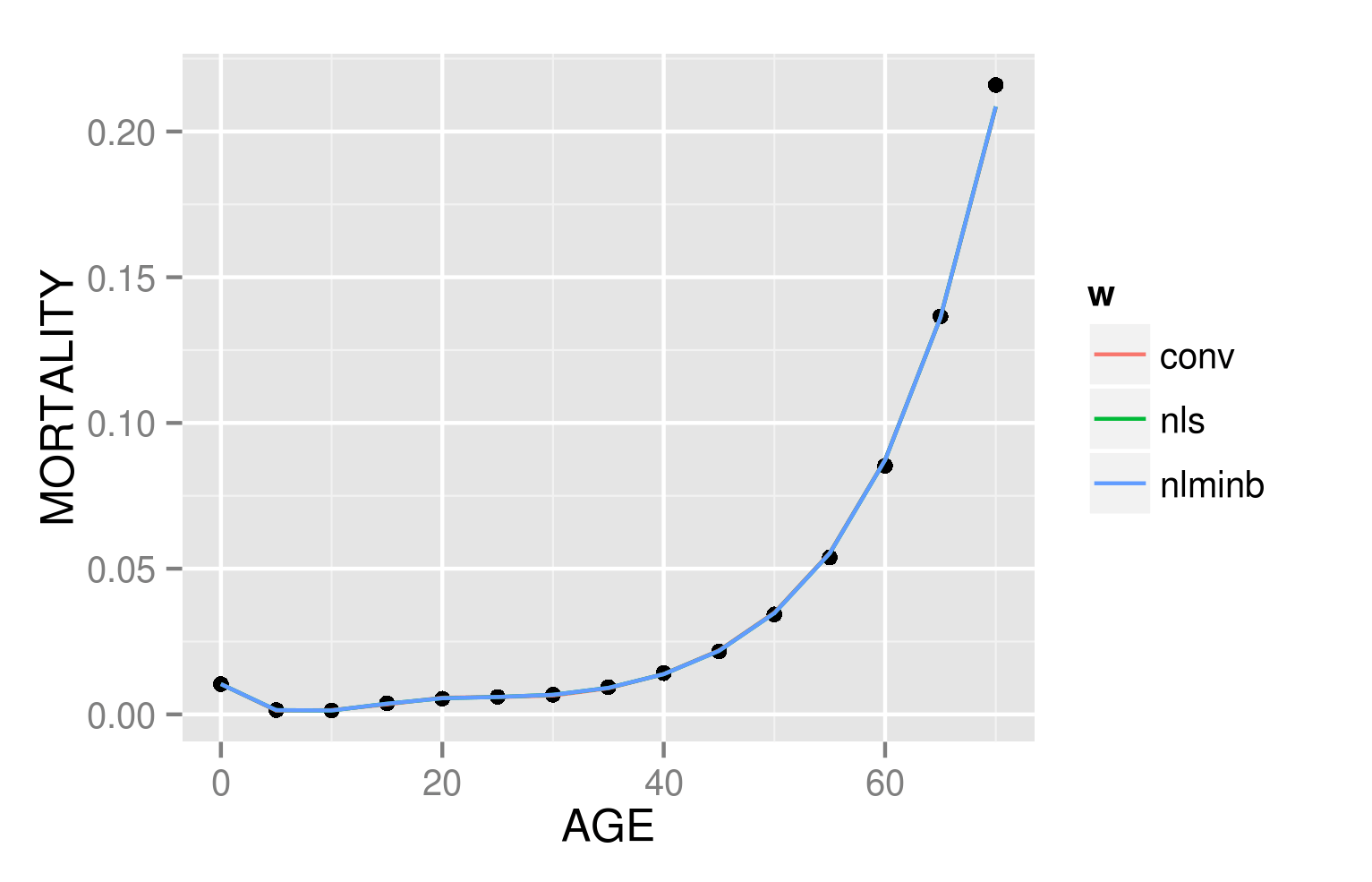
Это выглядит как nlminb а также minpack.lm получают аналогичные ответы, и на самом деле работают лучше, чем первоначально заявленные параметры (совсем немного):
pred2 <- nqxPred(as.list(parNLM),d$AGE)
dPred <- with(d,rbind(dPred,
data.frame(AGE,MORTALITY=pred2,w="nlminb")))
g1 + geom_line(data=dPred,aes(colour=w))
ggsave("cmpplot.png")
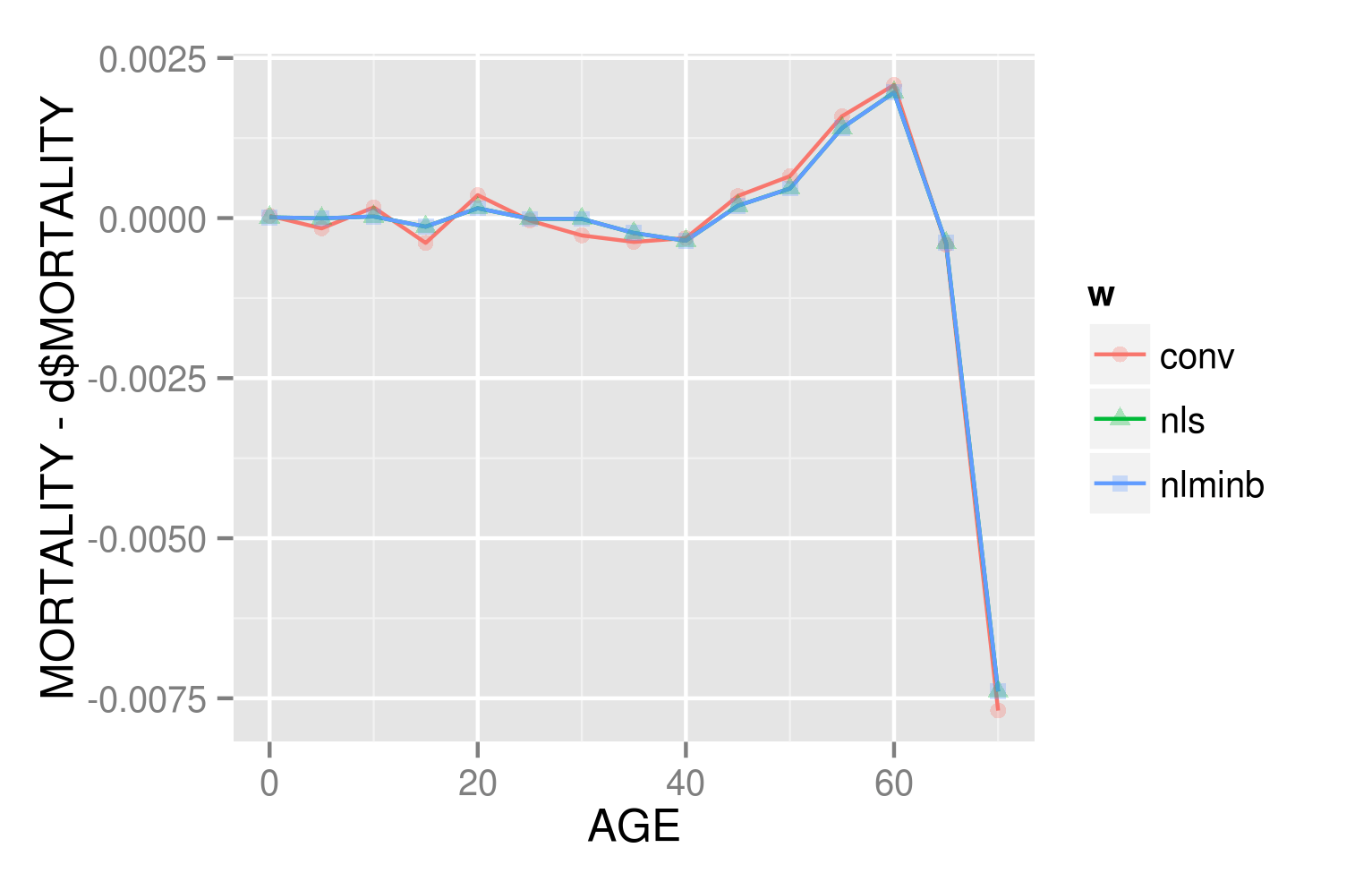
ggplot(data=dPred,aes(x=AGE,y=MORTALITY-d$MORTALITY,colour=w))+
geom_line()+geom_point(aes(shape=w),alpha=0.3)
ggsave("residplot.png")
Другие вещи можно было бы попробовать:
- соответствующее масштабирование - хотя быстрая проверка этого не очень помогает
- предоставить аналитические градиенты
- использовать AD Model Builder
- использовать
sliceфункция отbbmleчтобы выяснить, представляют ли старые и новые параметры разные минимумы или старые параметры являются просто ложной сходимостью... - получить калькуляторы критериев KKT (Karsh-Kuhn-Tucker) от
optimxили связанные пакеты, работающие для аналогичных проверок
PS: наибольшие отклонения (безусловно) относятся к самым старым возрастным классам, которые, вероятно, также имеют небольшие выборки. С статистической точки зрения, вероятно, стоило бы сделать подбор, взвешенный с точностью до отдельных точек...
@BenBolker, подгонка параметров ко всем значениям набора данных (лежащих в основе qx). Все еще не в состоянии воспроизвести параметры
library(minpack.lm)
library(ggplot2)
library(optimx)
getwd()
d <- data.frame(AGE = seq(0,74), MORTALITY=c(869,58,40,37,36,35,32,28,29,23,24,22,24,28,
33,52,57,77,93,103,103,109,105,114,108,112,119,
125,117,127,125,134,134,131,152,179,173,182,199,
203,232,245,296,315,335,356,405,438,445,535,594,
623,693,749,816,915,994,1128,1172,1294,1473,
1544,1721,1967,2129,2331,2559,2901,3203,3470,
3782,4348,4714,5245,5646))
d$MORTALITY <- d$MORTALITY/100000
ggplot(d,aes(AGE,MORTALITY))+geom_point()
##Not allowed to post Images
g1 <- ggplot(d,aes(AGE,MORTALITY))+geom_point()
g1+geom_smooth()## with loess fit
Сообщаемые параметры:
parConv <- c(a=0.0005893,b=0.0043836,c=0.0828424,d=0.000706,e=9.927863,f=22.197312,
g=0.00004948,h=1.10003)
parStart <- parConv
parStart["a"] <- parStart["a"]+3e-4
## Define qx = HP8/(1+HP8)
HP8 <-function(parS,x)
with(as.list(parS),
ifelse(x==0, a^((x+b)^c) + g*h^x, a^((x+b)^c) + d*exp(-e*(log(x/f))^2) + g*h^x))
qxPred <- function(parS,x) {
h <- HP8(parS,x)
h/(1+h)
}
##Define Residual Function, the relative squared distance is minimized,
ResidFun <- function(parS, Observed,x) (qxPred(parS,x)/Observed-1)
ssqfun <- function(parS, Observed, x) {
sum(ResidFun(parS, Observed, x)^2)
}
nls.out <- nls.lm(par=parStart, fn = ResidFun, Observed = d$MORTALITY, x = d$AGE,
control = nls.lm.control(nprint=1, ftol = sqrt(.Machine$double.eps),
ptol = sqrt(.Machine$double.eps), maxfev=1000, maxiter=1000))
parNLS <- coef(nls.out)
pred0 <- qxPred(as.list(parConv),d$AGE)
pred1 <- qxPred(as.list(parNLS),d$AGE)
#Binds Row wise the dataframes from pred0 and pred1
dPred <- with(d,rbind(data.frame(AGE,MORTALITY=pred0,w="conv"),
data.frame(AGE,MORTALITY=pred1,w="nls")))
g1 + geom_line(data=dPred,aes(colour=w))
round(cbind(parNLS,parConv),7)
mvec <- c('Nelder-Mead','BFGS','CG','L-BFGS-B','nlm','nlminb','spg','ucminf')
opt1 <- optimx(par=parStart, fn = ssqfun,
Observed = d$MORTALITY, x = d$AGE,
itnmax=5000,
method=mvec, control=list(all.methods=TRUE,kkt=TRUE,)
## control=list(all.methods=TRUE,kkt=TRUE)) ## Boom
get.result(opt1, attribute= c("fvalues","method", "grs", "itns",
"conv", "KKT1", "KKT2", "xtimes"))
## method fvalues grs itns conv KKT1 KKT2 xtimes
##5 nlm 8.988466e+307 NA NA 9999 NA NA 0
##4 L-BFGS-B 8.988466e+307 NULL NULL 9999 NA NA 0
##2 CG 8.988466e+307 NULL NULL 9999 NA NA 0.02
##1 BFGS 8.988466e+307 NULL NULL 9999 NA NA 0
##3 Nelder-Mead 0.5673864 NA NULL 0 NA NA 0.42
##6 nlminb 0.4127198 546 62 0 NA NA 0.17
opt2 <- nlminb(start=parStart, objective = ssqfun,
Observed = d$MORTALITY, x = d$AGE,
control= list(eval.max=5000,iter.max=5000))
parNLM <- opt2$par
Проверка по параметрам:
round(cbind(parNLS,parConv,parNLM),5)
## parNLS parConv parNLM
##a 0.00058 0.00059 0.00058
##b 0.00369 0.00438 0.00369
##c 0.08065 0.08284 0.08065
##d 0.00070 0.00071 0.00070
##e 9.30948 9.92786 9.30970
##f 22.30769 22.19731 22.30769
##g 0.00005 0.00005 0.00005
##h 1.10084 1.10003 1.10084
Обзор SSE:
sapply(list(parNLS,parConv,parNLM),
ssqfun,Observed=d$MORTALITY,x=d$AGE)
##[1] 0.4127198 0.4169513 0.4127198
Невозможно загрузить графики, но код здесь. Тем не менее, кажется, что параметры, найденные в статье, не подходят лучше всего, когда используются полные данные о смертности (не сокращенные или подмножество)
##pred2 <- qxPred(as.list(parNLM),d$AGE)
##dPred <- with(d,rbind(dPred,
data.frame(AGE,MORTALITY=pred2,w="nlminb")))
##g1 + geom_line(data=dPred,aes(colour=w))
ggplot(data=dPred,aes(x=AGE,y=MORTALITY-d$MORTALITY,colour=w))
+ geom_line()+geom_point(aes(shape=w),alpha=0.3)