Как заменить значения NA нулями в кадре данных R?
У меня есть фрейм данных и некоторые столбцы NA ценности.
Как мне заменить эти NA значения с нулями?
30 ответов
Смотрите мой комментарий в ответе @gsk3. Простой пример:
> m <- matrix(sample(c(NA, 1:10), 100, replace = TRUE), 10)
> d <- as.data.frame(m)
V1 V2 V3 V4 V5 V6 V7 V8 V9 V10
1 4 3 NA 3 7 6 6 10 6 5
2 9 8 9 5 10 NA 2 1 7 2
3 1 1 6 3 6 NA 1 4 1 6
4 NA 4 NA 7 10 2 NA 4 1 8
5 1 2 4 NA 2 6 2 6 7 4
6 NA 3 NA NA 10 2 1 10 8 4
7 4 4 9 10 9 8 9 4 10 NA
8 5 8 3 2 1 4 5 9 4 7
9 3 9 10 1 9 9 10 5 3 3
10 4 2 2 5 NA 9 7 2 5 5
> d[is.na(d)] <- 0
> d
V1 V2 V3 V4 V5 V6 V7 V8 V9 V10
1 4 3 0 3 7 6 6 10 6 5
2 9 8 9 5 10 0 2 1 7 2
3 1 1 6 3 6 0 1 4 1 6
4 0 4 0 7 10 2 0 4 1 8
5 1 2 4 0 2 6 2 6 7 4
6 0 3 0 0 10 2 1 10 8 4
7 4 4 9 10 9 8 9 4 10 0
8 5 8 3 2 1 4 5 9 4 7
9 3 9 10 1 9 9 10 5 3 3
10 4 2 2 5 0 9 7 2 5 5
Там нет необходимости применять apply, знак равно
РЕДАКТИРОВАТЬ
Вы также должны взглянуть на norm пакет. Он имеет много приятных возможностей для анализа отсутствующих данных. знак равно
Гибридный вариант dplyr/Base R: mutate_all(funs(replace(., is.na(.), 0)))) более чем в два раза быстрее, чем база R d[is.na(d)] <- 0 вариант. (см. анализ производительности ниже.)
Если вы боретесь с большими массивами данных, data.table это самый быстрый вариант из всех: на 30% меньше времени, чем у dplyr, и в 3 раза быстрее, чем у Base R. Он также изменяет данные на месте, эффективно позволяя работать с почти вдвое большим количеством данных одновременно.
Кластеризация других полезных подходов замены Tidyverse
Locationally:
- индекс
mutate_at(c(5:10), funs(replace(., is.na(.), 0))) - прямая ссылка
mutate_at(vars(var5:var10), funs(replace(., is.na(.), 0))) - фиксированный матч
mutate_at(vars(contains("1")), funs(replace(., is.na(.), 0)))- или вместо
contains(), пытатьсяends_with(),starts_with()
- или вместо
- образец соответствия
mutate_at(vars(matches("\\d{2}")), funs(replace(., is.na(.), 0)))
Условно:
(измените только число (столбцы) и оставьте строку (столбцы) в покое.)
- целые
mutate_if(is.integer, funs(replace(., is.na(.), 0))) - двойники
mutate_if(is.numeric, funs(replace(., is.na(.), 0))) - строки
mutate_if(is.character, funs(replace(., is.na(.), 0)))
Полный анализ -
Подходы проверены:
# Base R:
baseR.sbst.rssgn <- function(x) { x[is.na(x)] <- 0; x }
baseR.replace <- function(x) { replace(x, is.na(x), 0) }
baseR.for <- function(x) { for(j in 1:ncol(x))
x[[j]][is.na(x[[j]])] = 0 }
# tidyverse
## dplyr
library(tidyverse)
dplyr_if_else <- function(x) { mutate_all(x, funs(if_else(is.na(.), 0, .))) }
dplyr_coalesce <- function(x) { mutate_all(x, funs(coalesce(., 0))) }
## tidyr
tidyr_replace_na <- function(x) { replace_na(x, as.list(setNames(rep(0, 10), as.list(c(paste0("var", 1:10)))))) }
## hybrid
hybrd.ifelse <- function(x) { mutate_all(x, funs(ifelse(is.na(.), 0, .))) }
hybrd.rplc_all <- function(x) { mutate_all(x, funs(replace(., is.na(.), 0))) }
hybrd.rplc_at.idx<- function(x) { mutate_at(x, c(1:10), funs(replace(., is.na(.), 0))) }
hybrd.rplc_at.nse<- function(x) { mutate_at(x, vars(var1:var10), funs(replace(., is.na(.), 0))) }
hybrd.rplc_at.stw<- function(x) { mutate_at(x, vars(starts_with("var")), funs(replace(., is.na(.), 0))) }
hybrd.rplc_at.ctn<- function(x) { mutate_at(x, vars(contains("var")), funs(replace(., is.na(.), 0))) }
hybrd.rplc_at.mtc<- function(x) { mutate_at(x, vars(matches("\\d+")), funs(replace(., is.na(.), 0))) }
hybrd.rplc_if <- function(x) { mutate_if(x, is.numeric, funs(replace(., is.na(.), 0))) }
# data.table
library(data.table)
DT.for.set.nms <- function(x) { for (j in names(x))
set(x,which(is.na(x[[j]])),j,0) }
DT.for.set.sqln <- function(x) { for (j in seq_len(ncol(x)))
set(x,which(is.na(x[[j]])),j,0) }
Код для этого анализа:
library(microbenchmark)
# 20% NA filled dataframe of 5 Million rows and 10 columns
set.seed(42) # to recreate the exact dataframe
dfN <- as.data.frame(matrix(sample(c(NA, as.numeric(1:4)), 5e6*10, replace = TRUE),
dimnames = list(NULL, paste0("var", 1:10)),
ncol = 10))
# Running 250 trials with each replacement method
# (the functions are excecuted locally - so that the original dataframe remains unmodified in all cases)
perf_results <- microbenchmark(
hybrid.ifelse = hybrid.ifelse(copy(dfN)),
dplyr_if_else = dplyr_if_else(copy(dfN)),
baseR.sbst.rssgn = baseR.sbst.rssgn(copy(dfN)),
baseR.replace = baseR.replace(copy(dfN)),
dplyr_coalesce = dplyr_coalesce(copy(dfN)),
hybrd.rplc_at.nse= hybrd.rplc_at.nse(copy(dfN)),
hybrd.rplc_at.stw= hybrd.rplc_at.stw(copy(dfN)),
hybrd.rplc_at.ctn= hybrd.rplc_at.ctn(copy(dfN)),
hybrd.rplc_at.mtc= hybrd.rplc_at.mtc(copy(dfN)),
hybrd.rplc_at.idx= hybrd.rplc_at.idx(copy(dfN)),
hybrd.rplc_if = hybrd.rplc_if(copy(dfN)),
tidyr_replace_na = tidyr_replace_na(copy(dfN)),
baseR.for = baseR.for(copy(dfN)),
DT.for.set.nms = DT.for.set.nms(copy(dfN)),
DT.for.set.sqln = DT.for.set.sqln(copy(dfN)),
times = 250L
)
Сводка результатов
> perf_results Unit: milliseconds expr min lq mean median uq max neval hybrid.ifelse 5250.5259 5620.8650 5809.1808 5759.3997 5947.7942 6732.791 250 dplyr_if_else 3209.7406 3518.0314 3653.0317 3620.2955 3746.0293 4390.888 250 baseR.sbst.rssgn 1611.9227 1878.7401 1964.6385 1942.8873 2031.5681 2485.843 250 baseR.replace 1559.1494 1874.7377 1946.2971 1920.8077 2002.4825 2516.525 250 dplyr_coalesce 949.7511 1231.5150 1279.3015 1288.3425 1345.8662 1624.186 250 hybrd.rplc_at.nse 735.9949 871.1693 1016.5910 1064.5761 1104.9590 1361.868 250 hybrd.rplc_at.stw 704.4045 887.4796 1017.9110 1063.8001 1106.7748 1338.557 250 hybrd.rplc_at.ctn 723.9838 878.6088 1017.9983 1063.0406 1110.0857 1296.024 250 hybrd.rplc_at.mtc 686.2045 885.8028 1013.8293 1061.2727 1105.7117 1269.949 250 hybrd.rplc_at.idx 696.3159 880.7800 1003.6186 1038.8271 1083.1932 1309.635 250 hybrd.rplc_if 705.9907 889.7381 1000.0113 1036.3963 1083.3728 1338.190 250 tidyr_replace_na 680.4478 973.1395 978.2678 1003.9797 1051.2624 1294.376 250 baseR.for 670.7897 965.6312 983.5775 1001.5229 1052.5946 1206.023 250 DT.for.set.nms 496.8031 569.7471 695.4339 623.1086 861.1918 1067.640 250 DT.for.set.sqln 500.9945 567.2522 671.4158 623.1454 764.9744 1033.463 250
Boxplot of Results (в логарифмическом масштабе)
# adjust the margins to prepare for better boxplot printing
par(mar=c(8,5,1,1) + 0.1)
# generate boxplot
boxplot(opN, las = 2, xlab = "", ylab = "log(time)[milliseconds]")
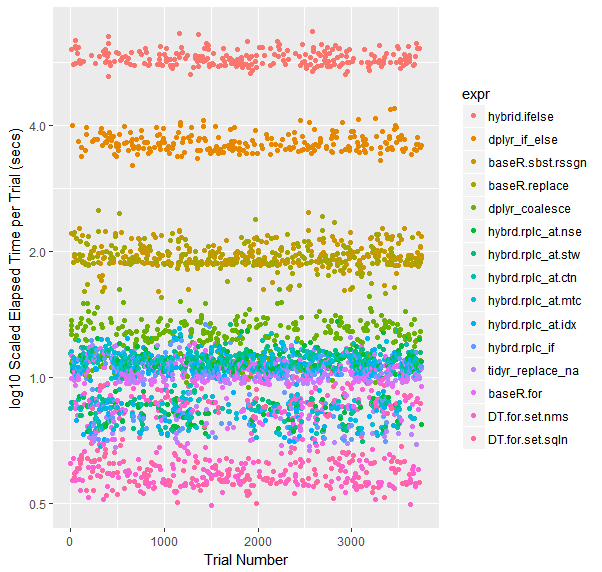
Цветовая диаграмма рассеивания испытаний (в логарифмическом масштабе)
qplot(y=time/10^9, data=opN, colour=expr) +
labs(y = "log10 Scaled Elapsed Time per Trial (secs)", x = "Trial Number") +
scale_y_log10(breaks=c(1, 2, 4))
Примечание о других высоких исполнителей
Когда наборы данных становятся больше, Tidyr's replace_na исторически вытащил впереди. Благодаря текущему набору 50M точек данных, он работает почти так же хорошо, как и Base R For Loop. Мне любопытно посмотреть, что происходит для разных размеров данных.
Дополнительные примеры для mutate а также summarize_at а также _all Варианты функций можно найти здесь: https://rdrr.io/cran/dplyr/man/summarise_all.html Кроме того, я нашел полезные демонстрации и коллекции примеров здесь: https://blog.exploratory.io/dplyr-0-5-is-awesome-heres-why-be095fd4eb8a
Атрибуты и благодарности
С особой благодарностью:
- Тайлер Ринкер и Акрун за демонстрацию микробенчмарка.
- alexis_laz за работу над тем, чтобы помочь мне понять использование
local()и (с помощью пациента Фрэнка тоже) роль, которую тихое принуждение играет в ускорении многих из этих подходов. - ArthurYip для тыка, чтобы добавить новые
coalesce()функционировать и обновлять анализ. - Грегор за толчок, чтобы выяснить
data.tableфункционирует достаточно хорошо, чтобы наконец включить их в состав. - База R для цикла: alexis_laz
- data.table для циклов: Matt_Dowle
(Конечно, пожалуйста, подойдите и отдайте им голоса, если вы найдете такие подходы полезными.)
Примечание по использованию чисел: если у вас есть чистый набор целочисленных данных, все ваши функции будут работать быстрее. Пожалуйста, смотрите работу alexiz_laz для получения дополнительной информации. IRL, я не могу вспомнить, чтобы встретил набор данных, содержащий более 10-15% целых чисел, поэтому я запускаю эти тесты на полностью числовых фреймах данных.
Для одного вектора:
x <- c(1,2,NA,4,5)
x[is.na(x)] <- 0
Для data.frame сделайте функцию из вышеперечисленного, затем apply это к столбцам.
Пожалуйста, предоставьте воспроизводимый пример в следующий раз, как подробно здесь:
Пример dplyr:
library(dplyr)
df1 <- df1 %>%
mutate(myCol1 = if_else(is.na(myCol1), 0, myCol1))
Примечание. Это работает для каждого выбранного столбца. Если нам нужно сделать это для всех столбцов, см. Ответ @reidjax с использованием mutate_each.
Также возможно использовать tidyr::replace_na,
library(tidyr)
df <- df %>% mutate_all(funs(replace_na(.,0)))
Если мы пытаемся заменить NAs при экспорте, например, при записи в csv, тогда мы можем использовать:
write.csv(data, "data.csv", na = "0")
Я знаю, что на этот вопрос уже дан ответ, но для некоторых это может быть полезно:
Определите эту функцию:
na.zero <- function (x) {
x[is.na(x)] <- 0
return(x)
}
Теперь, когда вам нужно преобразовать NA в векторе в ноль, вы можете сделать:
na.zero(some.vector)
Более общий подход к использованию replace() в матрице или векторе для замены NA в 0
Например:
> x <- c(1,2,NA,NA,1,1)
> x1 <- replace(x,is.na(x),0)
> x1
[1] 1 2 0 0 1 1
Это также альтернатива использованию ifelse() в dplyr
df = data.frame(col = c(1,2,NA,NA,1,1))
df <- df %>%
mutate(col = replace(col,is.na(col),0))
С dplyr 0.5.0, вы можете использовать coalesce функция, которая может быть легко интегрирована в %>% трубопровод, делая coalesce(vec, 0), Это заменяет все НС в vec с 0:
Скажем, у нас есть фрейм данных с NAs:
library(dplyr)
df <- data.frame(v = c(1, 2, 3, NA, 5, 6, 8))
df
# v
# 1 1
# 2 2
# 3 3
# 4 NA
# 5 5
# 6 6
# 7 8
df %>% mutate(v = coalesce(v, 0))
# v
# 1 1
# 2 2
# 3 3
# 4 0
# 5 5
# 6 6
# 7 8
Чтобы заменить все NA в кадре данных, вы можете использовать:
df %>% replace(is.na(.), 0)
Я бы прокомментировал сообщение @ianmunoz, но мне не хватает репутации. Вы можете объединить dplyr"s mutate_each а также replace заботиться о NA в 0 замена. Используя фрейм данных из ответа @aL3xa...
> m <- matrix(sample(c(NA, 1:10), 100, replace = TRUE), 10)
> d <- as.data.frame(m)
> d
V1 V2 V3 V4 V5 V6 V7 V8 V9 V10
1 4 8 1 9 6 9 NA 8 9 8
2 8 3 6 8 2 1 NA NA 6 3
3 6 6 3 NA 2 NA NA 5 7 7
4 10 6 1 1 7 9 1 10 3 10
5 10 6 7 10 10 3 2 5 4 6
6 2 4 1 5 7 NA NA 8 4 4
7 7 2 3 1 4 10 NA 8 7 7
8 9 5 8 10 5 3 5 8 3 2
9 9 1 8 7 6 5 NA NA 6 7
10 6 10 8 7 1 1 2 2 5 7
> d %>% mutate_each( funs_( interp( ~replace(., is.na(.),0) ) ) )
V1 V2 V3 V4 V5 V6 V7 V8 V9 V10
1 4 8 1 9 6 9 0 8 9 8
2 8 3 6 8 2 1 0 0 6 3
3 6 6 3 0 2 0 0 5 7 7
4 10 6 1 1 7 9 1 10 3 10
5 10 6 7 10 10 3 2 5 4 6
6 2 4 1 5 7 0 0 8 4 4
7 7 2 3 1 4 10 0 8 7 7
8 9 5 8 10 5 3 5 8 3 2
9 9 1 8 7 6 5 0 0 6 7
10 6 10 8 7 1 1 2 2 5 7
Мы используем стандартную оценку (SE) здесь, поэтому нам нужно подчеркнуть "funs_Мы также используем lazyeval"s interp/~ и . ссылается на "все, с чем мы работаем", то есть на фрейм данных. Теперь есть нули!
Еще один пример использования пакета imputeTS:
library(imputeTS)
na.replace(yourDataframe, 0)
Выделенная функция (nafill / setnafill) для этого подходит к data.table пакет, уже можно проверить установку из ветки
devtools::install_github("Rdatatable/data.table@nafill")
library(data.table)
ans_df = nafill(df, fill=0)
setnafill(df, fill=0) # this one updates in-place
Если вы хотите заменить NA в факторных переменных, это может быть полезно:
n <- length(levels(data.vector))+1
data.vector <- as.numeric(data.vector)
data.vector[is.na(data.vector)] <- n
data.vector <- as.factor(data.vector)
levels(data.vector) <- c("level1","level2",...,"leveln", "NAlevel")
Он преобразует фактор-вектор в числовой вектор и добавляет еще один искусственный уровень числового фактора, который затем преобразуется обратно в фактор-вектор с одним дополнительным "уровнем NA" по вашему выбору.
dplyr>= 1.0.0
В более новых версиях
dplyr:
через () заменяет семейство «вариантов с ограниченным объемом», таких как summarise_at(), summarise_if() и summarise_all().
df <- data.frame(a = c(LETTERS[1:3], NA), b = c(NA, 1:3))
library(tidyverse)
df %>%
mutate(across(where(anyNA), ~ replace_na(., 0)))
a b
1 A 0
2 B 1
3 C 2
4 0 3
Этот код заставит
0быть символом в первом столбце. Заменить
NA в зависимости от типа столбца вы можете использовать формулу типа мурлыканья в
where:
df %>%
mutate(across(where(~ anyNA(.) & is.character(.)), ~ replace_na(., "0")))
Нет необходимости использовать какую-либо библиотеку.
df <- data.frame(a=c(1,3,5,NA))
df$a[is.na(df$a)] <- 0
df
В cleaner пакет имеет na_replace()общий, который по умолчанию заменяет числовые значения нулями, логические значения -FALSE, даты с сегодняшним днем и т.д.:
starwars %>% na_replace()
na_replace(starwars)
Он даже поддерживает векторизованные замены:
mtcars[1:6, c("mpg", "hp")] <- NA
na_replace(mtcars, mpg, hp, replacement = c(999, 123))
Документация: https://msberends.github.io/cleaner/reference/na_replace.html
Ты можешь использовать replace()
Например:
> x <- c(-1,0,1,0,NA,0,1,1)
> x1 <- replace(x,5,1)
> x1
[1] -1 0 1 0 1 0 1 1
> x1 <- replace(x,5,mean(x,na.rm=T))
> x1
[1] -1.00 0.00 1.00 0.00 0.29 0.00 1.00 1.00
Другая dplyr совместимый с трубой вариант с tidyrметод replace_na это работает для нескольких столбцов:
require(dplyr)
require(tidyr)
m <- matrix(sample(c(NA, 1:10), 100, replace = TRUE), 10)
d <- as.data.frame(m)
myList <- setNames(lapply(vector("list", ncol(d)), function(x) x <- 0), names(d))
df <- d %>% replace_na(myList)
Вы можете легко ограничиться, например, числовыми столбцами:
d$str <- c("string", NA)
myList <- myList[sapply(d, is.numeric)]
df <- d %>% replace_na(myList)
Эта простая функция, извлеченная из Datacamp, может помочь:
replace_missings <- function(x, replacement) {
is_miss <- is.na(x)
x[is_miss] <- replacement
message(sum(is_miss), " missings replaced by the value ", replacement)
x
}
затем
replace_missings(df, replacement = 0)
Простой способ написать это с if_na из hablar:
library(dplyr)
library(hablar)
df <- tibble(a = c(1, 2, 3, NA, 5, 6, 8))
df %>%
mutate(a = if_na(a, 0))
который возвращает:
a
<dbl>
1 1
2 2
3 3
4 0
5 5
6 6
7 8
Заменить is.na & NULL во фрейме данных.
- фрейм данных с колонками
$ Name[is.na(A$name)]<-0
ИЛИ
A$name[is.na(A$name)]<-"NA"
- со всем фреймом данных
df [is.na(df)]<-0
- с заменить na на пробел во фрейме данных
df [is.na(df)]<-""
- заменить NULL на NA
df[is.null(df)] <- НЕТ
Если вы хотите присвоить новое имя после изменения NA в определенном столбце, в этом случае столбец V3, используйте вы также можете сделать это
my.data.frame$the.new.column.name <- ifelse(is.na(my.data.frame$V3),0,1)
Другой вариант — использоватьcollapse::replace_NA. По умолчанию,replace_NAзаменяет NA на 0.
library(collapse)
replace_NA(df)
Только для некоторых столбцов:
replace_NA(df, cols = c("V1", "V5"))
#Alternatively, one can use a function, indices or a logical vector to select the columns
Это также быстрее, чем любой другой ответ (см. этот ответ для сравнения):
set.seed(42) # to recreate the exact dataframe
dfN <- as.data.frame(matrix(sample(c(NA, as.numeric(1:4)), 1e7*10, replace = TRUE),
dimnames = list(NULL, paste0("var", 1:10)),
ncol = 10))
microbenchmark(collapse = replace_NA(dfN))
# Unit: milliseconds
# expr min lq mean median uq max neval
# collapse 508.9198 621.405 751.3413 714.835 859.5437 1298.69 100
Я хочу добавить следующее решение, использующее популярный Hmiscпакет .
library(Hmisc)
data(airquality)
# imputing with 0 - all columns
# although my favorite one for simple imputations is Hmisc::impute(x, "random")
> dd <- data.frame(Map(function(x) Hmisc::impute(x, 0), airquality))
> str(dd[[1]])
'impute' Named num [1:153] 41 36 12 18 0 28 23 19 8 0 ...
- attr(*, "names")= chr [1:153] "1" "2" "3" "4" ...
- attr(*, "imputed")= int [1:37] 5 10 25 26 27 32 33 34 35 36 ...
> dd[[1]][1:10]
1 2 3 4 5 6 7 8 9 10
41 36 12 18 0* 28 23 19 8 0*
Видно, что все метаданные вменения распределяются как атрибуты. Таким образом, его можно было использовать позже.
Другой вариант использованияsapplyзаменить всеNAс нулями. Вот воспроизводимый код (данные @aL3xa):
set.seed(7) # for reproducibility
m <- matrix(sample(c(NA, 1:10), 100, replace = TRUE), 10)
d <- as.data.frame(m)
d
#> V1 V2 V3 V4 V5 V6 V7 V8 V9 V10
#> 1 9 7 5 5 7 7 4 6 6 7
#> 2 2 5 10 7 8 9 8 8 1 8
#> 3 6 7 4 10 4 9 6 8 NA 10
#> 4 1 10 3 7 5 7 7 7 NA 8
#> 5 9 9 10 NA 7 10 1 5 NA 5
#> 6 5 2 5 10 8 1 1 5 10 3
#> 7 7 3 9 3 1 6 7 3 1 10
#> 8 7 7 6 8 4 4 5 NA 8 7
#> 9 2 1 1 2 7 5 9 10 9 3
#> 10 7 5 3 4 9 2 7 6 NA 5
d[sapply(d, \(x) is.na(x))] <- 0
d
#> V1 V2 V3 V4 V5 V6 V7 V8 V9 V10
#> 1 9 7 5 5 7 7 4 6 6 7
#> 2 2 5 10 7 8 9 8 8 1 8
#> 3 6 7 4 10 4 9 6 8 0 10
#> 4 1 10 3 7 5 7 7 7 0 8
#> 5 9 9 10 0 7 10 1 5 0 5
#> 6 5 2 5 10 8 1 1 5 10 3
#> 7 7 3 9 3 1 6 7 3 1 10
#> 8 7 7 6 8 4 4 5 0 8 7
#> 9 2 1 1 2 7 5 9 10 9 3
#> 10 7 5 3 4 9 2 7 6 0 5
Создано 15 января 2023 г. с использованием репрекса версии 2.0.2.
Обратите внимание: начиная с версии R 4.1.0 вы можете использовать\(x)вместоfunction(x).
Это не совсем новое решение, но мне нравится писать встроенные лямбды, которые обрабатывают вещи, которые я не могу заставить делать пакеты. В таком случае,
df %>%
(function(x) { x[is.na(x)] <- 0; return(x) })
Поскольку R никогда не «проходит мимо объекта», как вы могли бы видеть в Python, это решение не изменяет исходную переменную.
Обратите внимание на скобки вокруг определения функции! Хотя мне это кажется немного избыточным, поскольку определение функции заключено в фигурные скобки, требуется, чтобы встроенные функции были определены в скобках для
Это более гибкое решение. Это работает независимо от того, насколько велик ваш фрейм данных, или ноль обозначен0илиzeroили что угодно.
library(dplyr) # make sure dplyr ver is >= 1.00
df %>%
mutate(across(everything(), na_if, 0)) # if 0 is indicated by `zero` then replace `0` with `zero`
в data.frame нет необходимости создавать новый столбец путем изменения.
library(tidyverse)
k <- c(1,2,80,NA,NA,51)
j <- c(NA,NA,3,31,12,NA)
df <- data.frame(k,j)%>%
replace_na(list(j=0))#convert only column j, for example
результат
k j
1 0
2 0
80 3
NA 31
NA 12
51 0
Я использовал это лично и отлично работает:
players_wd$APPROVED_WD[is.na(players_wd$APPROVED_WD)] <- 0