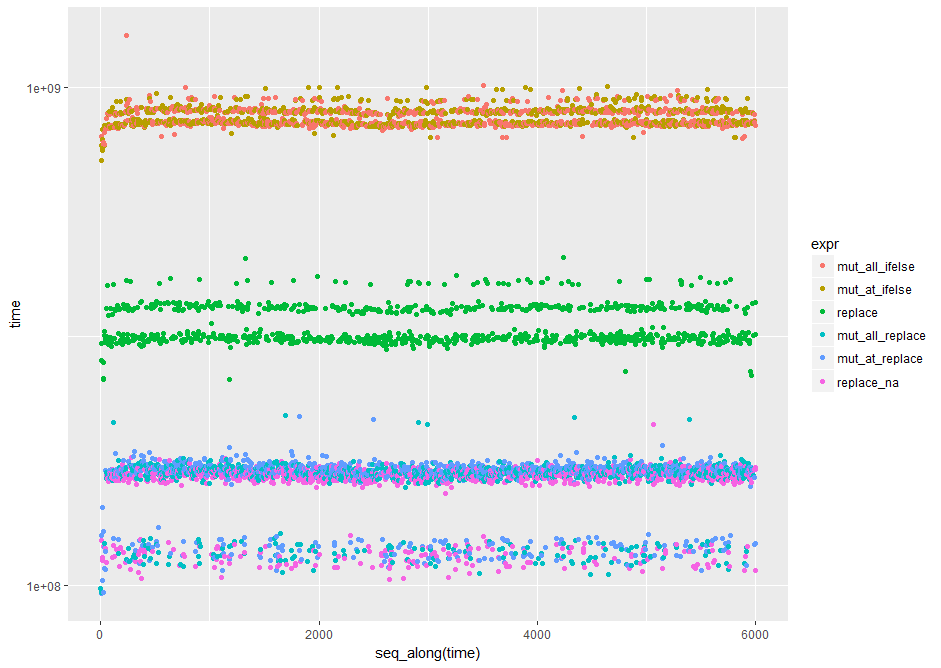
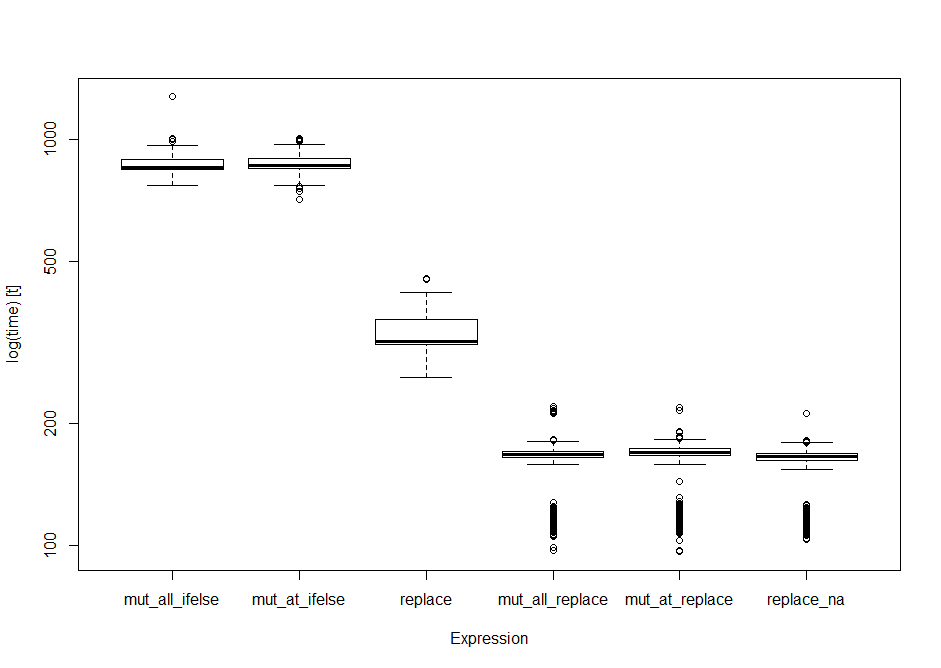Расхождения в контрольных показателях и результатах обработки
Я пытался провести небольшое тестирование наиболее эффективных способов замены NA в кадрах данных.
Я начал со сравнения NA с 0 заменяющими решениями для набора данных с 1 миллионом строк и 12 столбцов. Бросив все способные трубы в microbenchmark Я получил следующие результаты.
Вопрос 1: Есть ли способ проверить подмножество левых операторов присваивания (например:df1[is.na(df1)] <- 0) внутри benchmark функционировать?
library(dplyr)
library(tidyr)
library(microbenchmark)
set.seed(24)
df1 <- as.data.frame(matrix(sample(c(NA, 1:5), 1e6 *12, replace=TRUE),
dimnames = list(NULL, paste0("var", 1:12)), ncol=12))
op <- microbenchmark(
mut_all_ifelse = df1 %>% mutate_all(funs(ifelse(is.na(.), 0, .))),
mut_at_ifelse = df1 %>% mutate_at(funs(ifelse(is.na(.), 0, .)), .cols = c(1:12)),
# df1[is.na(df1)] <- 0 would sit here, but I can't make it work inside this function
replace = df1 %>% replace(., is.na(.), 0),
mut_all_replace = df1 %>% mutate_all(funs(replace(., is.na(.), 0))),
mut_at_replace = df1 %>% mutate_at(funs(replace(., is.na(.), 0)), .cols = c(1:12)),
replace_na = df1 %>% replace_na(list(var1 = 0, var2 = 0, var3 = 0, var4 = 0, var5 = 0, var6 = 0, var7 = 0, var8 = 0, var9 = 0, var10 = 0, var11 = 0, var12 = 0)),
times = 1000L
)
print(op) #standard data frame of the output
Unit: milliseconds
expr min lq mean median uq max neval
mut_all_ifelse 769.87848 844.5565 871.2476 856.0941 895.4545 1274.5610 1000
mut_at_ifelse 713.48399 847.0322 875.9433 861.3224 899.7102 1006.6767 1000
replace 258.85697 311.9708 334.2291 317.3889 360.6112 455.7596 1000
mut_all_replace 96.81479 164.1745 160.6151 167.5426 170.5497 219.5013 1000
mut_at_replace 96.23975 166.0804 161.9302 169.3984 172.7442 219.0359 1000
replace_na 103.04600 161.2746 156.7804 165.1649 168.3683 210.9531 1000
boxplot(op) #boxplot of output
library(ggplot2) #nice log plot of the output
qplot(y=time, data=op, colour=expr) + scale_y_log10()
Для проверки оператора присваивания я сначала запустил эти тесты.
set.seed(24)
> Book1 <- as.data.frame(matrix(sample(c(NA, 1:5), 1e8 *12, replace=TRUE),
+ dimnames = list(NULL, paste0("var", 1:12)), ncol=12))
> system.time({
+ Book1 %>% mutate_all(funs(ifelse(is.na(.), 0, .))) })
user system elapsed
52.79 24.66 77.45
>
> system.time({
+ Book1 %>% mutate_at(funs(ifelse(is.na(.), 0, .)), .cols = c(1:12)) })
user system elapsed
52.74 25.16 77.91
>
> system.time({
+ Book1[is.na(Book1)] <- 0 })
user system elapsed
16.65 7.86 24.51
>
> system.time({
+ Book1 %>% replace_na(list(var1 = 0, var2 = 0, var3 = 0, var4 = 0, var5 = 0, var6 = 0, var7 = 0, var8 = 0, var9 = 0,var10 = 0, var11 = 0, var12 = 0)) })
user system elapsed
3.54 2.13 5.68
>
> system.time({
+ Book1 %>% mutate_at(funs(replace(., is.na(.), 0)), .cols = c(1:12)) })
user system elapsed
3.37 2.26 5.63
>
> system.time({
+ Book1 %>% mutate_all(funs(replace(., is.na(.), 0))) })
user system elapsed
3.33 2.26 5.58
>
> system.time({
+ Book1 %>% replace(., is.na(.), 0) })
user system elapsed
3.42 1.09 4.51
В этих тестах база replace() приходит первым В бенчмаркинговых испытаниях replace падает дальше в строю, пока тидир replace_na() выигрывает (по носу) Выполнение единичных тестов многократно и для разных форм и размеров фреймов данных всегда находит основу replace() во главе.
Вопрос 2: Как его производительность может быть единственным результатом, который не соответствует результатам простого теста?
Еще более озадачивающе - Вопрос 3: Как все mutate_all/_at(replace()) работать быстрее простого replace()? Многие люди сообщают об этом: http://datascience.la/dplyr-and-a-very-basic-benchmark/ (и все ссылки в этой статье), но я до сих пор не нашел объяснения, почему помимо этого хеширования и C++ используются.)
с особой благодарностью уже Тайлеру Ринкеру: https://www.r-bloggers.com/microbenchmarking-with-r/ и akrun: /questions/9148514/dplyrmutate-naznachit-narm-true/9148524#9148524
1 ответ
Вы можете включить сложное / многократное утверждение в microbenchmark оборачивая его {} который, в основном, преобразуется в одно выражение:
microbenchmark(expr1 = { df1[is.na(df1)] = 0 },
exp2 = { tmp = 1:10; tmp[3] = 0L; tmp2 = tmp + 12L; tmp2 ^ 2 },
times = 10)
#Unit: microseconds
# expr min lq mean median uq max neval cld
# expr1 124953.716 137244.114 158576.030 142405.685 156744.076 284779.353 10 b
# exp2 2.784 3.132 17.748 23.142 24.012 38.976 10 a
Стоит отметить побочные эффекты этого:
tmp
#[1] 1 2 0 4 5 6 7 8 9 10
в отличие, скажем, что-то вроде:
rm(tmp)
microbenchmark(expr1 = { df1[is.na(df1)] = 0 },
exp2 = local({ tmp = 1:10; tmp[3] = 0L; tmp2 = tmp + 12L; tmp2 ^ 2 }),
times = 10)
#Unit: microseconds
# expr min lq mean median uq max neval cld
# expr1 127250.18 132935.149 165296.3030 154509.553 169917.705 314820.306 10 b
# exp2 10.44 12.181 42.5956 54.636 57.072 97.789 10 a
tmp
#Error: object 'tmp' not found
Заметив побочный эффект теста, мы видим, что первая операция, которая удаляет NA значения оставляют довольно легкую работу для следующих альтернатив:
# re-assign because we changed it before
set.seed(24)
df1 = as.data.frame(matrix(sample(c(NA, 1:5), 1e6 * 12, TRUE),
dimnames = list(NULL, paste0("var", 1:12)), ncol = 12))
unique(sapply(df1, typeof))
#[1] "integer"
any(sapply(df1, anyNA))
#[1] TRUE
system.time({ df1[is.na(df1)] <- 0 })
# user system elapsed
# 0.39 0.14 0.53
Предыдущий тест оставляет нам:
unique(sapply(df1, typeof))
#[1] "double"
any(sapply(df1, anyNA))
#[1] FALSE
И замена NAкогда их нет, следует / следует учитывать, чтобы ничего не делать на входе.
Кроме того, обратите внимание, что во всех ваших альтернативах вы назначаете "двойной" (typeof(0)) к "целочисленным" столбцам-векторам (sapply(df1, typeof)). В то время как, я не думаю, что есть какой-либо случай (в вышеупомянутых альтернативах), где df1 модифицируется на месте (так как после создания "data.frame" сохраняется информация для копирования его векторных столбцов в случае изменения), -still- это небольшие, но неизбежные издержки при приведении к "double" и хранение как "двойник". R перед заменой элементов в "целочисленном" векторе выделит и скопирует (в случае "целочисленной" замены) или выделит и принудительно (в случае "двойной" замены). Кроме того, после первого принуждения (из-за побочного эффекта эталонного теста, как отмечено выше), R будет работать на "двойных" и это содержит более медленные манипуляции, чем на "целых". Я не могу найти простой способ R исследовать эту разницу, но в двух словах (рискуя быть не совсем точным), мы можем смоделировать эти операции следующим образом:
# simulate R's copying of int to int
# allocate a new int and copy
int2int = inline::cfunction(sig = c(x = "integer"), body = '
SEXP ans = PROTECT(allocVector(INTSXP, LENGTH(x)));
memcpy(INTEGER(ans), INTEGER(x), LENGTH(x) * sizeof(int));
UNPROTECT(1);
return(ans);
')
# R's coercing of int to double
# 'coerceVector', internally, allocates a double and coerces to populate it
int2dbl = inline::cfunction(sig = c(x = "integer"), body = '
SEXP ans = PROTECT(coerceVector(x, REALSXP));
UNPROTECT(1);
return(ans);
')
# simulate R's copying form double to double
dbl2dbl = inline::cfunction(sig = c(x = "double"), body = '
SEXP ans = PROTECT(allocVector(REALSXP, LENGTH(x)));
memcpy(REAL(ans), REAL(x), LENGTH(x) * sizeof(double));
UNPROTECT(1);
return(ans);
')
И на бенчмарке:
x.int = 1:1e7; x.dbl = as.numeric(x.int)
microbenchmark(int2int(x.int), int2dbl(x.int), dbl2dbl(x.dbl), times = 50)
#Unit: milliseconds
# expr min lq mean median uq max neval cld
# int2int(x.int) 16.42710 16.91048 21.93023 17.42709 19.38547 54.36562 50 a
# int2dbl(x.int) 35.94064 36.61367 47.15685 37.40329 63.61169 78.70038 50 b
# dbl2dbl(x.dbl) 33.51193 34.18427 45.30098 35.33685 63.45788 75.46987 50 b
Завершить (!) Всю предыдущую заметку, заменив 0 с 0L сэкономим время...
Наконец, чтобы более точно воспроизвести эталонный тест, мы могли бы использовать:
library(dplyr)
library(tidyr)
library(microbenchmark)
set.seed(24)
df1 = as.data.frame(matrix(sample(c(NA, 1:5), 1e6 * 12, TRUE),
dimnames = list(NULL, paste0("var", 1:12)), ncol = 12))
Завернуть в функции:
stopifnot(ncol(df1) == 12) #some of the alternatives are hardcoded to 12 columns
mut_all_ifelse = function(x, val) x %>% mutate_all(funs(ifelse(is.na(.), val, .)))
mut_at_ifelse = function(x, val) x %>% mutate_at(funs(ifelse(is.na(.), val, .)), .cols = c(1:12))
baseAssign = function(x, val) { x[is.na(x)] <- val; x }
baseFor = function(x, val) { for(j in 1:ncol(x)) x[[j]][is.na(x[[j]])] = val; x }
base_replace = function(x, val) x %>% replace(., is.na(.), val)
mut_all_replace = function(x, val) x %>% mutate_all(funs(replace(., is.na(.), val)))
mut_at_replace = function(x, val) x %>% mutate_at(funs(replace(., is.na(.), val)), .cols = c(1:12))
myreplace_na = function(x, val) x %>% replace_na(list(var1 = val, var2 = val, var3 = val, var4 = val, var5 = val, var6 = val, var7 = val, var8 = val, var9 = val, var10 = val, var11 = val, var12 = val))
Тест на равенство результатов перед контрольными показателями:
identical(mut_all_ifelse(df1, 0), mut_at_ifelse(df1, 0))
#[1] TRUE
identical(mut_at_ifelse(df1, 0), baseAssign(df1, 0))
#[1] TRUE
identical(baseAssign(df1, 0), baseFor(df1, 0))
#[1] TRUE
identical(baseFor(df1, 0), base_replace(df1, 0))
#[1] TRUE
identical(base_replace(df1, 0), mut_all_replace(df1, 0))
#[1] TRUE
identical(mut_all_replace(df1, 0), mut_at_replace(df1, 0))
#[1] TRUE
identical(mut_at_replace(df1, 0), myreplace_na(df1, 0))
#[1] TRUE
Тест с принуждением к "двойному":
benchnum = microbenchmark(mut_all_ifelse(df1, 0),
mut_at_ifelse(df1, 0),
baseAssign(df1, 0),
baseFor(df1, 0),
base_replace(df1, 0),
mut_all_replace(df1, 0),
mut_at_replace(df1, 0),
myreplace_na(df1, 0),
times = 10)
benchnum
#Unit: milliseconds
# expr min lq mean median uq max neval cld
# mut_all_ifelse(df1, 0) 1368.5091 1441.9939 1497.5236 1509.2233 1550.1416 1629.6959 10 c
# mut_at_ifelse(df1, 0) 1366.1674 1389.2256 1458.1723 1464.5962 1503.4337 1553.7110 10 c
# baseAssign(df1, 0) 532.4975 548.9444 586.8198 564.3940 655.8083 667.8634 10 b
# baseFor(df1, 0) 169.6048 175.9395 206.7038 189.5428 197.6472 308.6965 10 a
# base_replace(df1, 0) 518.7733 547.8381 597.8842 601.1544 643.4970 666.6872 10 b
# mut_all_replace(df1, 0) 169.1970 183.5514 227.1978 194.0903 291.6625 346.4649 10 a
# mut_at_replace(df1, 0) 176.7904 186.4471 227.3599 202.9000 303.4643 309.2279 10 a
# myreplace_na(df1, 0) 172.4926 177.8518 199.1469 186.3645 192.1728 297.0419 10 a
Тест без принуждения к "двойному":
benchint = microbenchmark(mut_all_ifelse(df1, 0L),
mut_at_ifelse(df1, 0L),
baseAssign(df1, 0L),
baseFor(df1, 0L),
base_replace(df1, 0L),
mut_all_replace(df1, 0L),
mut_at_replace(df1, 0L),
myreplace_na(df1, 0L),
times = 10)
benchint
#Unit: milliseconds
# expr min lq mean median uq max neval cld
# mut_all_ifelse(df1, 0L) 1291.17494 1313.1910 1377.9265 1353.2812 1417.4389 1554.6110 10 c
# mut_at_ifelse(df1, 0L) 1295.34053 1315.0308 1372.0728 1353.0445 1431.3687 1478.8613 10 c
# baseAssign(df1, 0L) 451.13038 461.9731 477.3161 471.0833 484.9318 528.4976 10 b
# baseFor(df1, 0L) 98.15092 102.4996 115.7392 107.9778 136.2227 139.7473 10 a
# base_replace(df1, 0L) 428.54747 451.3924 471.5011 470.0568 497.7088 516.1852 10 b
# mut_all_replace(df1, 0L) 101.66505 102.2316 137.8128 130.5731 161.2096 243.7495 10 a
# mut_at_replace(df1, 0L) 103.79796 107.2533 119.1180 112.1164 127.7959 166.9113 10 a
# myreplace_na(df1, 0L) 100.03431 101.6999 120.4402 121.5248 137.1710 141.3913 10 a
И простой способ визуализации:
boxplot(benchnum, ylim = range(min(summary(benchint)$min, summary(benchnum)$min),
max(summary(benchint)$max, summary(benchnum)$max)))
boxplot(benchint, add = TRUE, border = "red", axes = FALSE)
legend("topright", c("coerce", "not coerce"), fill = c("black", "red"))
Обратите внимание, что df1 после всего этого остается неизменным (str(df1)).