r - phyloseq - plot_tree - упорядочивает выборки по переменной, а не по SampleType
У меня есть объект phyloseq, и я хочу посмотреть, как изменяется численность видов по отношению к некоторой переменной "rand"
С http://joey711.github.io/phyloseq-demo/phyloseq-demo.html
library(phyloseq)
library(tidyverse)
data(GlobalPatterns)
GP.chl <- subset_taxa(GlobalPatterns, Phylum == "Chlamydiae") %>% transform_sample_counts(function(OTU) OTU + 1) %>%
transform_sample_counts(function(OTU) (OTU/sum(OTU)))
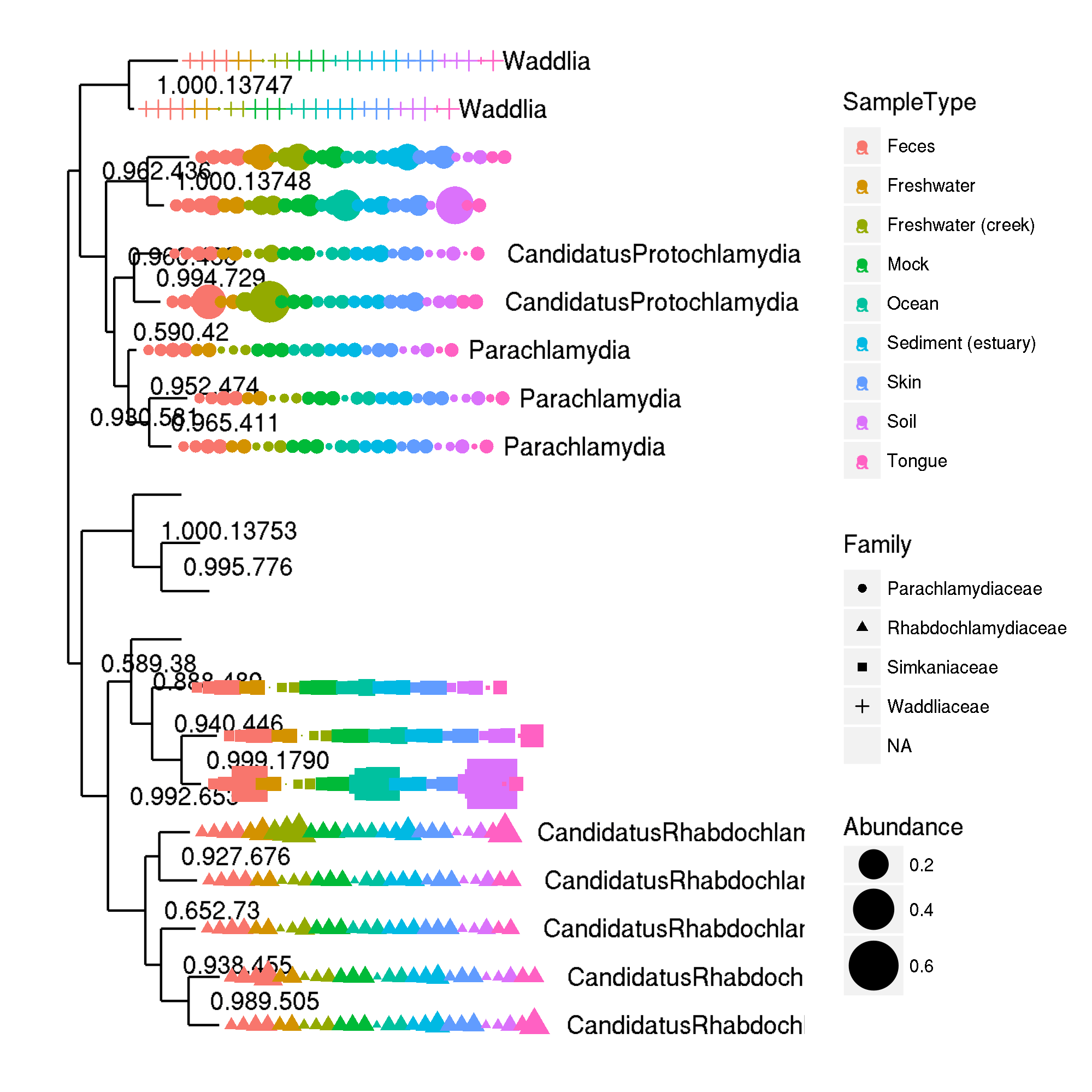
pt <- plot_tree(GP.chl2, color = "SampleType", shape = "Family", label.tips = "Genus",
size = "abundance", plot.margin = 0.5, ladderize = TRUE, base.spacing = 0.08) + scale_size(range = c(0,10))
pt
Я хочу показать, как переменные (представленные в виде цветных символов) меняются по отношению к переменной rand.
Обычно можно переупорядочить переменные в большинстве графиков phyloseq, как описано в первом комментарии Джои здесь. https://github.com/joey711/phyloseq/issues/518
В этом случае, однако, pt $ data представляет собой таблицу с одной записью для каждого таксона, а не для каждого образца, поэтому повторное упорядочение, похоже, не помогает. Также не производится переупорядочение столбцов в таблице OTU. Я хотел бы иметь возможность раскрасить код по типу образца, поэтому изменение переменной цвета не кажется достаточным исправлением. Какие-либо предложения?