Схема аккордов с несколькими уровнями данных
Я нахожусь немного застрявшим, я хочу показать потоки между регионами для выловленных видов через диаграмму аккордов на циклическом цикле, но не могу понять, как построить график, когда столбцы 1 и 2 представляют "связь", столбец 3 - " фактор "интереса" и столбец 4 являются значениями. Я включил образец данных ниже (да, я знаю, что Индонезия - это регион), так как вы можете видеть, что каждый вид не уникален для конкретного региона. Я хотел бы подготовить сюжет, аналогичный приведенному ниже, но заменить "страны" на "виды" для каждого региона. Возможно ли это сделать?
import_region export_region species flow
North America Europe Acanthosaura armata 0.0104
Southeast Asia Europe Acanthosaura armata 0.0022
Indonesia Europe Acanthosaura armata 0.1971
Indonesia Europe Acrochordus granulatus 0.7846
Southeast Asia Europe Acrochordus granulatus 0.1101
Indonesia Europe Acrochordus javanicus 2.00E-04
Southeast Asia Europe Acrochordus javanicus 0.0015
Indonesia North America Acrochordus javanicus 0.0024
East Asia Europe Acrochordus javanicus 0.0028
Indonesia Europe Ahaetulla prasina 4.00E-04
Southeast Asia Europe Ahaetulla prasina 4.00E-04
Southeast Asia East Asia Amyda cartilaginea 0.0027
Indonesia East Asia Amyda cartilaginea 5.00E-04
Indonesia Europe Amyda cartilaginea 0.004
Indonesia Southeast Asia Amyda cartilaginea 0.0334
Europe North America Amyda cartilaginea 4.00E-04
Indonesia North America Amyda cartilaginea 0.1291
Southeast Asia Southeast Asia Amyda cartilaginea 0.0283
Indonesia West Asia Amyda cartilaginea 0.7614
South Asia Europe Amyda cartilaginea 2.8484
Australasia Europe Apodora papuana 0.0368
Indonesia North America Apodora papuana 0.324
Indonesia Europe Apodora papuana 0.0691
Europe Europe Apodora papuana 0.0106
Indonesia East Asia Apodora papuana 0.0129
Europe North America Apodora papuana 0.0034
East Asia East Asia Apodora papuana 2.00E-04
Indonesia Southeast Asia Apodora papuana 0.0045
East Asia North America Apodora papuans 0.0042
пример диаграммы, аналогичной той, что я хотел бы, пожалуйста, нажмите на ссылку ниже: диаграмма аккордов
1 ответ
В круглом пакете ChordDiagram() Функция допускает только столбец "from", столбец "to" и необязательный столбец "value". Однако, в вашем случае, на самом деле мы можем сделать некоторое преобразование для исходного фрейма данных, чтобы преобразовать его в фрейм данных с тремя столбцами.
В вашем примере вы хотите отличить, например, Acanthosaura_armata в Северной Америке от Acanthosaura_armata в Европе, одно решение - объединить названия регионов и названия видов, такие как Acanthosaura_armata|North_America сформировать уникальный идентификатор. Далее я покажу, как визуализировать этот набор данных с помощью пакета circlize.
Читайте в данных. Обратите внимание, я заменил пробел с подчеркиванием.
df = read.table(textConnection(
"import_region export_region species flow
North_America Europe Acanthosaura_armata 0.0104
Southeast_Asia Europe Acanthosaura_armata 0.0022
Indonesia Europe Acanthosaura_armata 0.1971
Indonesia Europe Acrochordus_granulatus 0.7846
Southeast_Asia Europe Acrochordus_granulatus 0.1101
Indonesia Europe Acrochordus_javanicus 2.00E-04
Southeast_Asia Europe Acrochordus_javanicus 0.0015
Indonesia North_America Acrochordus_javanicus 0.0024
East_Asia Europe Acrochordus_javanicus 0.0028
Indonesia Europe Ahaetulla_prasina 4.00E-04
Southeast_Asia Europe Ahaetulla_prasina 4.00E-04
Southeast_Asia East_Asia Amyda_cartilaginea 0.0027
Indonesia East_Asia Amyda_cartilaginea 5.00E-04
Indonesia Europe Amyda_cartilaginea 0.004
Indonesia Southeast_Asia Amyda_cartilaginea 0.0334
Europe North_America Amyda_cartilaginea 4.00E-04
Indonesia North_America Amyda_cartilaginea 0.1291
Southeast_Asia Southeast_Asia Amyda_cartilaginea 0.0283
Indonesia West_Asia Amyda_cartilaginea 0.7614
South_Asia Europe Amyda_cartilaginea 2.8484
Australasia Europe Apodora_papuana 0.0368
Indonesia North_America Apodora_papuana 0.324
Indonesia Europe Apodora_papuana 0.0691
Europe Europe Apodora_papuana 0.0106
Indonesia East_Asia Apodora_papuana 0.0129
Europe North_America Apodora_papuana 0.0034
East_Asia East_Asia Apodora_papuana 2.00E-04
Indonesia Southeast_Asia Apodora_papuana 0.0045
East_Asia North_America Apodora_papuans 0.0042"),
header = TRUE, stringsAsFactors = FALSE)
Кроме того, я удалил несколько строк, которые имеют очень маленькие значения.
df = df[df[[4]] > 0.01, ]
Назначьте цвета для видов и регионов.
library(circlize)
library(RColorBrewer)
all_species = unique(df[[3]])
color_species = structure(brewer.pal(length(all_species), "Set1"), names = all_species)
all_regions = unique(c(df[[1]], df[[2]]))
color_regions = structure(brewer.pal(length(all_regions), "Set2"), names = all_regions)
Группировка по видам
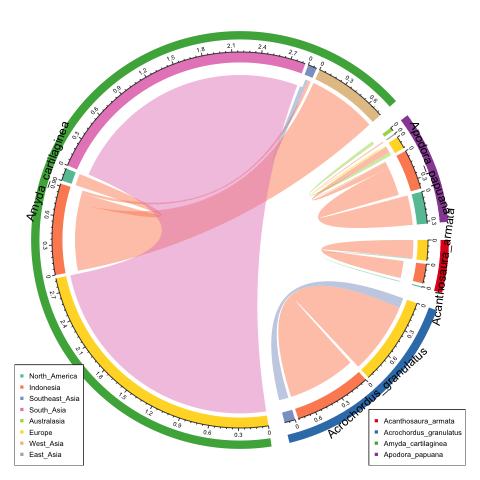
Сначала я покажу, как сгруппировать диаграмму аккордов по видам.
Как упоминалось ранее, мы используем species|region как уникальный идентификатор.
df2 = data.frame(from = paste(df[[3]], df[[1]], sep = "|"),
to = paste(df[[3]], df[[2]], sep = "|"),
value = df[[4]], stringsAsFactors = FALSE)
Затем мы корректируем порядок всех секторов, чтобы сначала упорядочить по видам, а затем по регионам.
combined = unique(data.frame(regions = c(df[[1]], df[[2]]),
species = c(df[[3]], df[[3]]), stringsAsFactors = FALSE))
combined = combined[order(combined$species, combined$regions), ]
order = paste(combined$species, combined$regions, sep = "|")
Мы хотим, чтобы цвет ссылок был таким же, как цвет regoins
grid.col = structure(color_regions[combined$regions], names = order)
Поскольку диаграмма аккордов сгруппирована по видам, промежутки между видами должны быть больше, чем внутри каждого вида.
gap = rep(1, length(order))
gap[which(!duplicated(combined$species, fromLast = TRUE))] = 5
Теперь, когда все настройки готовы, мы можем создать диаграмму аккордов:
В следующем коде мы устанавливаем preAllocateTracks так что круговые линии, которые представляют виды, будут добавлены позже.
circos.par(gap.degree = gap)
chordDiagram(df2, order = order, annotationTrack = c("grid", "axis"),
grid.col = grid.col, directional = TRUE,
preAllocateTracks = list(
track.height = 0.04,
track.margin = c(0.05, 0)
)
)
Круговые линии добавлены для представления видов:
for(species in unique(combined$species)) {
l = combined$species == species
sn = paste(combined$species[l], combined$regions[l], sep = "|")
highlight.sector(sn, track.index = 1, col = color_species[species],
text = species, niceFacing = TRUE)
}
circos.clear()
И легенды для регионов и видов:
legend("bottomleft", pch = 15, col = color_regions,
legend = names(color_regions), cex = 0.6)
legend("bottomright", pch = 15, col = color_species,
legend = names(color_species), cex = 0.6)
Сюжет выглядит так:
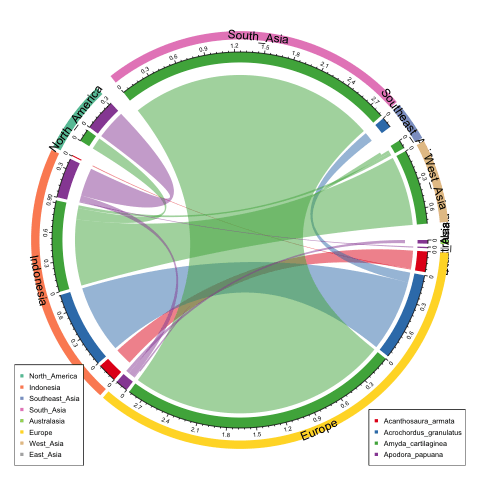
Группировка по регионам
Код похож на то, что я не буду его объяснять, а просто приложу код в посте. Сюжет выглядит так:
## group by regions
df2 = data.frame(from = paste(df[[1]], df[[3]], sep = "|"),
to = paste(df[[2]], df[[3]], sep = "|"),
value = df[[4]], stringsAsFactors = FALSE)
combined = unique(data.frame(regions = c(df[[1]], df[[2]]),
species = c(df[[3]], df[[3]]), stringsAsFactors = FALSE))
combined = combined[order(combined$regions, combined$species), ]
order = paste(combined$regions, combined$species, sep = "|")
grid.col = structure(color_species[combined$species], names = order)
gap = rep(1, length(order))
gap[which(!duplicated(combined$species, fromLast = TRUE))] = 5
circos.par(gap.degree = gap)
chordDiagram(df2, order = order, annotationTrack = c("grid", "axis"),
grid.col = grid.col, directional = TRUE,
preAllocateTracks = list(
track.height = 0.04,
track.margin = c(0.05, 0)
)
)
for(region in unique(combined$regions)) {
l = combined$regions == region
sn = paste(combined$regions[l], combined$species[l], sep = "|")
highlight.sector(sn, track.index = 1, col = color_regions[region],
text = region, niceFacing = TRUE)
}
circos.clear()
legend("bottomleft", pch = 15, col = color_regions,
legend = names(color_regions), cex = 0.6)
legend("bottomright", pch = 15, col = color_species, l
egend = names(color_species), cex = 0.6)