R: Добавление групповых и отдельных полиномиальных линий тренда к графику GMM
Я борюсь с тем, как добавить как индивидуальную, так и групповую линию тренда к своим графикам. (R и использование ggplot2).
Вот код, который я использую:
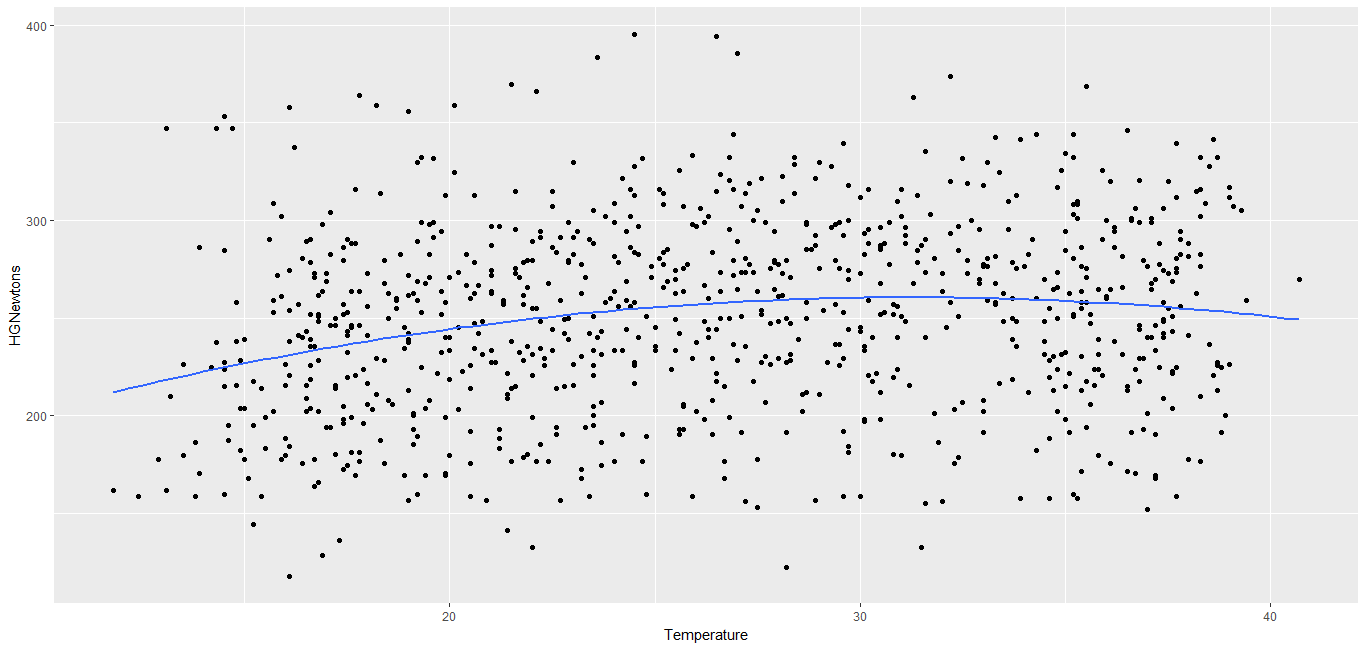
MensHG.fm2=lmer(HGNewtons~Temperature+QuadTemp+Run+(1|Subject),MenstrualData) #model
plot.hg<-data.frame(MensHG.fm2@frame,fitted.re=fitted(MensHG.fm2))
g1<-ggplot(plot.hg,aes(x=Temperature,y=HGNewtons))+geom_point()
g2<-g1+facet_wrap(~Subject, nrow=6)+ylab(bquote('HG MVF (N)'))+xlab(bquote('Hand ' ~T[sk] ~(degree*C)))
g3<-g2+geom_smooth(method="glm", formula=y~ploy(x,2), se=FALSE) #This gives me my individual trendlines
Теперь я хочу поставить линию тренда для части данных g1 (то есть общего тренда) на каждом из моих отдельных графиков - каков наилучший способ сделать это? Я могу видеть тенденцию, если я использую код:
g5=g1+geom_smooth(method="glm", formula=y~poly(x,2), se=FALSE)
НО эта линия тренда исчезает, как только я делаю фасетку (я получаю тот же результат, что и g3)
Похоже, это не решает проблему с помощью: g4<-g3 + geom_smooth (data = MensHG.fm2)
1 ответ
Без минимального рабочего примера ваших данных я использовал встроенные данные радужной оболочки. Здесь я сделал вид, что Виды были разными предметами ради демонстрации.
library(lme4)
library(ggplot2)
fit.iris <- lmer(Sepal.Width ~ Sepal.Length + I(Sepal.Length^2) + (1|Species), data = iris)
Я также использую два дополнительных пакета для простоты, broom а также dplyr, augment от broom делает то же самое, что вы сделали с ..., fitted.re=fitted(MensHG.fm2), но с некоторыми дополнительными прибамбасами. Я также использую dplyr::select, но это не обязательно, в зависимости от желаемого результата (разница между рисунком 2 и рисунком 3).
library(broom)
library(dplyr)
augment(fit.iris)
# output here truncated for simplicity
Sepal.Width Sepal.Length I.Sepal.Length.2. Species .fitted .resid .fixed ... 1 3.5 5.1 26.01 setosa 3.501175 -0.001175181 2.756738 2 3.0 4.9 24.01 setosa 3.371194 -0.371193601 2.626757 3 3.2 4.7 22.09 setosa 3.230650 -0.030649983 2.486213 4 3.1 4.6 21.16 setosa 3.156417 -0.056417409 2.411981 5 3.6 5.0 25 setosa 3.437505 0.162495354 2.693068 6 3.9 5.4 29.16 setosa 3.676344 0.223656271 2.931907
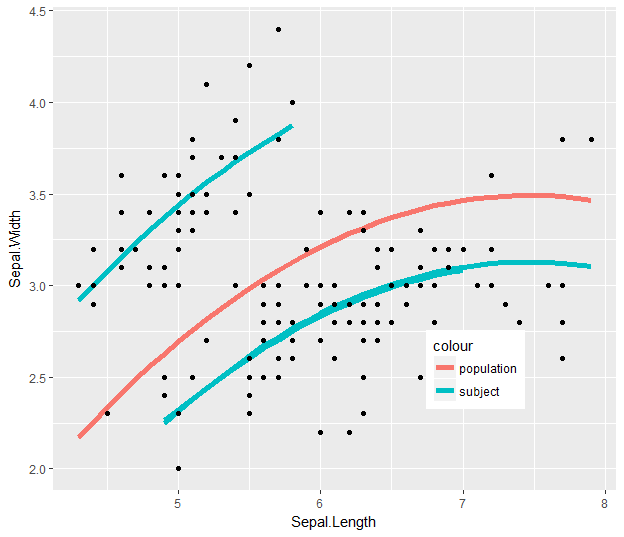
ggplot(augment(fit.iris),
aes(Sepal.Length, Sepal.Width)) +
geom_line(#data = augment(fit.iris) %>% select(-Species),
aes(y = .fixed, color = "population"), size = 2) +
geom_line(aes(y = .fitted, color = "subject", group = Species), size = 2) +
geom_point() +
#facet_wrap(~Species, ncol = 2) +
theme(legend.position = c(0.75,0.25))
Обратите внимание, что я #прокомментировал два высказывания: data = ... а также facet_wrap(...), С этими закомментированными строками вы получите такой вывод:
У тебя твое население ровное (.fixed для фиксированных эффектов) по всему диапазону, а затем у вас есть групповые сглаживания, которые показывают подогнанные значения модели (.fitted), с учетом перехватов на уровне объекта.
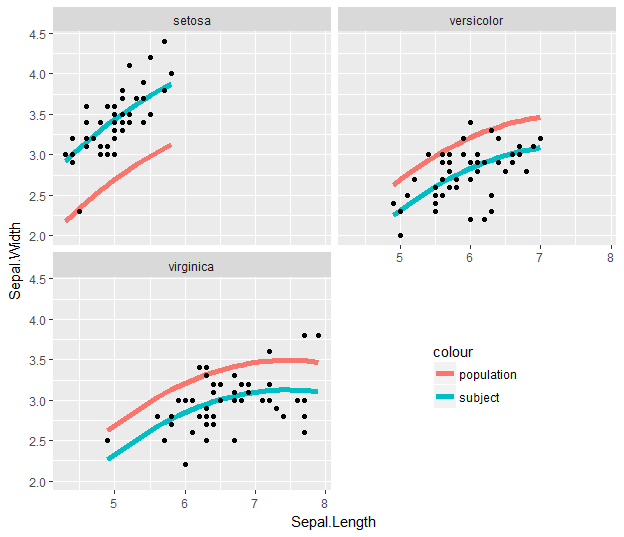
Тогда вы можете показать это в гранях, вынув второй #-комментарий в фрагменте кода:
Это то же самое, но так как подогнанные значения существуют только в пределах диапазона исходных данных для каждой панели уровня объекта, сглаживание популяции усекается только до этого диапазона.
Чтобы обойти это, мы можем удалить первый #-комментарий: