Диаграмма Венна пропорциональна и цветопередача с полупрозрачностью
У меня есть следующий тип подсчета данных.
A 450
B 1800
A and B both 230
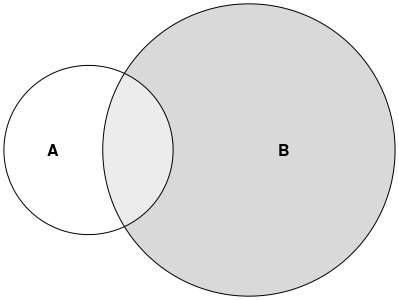
Я хочу создать красочную (возможно, полупрозрачную на пересечениях), как на следующей диаграмме Венна.
Примечание: этот рисунок - пример руки, нарисованной в PowerPoint, и он не в масштабе.
6 ответов
Вот пост, который обсуждает диаграмму Венна из списка кластеров и сопутствующих факторов.
Для простого решения используйте упаковку Веннеулер:
require(venneuler)
v <- venneuler(c(A=450, B=1800, "A&B"=230))
plot(v)
Для более продвинутых и индивидуальных решений проверьте пакет VennDiagram.
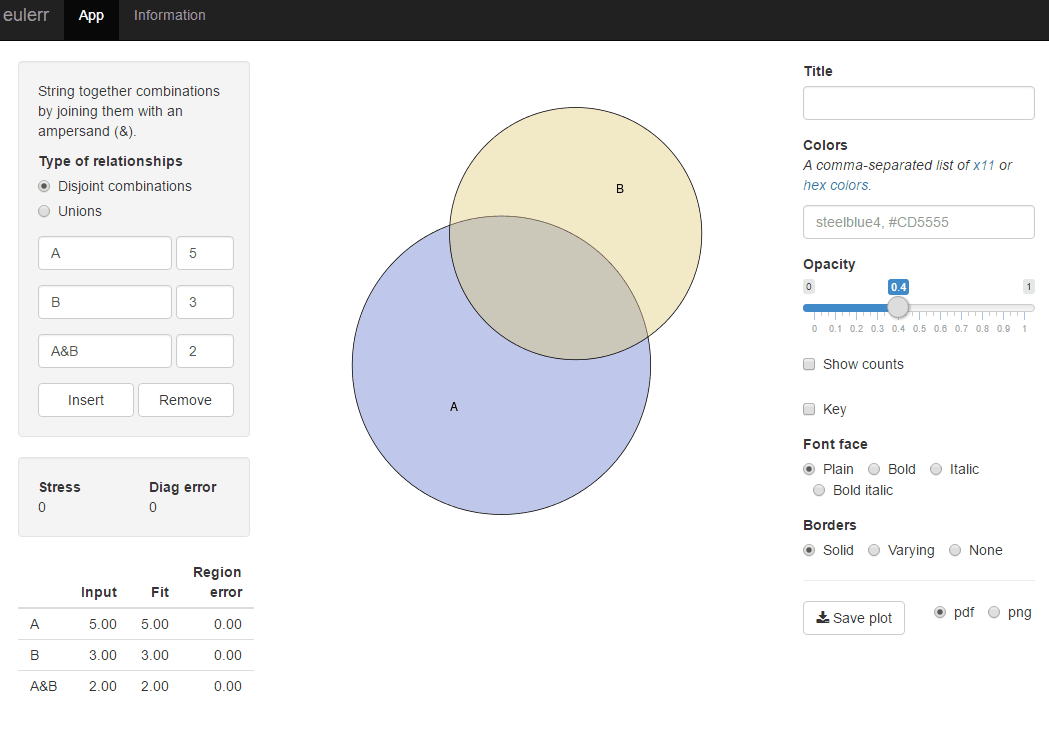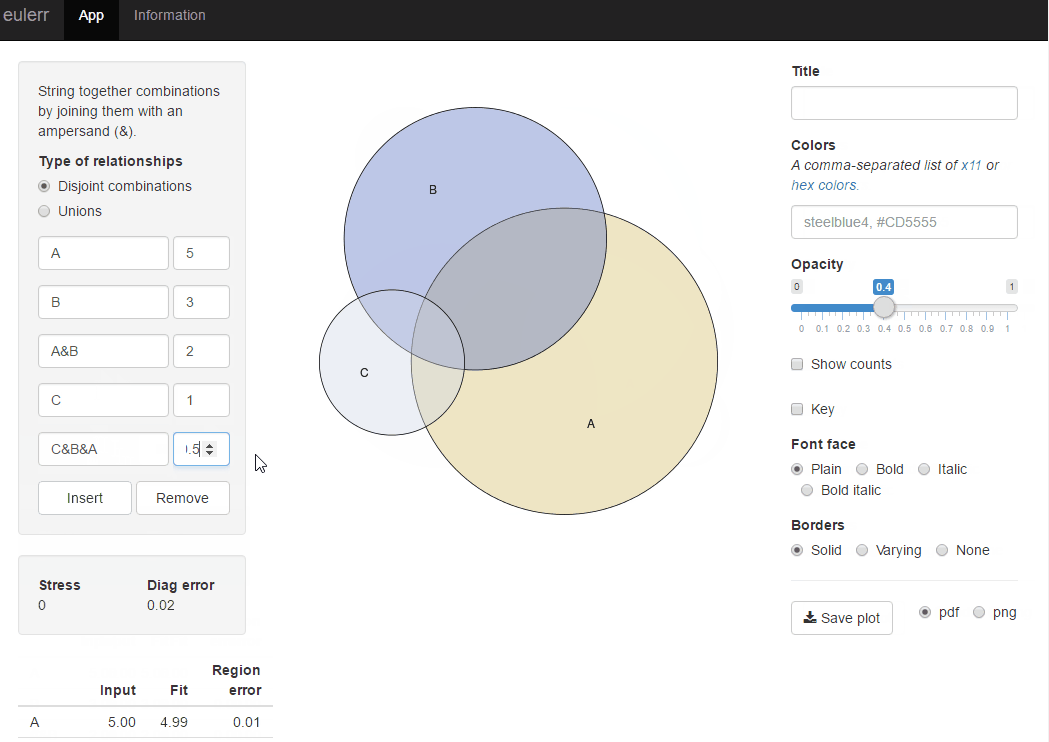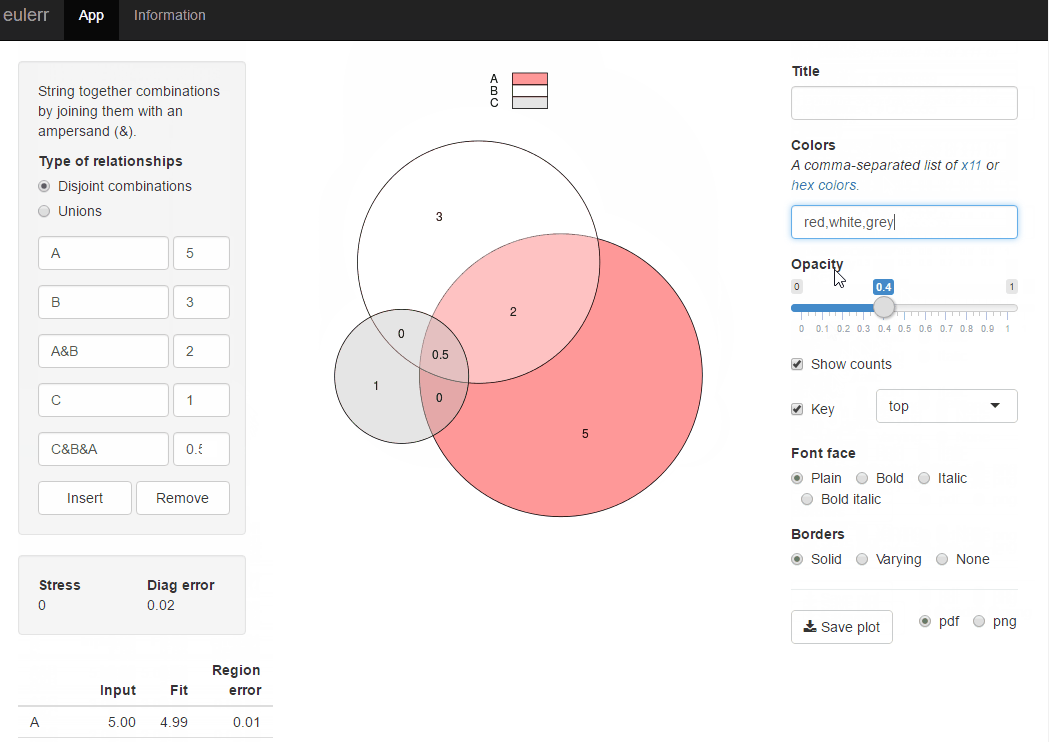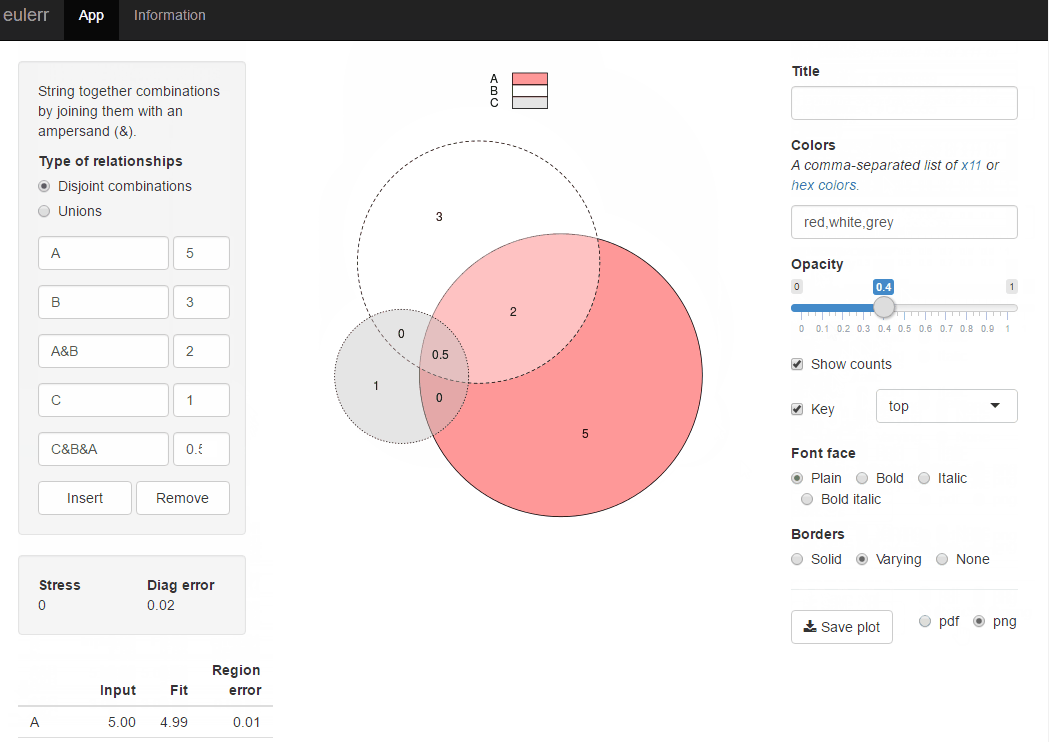
Недавно я опубликовал новый пакет R, eulerr, который делает то, что вы хотите. Это очень похоже на Venneuler, но без его несоответствий.
library(eulerr)
fit <- euler(c(A = 450, B = 1800, "A&B" = 230))
plot(fit)
Или вы можете попробовать блестящее приложение для того же пакета r на http://eulerr.co/
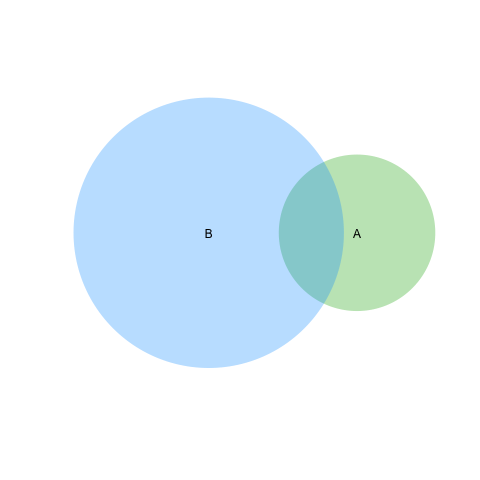
Основываясь на втором ответе Geek On Acid, на втором предложении (еще раз спасибо), я смог бы решить и проблему с линией. Я пишу, если это будет полезно для других googlers!
require(VennDiagram)
venn.diagram(list(B = 1:1800, A = 1571:2020),fill = c("red", "green"),
alpha = c(0.5, 0.5), cex = 2,cat.fontface = 4,lty =2, fontfamily =3,
filename = "trial2.emf");
Хотя это не дает полного ответа на ваш вопрос. Я думал, что это будет полезно для других людей, желающих построить диаграмму Венна. Можно использовать функцию venn() из пакета gplots: http://www.inside-r.org/packages/cran/gplots/docs/venn
## modified slightly from the example given in the documentation
## Example using a list of item names belonging to the
## specified group.
##
require(gplots)
## construct some fake gene names..
oneName <- function() paste(sample(LETTERS,5,replace=TRUE),collapse="")
geneNames <- replicate(1000, oneName())
##
GroupA <- sample(geneNames, 400, replace=FALSE)
GroupB <- sample(geneNames, 750, replace=FALSE)
GroupC <- sample(geneNames, 250, replace=FALSE)
GroupD <- sample(geneNames, 300, replace=FALSE)
venn(list(GrpA=GroupA,GrpB=GroupB,GrpC=GroupC,GrpD=GroupD))
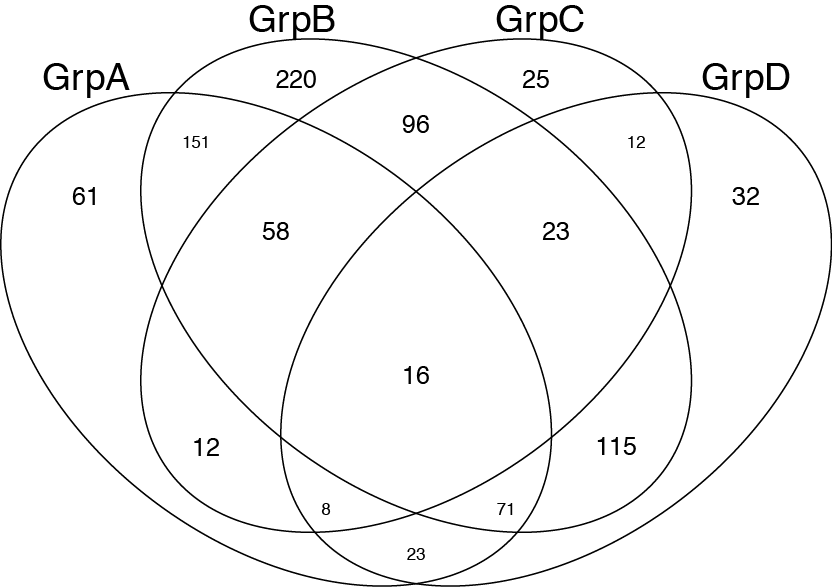 После этого я просто добавляю цвета и прозрачность, используя иллюстратор.
После этого я просто добавляю цвета и прозрачность, используя иллюстратор.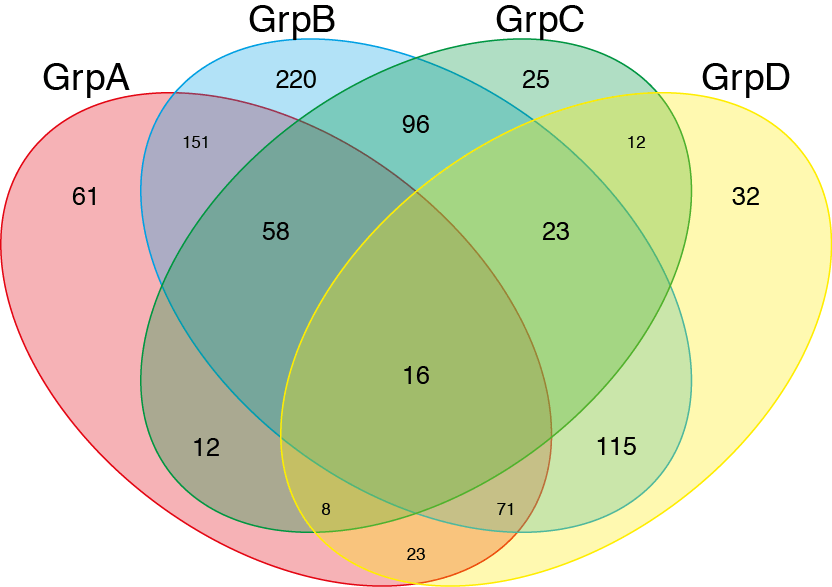
Существует интуитивно понятный и гибкий пропорциональный плоттер, который вы можете скачать и запустить. Найдите его по адресу: http://omics.pnl.gov/software/VennDiagramPlotter.php
а также
jvenn: интерактивный просмотрщик диаграмм Венна - GenoToul Bioinfo: http://bioinfo.genotoul.fr/jvenn/
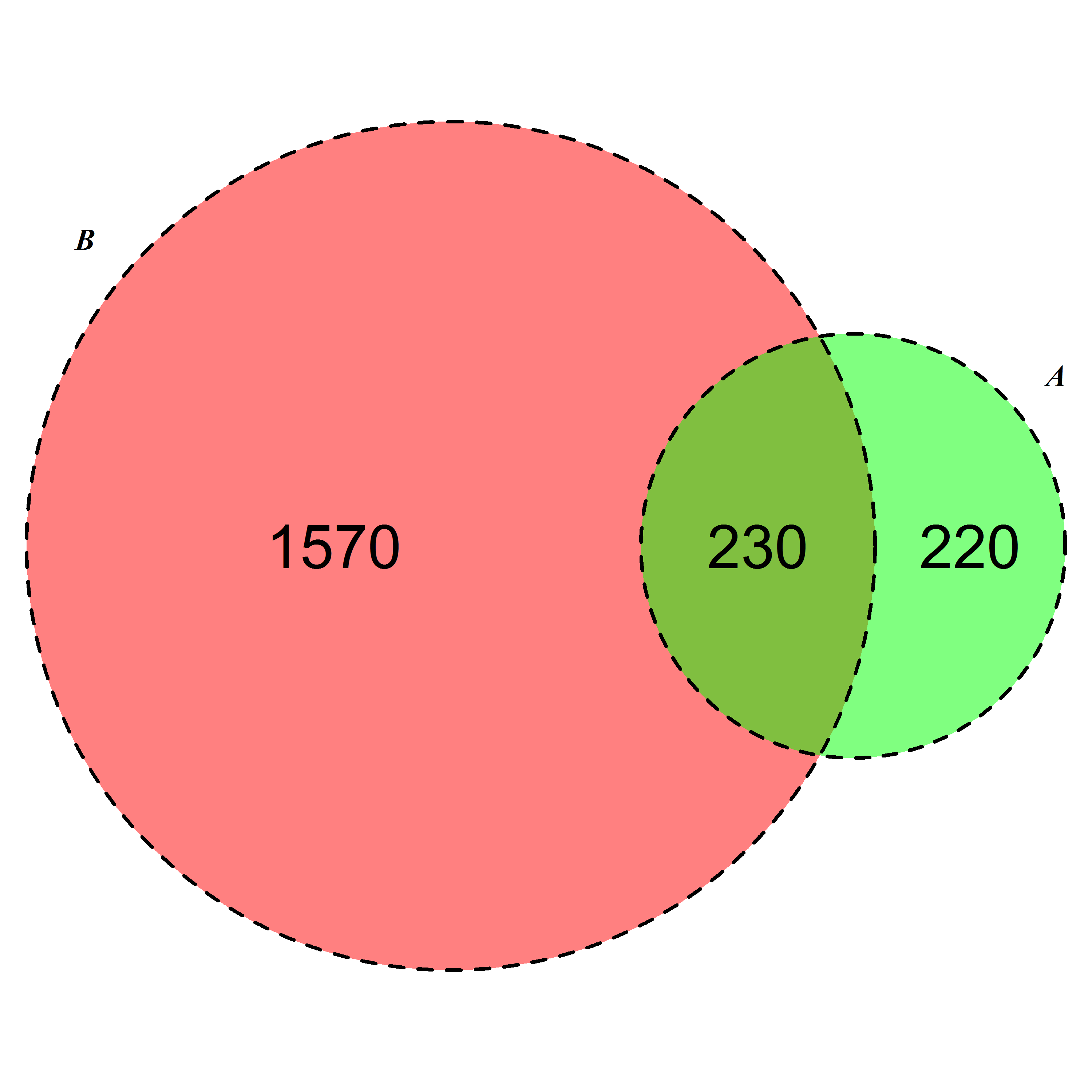
Я знаю, что ОП спрашивает о решении в R, но я хотел бы указать на веб-решение под названием BioVenn. Он берет до 3 списков элементов и рисует диаграмму Венна так, чтобы каждая поверхность была пропорциональна количеству элементов - как этот:
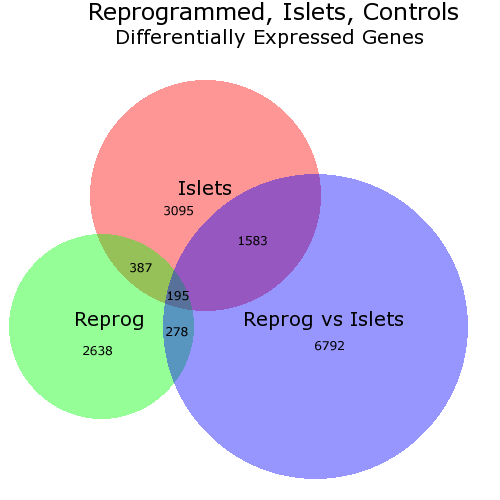
На этой диаграмме я вручную изменил (через PhotoShop) расположение чисел, так как мне не понравились места, выбранные BioVenn. Но вы можете выбрать не иметь номера.
Теоретически списки, используемые с BioVenn, должны состоять из идентификаторов генов, но на практике это не имеет значения - списки просто должны содержать строки.
FWIW: нашел этот пакет для python, который делает то же самое.