ggplot2: гистограмма с нормальной кривой
Я пытался наложить нормальную кривую на мою гистограмму с ggplot 2.
Моя формула:
data <- read.csv (path...)
ggplot(data, aes(V2)) +
geom_histogram(alpha=0.3, fill='white', colour='black', binwidth=.04)
Я попробовал несколько вещей:
+ stat_function(fun=dnorm)
.... ничего не изменилось
+ stat_density(geom = "line", colour = "red")
... дал мне прямую красную линию на оси х.
+ geom_density()
не работает для меня, потому что я хочу сохранить свои значения частоты на оси Y, и не хочу никаких значений плотности.
Какие-либо предложения?
Заранее спасибо за любые советы!
Решение найдено!
+geom_density(aes(y=0.045*..count..), colour="black", adjust=4)
5 ответов
Думаю, я понял:
set.seed(1)
df <- data.frame(PF = 10*rnorm(1000))
ggplot(df, aes(x = PF)) +
geom_histogram(aes(y =..density..),
breaks = seq(-50, 50, by = 10),
colour = "black",
fill = "white") +
stat_function(fun = dnorm, args = list(mean = mean(df$PF), sd = sd(df$PF)))
На это ответили здесь и частично здесь.
Если вы хотите, чтобы ось Y имела счетчики частоты, то нормальную кривую необходимо масштабировать в соответствии с количеством наблюдений и шириной бина.
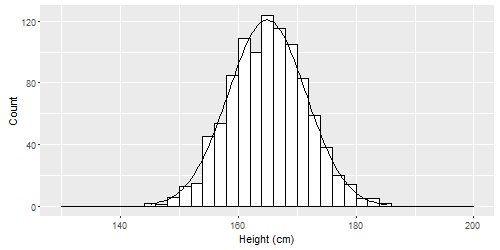
# Simulate some data. Individuals' heights in cm.
n <- 1000
mean <- 165
sd <- 6.6
binwidth <- 2
height <- rnorm(n, mean, sd)
qplot(height, geom = "histogram", breaks = seq(130, 200, binwidth),
colour = I("black"), fill = I("white"),
xlab = "Height (cm)", ylab = "Count") +
# Create normal curve, adjusting for number of observations and binwidth
stat_function(
fun = function(x, mean, sd, n, bw){
dnorm(x = x, mean = mean, sd = sd) * n * bw
},
args = c(mean = mean, sd = sd, n = n, bw = binwidth))
РЕДАКТИРОВАТЬ
Или, для более гибкого подхода, который позволяет использовать фасеты и опирается на подход, перечисленный здесь, создайте отдельный набор данных, содержащий данные для нормальных кривых, и наложите их.
library(plyr)
dd <- data.frame(
predicted = rnorm(720, mean = 2, sd = 2),
state = rep(c("A", "B", "C"), each = 240)
)
binwidth <- 0.5
grid <- with(dd, seq(min(predicted), max(predicted), length = 100))
normaldens <- ddply(dd, "state", function(df) {
data.frame(
predicted = grid,
normal_curve = dnorm(grid, mean(df$predicted), sd(df$predicted)) * length(df$predicted) * binwidth
)
})
ggplot(dd, aes(predicted)) +
geom_histogram(breaks = seq(-3,10, binwidth), colour = "black", fill = "white") +
geom_line(aes(y = normal_curve), data = normaldens, colour = "red") +
facet_wrap(~ state)
Это расширенный комментарий к ответу Дж. Виллимана. Я нашел ответ J очень полезным. Во время игры я нашел способ упростить код. Я не говорю, что это лучший способ, но я думал, что упомяну это.
Обратите внимание, что в ответе Дж. Виллимана содержится счетчик по оси Y и "хак" для масштабирования соответствующего нормального приближения плотности (который в противном случае охватил бы общую площадь 1 и, следовательно, имел бы гораздо более низкий пик).
Суть этого комментария: более простой синтаксис внутри stat_function, передав необходимые параметры эстетической функции, например
aes(x = x, mean = 0, sd = 1, binwidth = 0.3, n = 1000)
Это позволяет избежать необходимости проходить args = в stat_function и, следовательно, более удобный для пользователя. Хорошо, это не очень отличается, но, надеюсь, кто-то найдет это интересным.
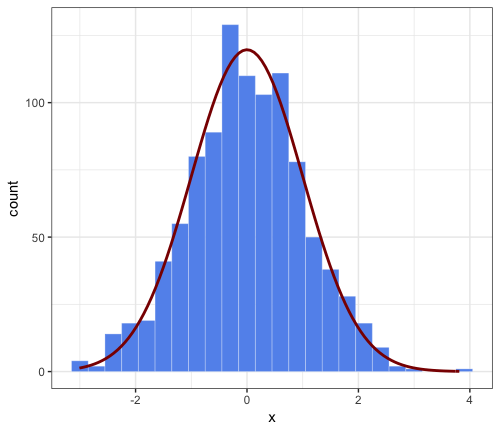
# parameters that will be passed to ``stat_function``
n = 1000
mean = 0
sd = 1
binwidth = 0.3 # passed to geom_histogram and stat_function
set.seed(1)
df <- data.frame(x = rnorm(n, mean, sd))
ggplot(df, aes(x = x, mean = mean, sd = sd, binwidth = binwidth, n = n)) +
theme_bw() +
geom_histogram(binwidth = binwidth,
colour = "white", fill = "cornflowerblue", size = 0.1) +
stat_function(fun = function(x) dnorm(x, mean = mean, sd = sd) * n * binwidth,
color = "darkred", size = 1)
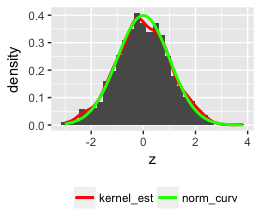
Этот код должен сделать это:
set.seed(1)
z <- rnorm(1000)
qplot(z, geom = "blank") +
geom_histogram(aes(y = ..density..)) +
stat_density(geom = "line", aes(colour = "bla")) +
stat_function(fun = dnorm, aes(x = z, colour = "blabla")) +
scale_colour_manual(name = "", values = c("red", "green"),
breaks = c("bla", "blabla"),
labels = c("kernel_est", "norm_curv")) +
theme(legend.position = "bottom", legend.direction = "horizontal")
Примечание: я использовал qplot, но вы можете использовать более универсальный ggplot.
Вот информированная версия tidyverse:
Настраивать
library(tidyverse)
Некоторые данные
d <- read_csv("https://vincentarelbundock.github.io/Rdatasets/csv/openintro/speed_gender_height.csv")
Подготовка данных
Мы будем использовать «общую» гистограмму для всей выборки, для этого нам нужно удалить информацию о группировке из данных.
d2 <-
d |>
select(-gender)
Вот набор данных со сводными данными:
d_summary <-
d %>%
group_by(gender) %>%
summarise(height_m = mean(height, na.rm = T),
height_sd = sd(height, na.rm = T))
d_summary
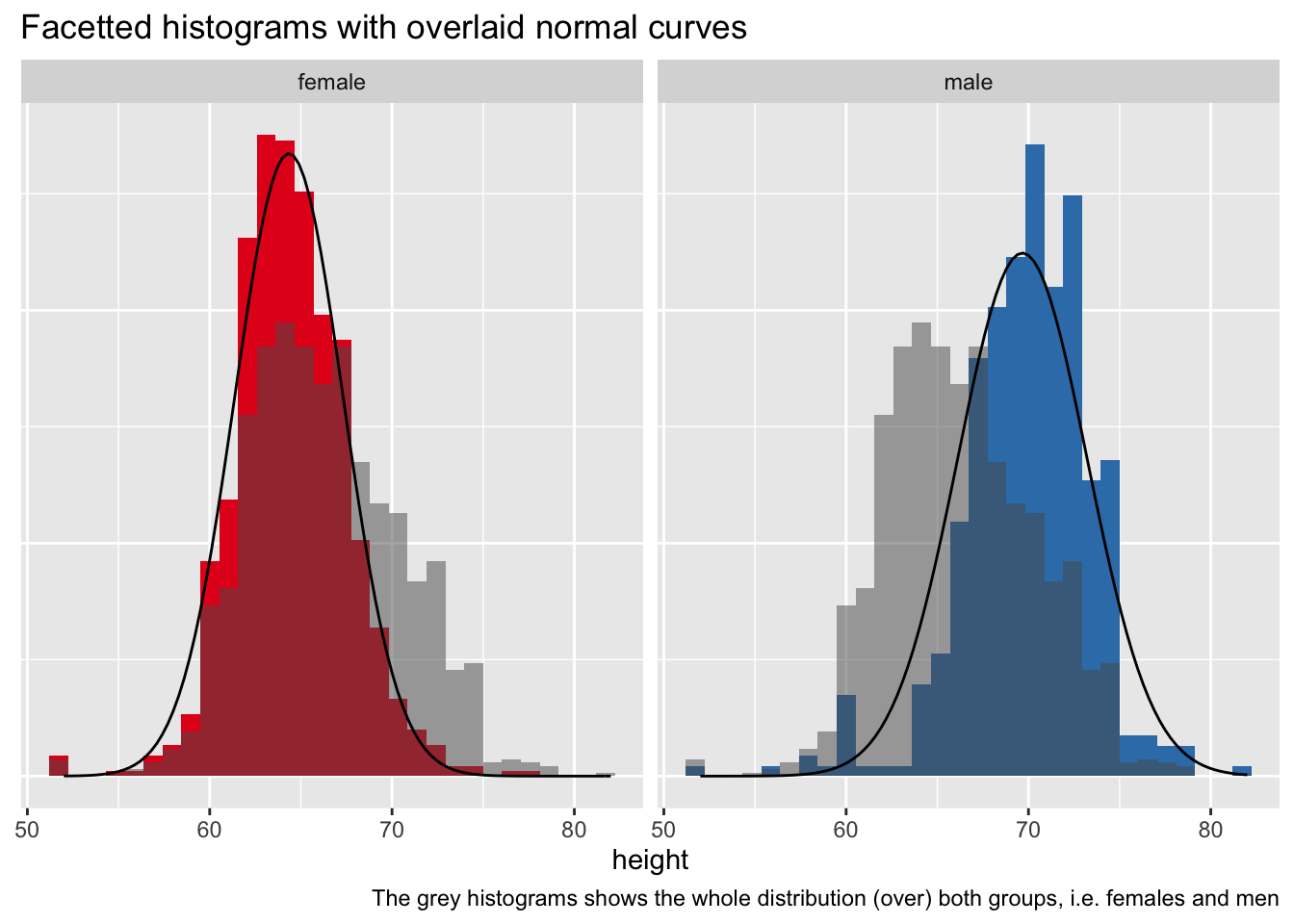
Постройте это
d %>%
ggplot() +
aes() +
geom_histogram(aes(y = ..density.., x = height, fill = gender)) +
facet_wrap(~ gender) +
geom_histogram(data = d2, aes(y = ..density.., x = height),
alpha = .5) +
stat_function(data = d_summary %>% filter(gender == "female"),
fun = dnorm,
#color = "red",
args = list(mean = filter(d_summary,
gender == "female")$height_m,
sd = filter(d_summary,
gender == "female")$height_sd)) +
stat_function(data = d_summary %>% filter(gender == "male"),
fun = dnorm,
#color = "red",
args = list(mean = filter(d_summary,
gender == "male")$height_m,
sd = filter(d_summary,
gender == "male")$height_sd)) +
theme(legend.position = "none",
axis.title.y = element_blank(),
axis.text.y = element_blank(),
axis.ticks.y = element_blank()) +
labs(title = "Facetted histograms with overlaid normal curves",
caption = "The grey histograms shows the whole distribution (over) both groups, i.e. females and men") +
scale_fill_brewer(type = "qual", palette = "Set1")