Используя stat_function и facet_wrap вместе в ggplot2 в R
Я пытаюсь построить данные типа решетки с помощью ggplot2, а затем наложить нормальное распределение на выборочные данные, чтобы показать, насколько далеки от нормы лежат базовые данные. Я бы хотел, чтобы обычный dist был сверху, чтобы иметь то же значение и stdev, что и на панели.
вот пример:
library(ggplot2)
#make some example data
dd<-data.frame(matrix(rnorm(144, mean=2, sd=2),72,2),c(rep("A",24),rep("B",24),rep("C",24)))
colnames(dd) <- c("x_value", "Predicted_value", "State_CD")
#This works
pg <- ggplot(dd) + geom_density(aes(x=Predicted_value)) + facet_wrap(~State_CD)
print(pg)
Все это прекрасно работает и дает хороший трехпанельный график данных. Как мне добавить нормальный dist сверху? Кажется, я бы использовал stat_function, но это не удается:
#this fails
pg <- ggplot(dd) + geom_density(aes(x=Predicted_value)) + stat_function(fun=dnorm) + facet_wrap(~State_CD)
print(pg)
Похоже, что stat_function не ладит с функцией facet_wrap. Как мне заставить этих двоих хорошо играть?
------------РЕДАКТИРОВАТЬ---------
Я попытался объединить идеи из двух ответов ниже, и я все еще не там:
используя комбинацию обоих ответов, я могу взломать это:
library(ggplot)
library(plyr)
#make some example data
dd<-data.frame(matrix(rnorm(108, mean=2, sd=2),36,2),c(rep("A",24),rep("B",24),rep("C",24)))
colnames(dd) <- c("x_value", "Predicted_value", "State_CD")
DevMeanSt <- ddply(dd, c("State_CD"), function(df)mean(df$Predicted_value))
colnames(DevMeanSt) <- c("State_CD", "mean")
DevSdSt <- ddply(dd, c("State_CD"), function(df)sd(df$Predicted_value) )
colnames(DevSdSt) <- c("State_CD", "sd")
DevStatsSt <- merge(DevMeanSt, DevSdSt)
pg <- ggplot(dd, aes(x=Predicted_value))
pg <- pg + geom_density()
pg <- pg + stat_function(fun=dnorm, colour='red', args=list(mean=DevStatsSt$mean, sd=DevStatsSt$sd))
pg <- pg + facet_wrap(~State_CD)
print(pg)
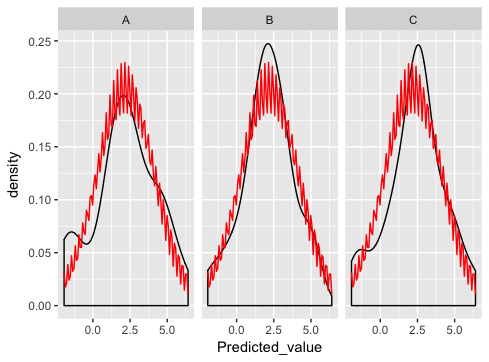
что действительно близко... за исключением того, что что-то не так с нормальным заговором dist:
что я тут не так делаю?
6 ответов
stat_function предназначен для наложения одной и той же функции на каждой панели. (Нет очевидного способа сопоставления параметров функции с различными панелями).
Как предлагает Ян, лучший способ - это самостоятельно сгенерировать нормальные кривые и представить их в виде отдельного набора данных (здесь вы ошиблись раньше - объединение просто не имеет смысла для этого примера, и если вы посмотрите внимательно, вы вот почему вы получаете странный пилообразный рисунок).
Вот как бы я решил проблему:
dd <- data.frame(
predicted = rnorm(72, mean = 2, sd = 2),
state = rep(c("A", "B", "C"), each = 24)
)
grid <- with(dd, seq(min(predicted), max(predicted), length = 100))
normaldens <- ddply(dd, "state", function(df) {
data.frame(
predicted = grid,
density = dnorm(grid, mean(df$predicted), sd(df$predicted))
)
})
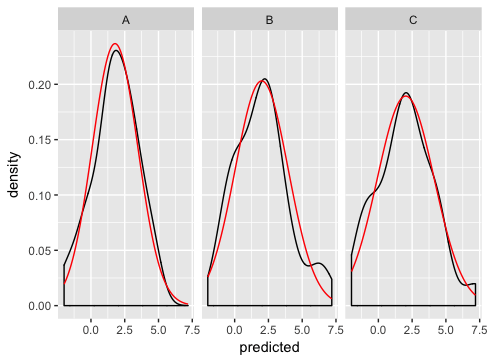
ggplot(dd, aes(predicted)) +
geom_density() +
geom_line(aes(y = density), data = normaldens, colour = "red") +
facet_wrap(~ state)
Первоначально опубликованный как ответ на этот вопрос , мне также было предложено поделиться своим решением здесь.
Меня тоже разочаровало наложение теоретических плотностей на эмпирические данные, поэтому я написал функцию, автоматизирующую этот процесс. С 2009 года, когда этот вопрос был впервые задан, ggplot2 значительно расширил расширяемость, поэтому я поместил его в пакет расширения на github.
library(ggplot2)
library(ggh4x)
set.seed(0)
# Make the example data
dd <- data.frame(matrix(rnorm(144, mean=2, sd=2),72,2),
c(rep("A",24),rep("B",24),rep("C",24)))
colnames(dd) <- c("x_value", "Predicted_value", "State_CD")
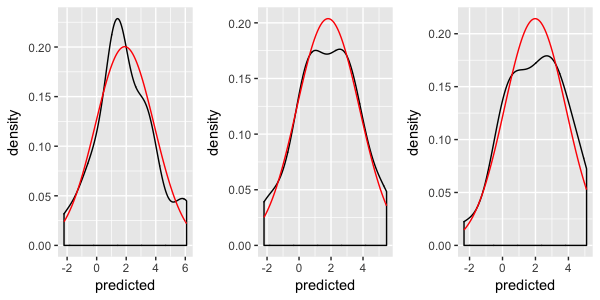
ggplot(dd, aes(Predicted_value)) +
geom_density() +
stat_theodensity(colour = "red") +
facet_wrap(~ State_CD)

Создано 28 января 2021 г. пакетом reprex (v0.3.0)
Если вы хотите использовать ggformula, то это довольно просто. (Также возможно смешивать, сопоставлять и использовать ggformula только для наложения дистрибутива, но я проиллюстрирую полный подход к ggformula.)
library(ggformula)
theme_set(theme_bw())
gf_dens( ~ Sepal.Length | Species, data = iris) %>%
gf_fitdistr(color = "red") %>%
gf_fitdistr(dist = "gamma", color = "blue")

Создано в 2019-01-15 пакетом представлением (v0.2.1)
Я думаю, что вам нужно предоставить больше информации. Это похоже на работу:
pg <- ggplot(dd, aes(Predicted_value)) ## need aesthetics in the ggplot
pg <- pg + geom_density()
## gotta provide the arguments of the dnorm
pg <- pg + stat_function(fun=dnorm, colour='red',
args=list(mean=mean(dd$Predicted_value), sd=sd(dd$Predicted_value)))
## wrap it!
pg <- pg + facet_wrap(~State_CD)
pg
Мы предоставляем одинаковое среднее и параметр SD для каждой панели. Получение панели специальных средств и стандартных отклонений оставлено в качестве упражнения для читателя *;)
'*' Другими словами, не уверен, как это можно сделать...
Если вы не хотите генерировать линейный график нормального распределения "вручную", по-прежнему использовать stat_function и показывать графики бок о бок - тогда вы можете рассмотреть возможность использования функции "multiplot", опубликованной в "Cookbook for R" в качестве альтернативы facet_wrap. Вы можете скопировать код мультиплота в ваш проект отсюда.
После того, как вы скопируете код, сделайте следующее:
# Some fake data (copied from hadley's answer)
dd <- data.frame(
predicted = rnorm(72, mean = 2, sd = 2),
state = rep(c("A", "B", "C"), each = 24)
)
# Split the data by state, apply a function on each member that converts it into a
# plot object, and return the result as a vector.
plots <- lapply(split(dd,dd$state),FUN=function(state_slice){
# The code here is the plot code generation. You can do anything you would
# normally do for a single plot, such as calling stat_function, and you do this
# one slice at a time.
ggplot(state_slice, aes(predicted)) +
geom_density() +
stat_function(fun=dnorm,
args=list(mean=mean(state_slice$predicted),
sd=sd(state_slice$predicted)),
color="red")
})
# Finally, present the plots on 3 columns.
multiplot(plotlist = plots, cols=3)
Я думаю, что вам лучше всего провести линию вручную с помощью geom_line.
dd<-data.frame(matrix(rnorm(144, mean=2, sd=2),72,2),c(rep("A",24),rep("B",24),rep("C",24)))
colnames(dd) <- c("x_value", "Predicted_value", "State_CD")
dd$Predicted_value<-dd$Predicted_value*as.numeric(dd$State_CD) #make different by state
##Calculate means and standard deviations by level
means<-as.numeric(by(dd[,2],dd$State_CD,mean))
sds<-as.numeric(by(dd[,2],dd$State_CD,sd))
##Create evenly spaced evaluation points +/- 3 standard deviations away from the mean
dd$vals<-0
for(i in 1:length(levels(dd$State_CD))){
dd$vals[dd$State_CD==levels(dd$State_CD)[i]]<-seq(from=means[i]-3*sds[i],
to=means[i]+3*sds[i],
length.out=sum(dd$State_CD==levels(dd$State_CD)[i]))
}
##Create normal density points
dd$norm<-with(dd,dnorm(vals,means[as.numeric(State_CD)],
sds[as.numeric(State_CD)]))
pg <- ggplot(dd, aes(Predicted_value))
pg <- pg + geom_density()
pg <- pg + geom_line(aes(x=vals,y=norm),colour="red") #Add in normal distribution
pg <- pg + facet_wrap(~State_CD,scales="free")
pg