Как настроить вычисление перекрытия эллипсов в перекрытии {siar}, чтобы оно соответствовало эллипсоидам с нормальной вероятностью в ggbiplot?
Возвращаясь к этой теме, я вычислял пересечение между двумя эллипсами, например, объем пересечения между версиколором и вирджинкой: 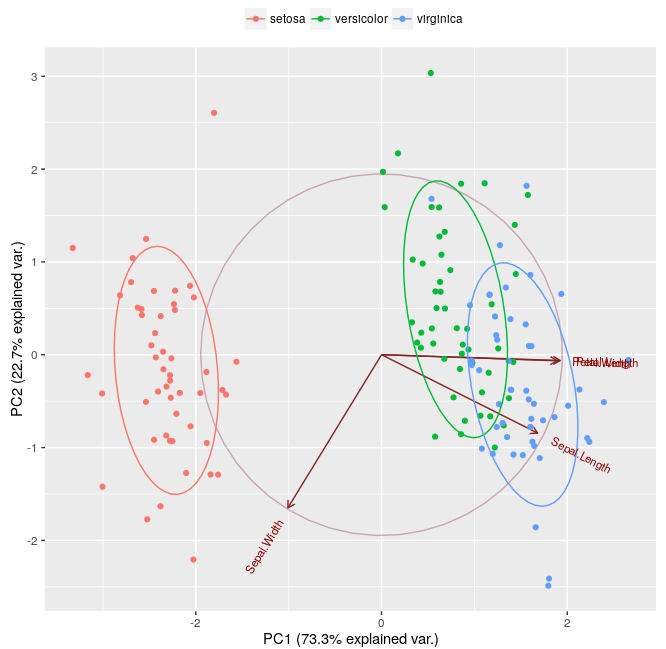 используя следующее mwe:
используя следующее mwe:
data(iris)
log.ir <- log(iris[, 1:4])
ir.species <- iris[, 5]
ir.pca <- prcomp(log.ir, center = TRUE, scale. = TRUE)
library(ggbiplot)
g <- ggbiplot(ir.pca, obs.scale = 1, var.scale = 1,
groups = ir.species, ellipse = TRUE,
circle = TRUE)
g <- g + scale_color_discrete(name = '')
g <- g + theme(legend.direction = 'horizontal',
legend.position = 'top')
print(g)
Затем я вычисляю эллипсоиды, используя перекрытие {siar} следующим образом:
library(siar)
setosa <- ir.pca$x[ir.species=="setosa",]
versicolor <- ir.pca$x[ir.species=="versicolor",]
virginica <- ir.pca$x[ir.species=="virginica",]
tmp <- overlap(versicolor[,1], versicolor[,2], virginica[,1], virginica[,2], steps = 5)
virginica.percentage <- round(x=(tmp$overlap/tmp$area2*100), digits = 2)
versicolor.percentage <- round(x=(tmp$overlap/tmp$area1*100), digits = 2)
> virginica.percentage [1] 1.54
> versicolor.percentage[1] 1.56
что, по-видимому, намного меньше, чем показано на рисунке 2 выше.
Как показывает код ggbiplot в строке 50, ggbiplot вычисляет эллипсоиды с нормальной вероятностью с вероятностью "ellipse.prob = 0.68" по умолчанию, что используется на графике выше.
Из-за документации overlap{siar} вычисляет эллипсы как "стандартные эллипсы с коррекцией малого размера выборки".
Поэтому я спрашивал себя, как можно изменить перекрытие {siar}, чтобы вычислить эллипсоид с нормальной вероятностью, подобный тому, который был построен / рассчитан в ggbiplot.
1 ответ
Последняя версия SIBER, доступная по https://github.com/AndrewLJackson/SIBER включает две новые функции для расчета перекрытия эллипсов определенного пользователем размера (т. е. различных квантилей).
maxLikOverlap()
а также
bayesianOverlap()
Я предлагаю вам установить из главной ветки с помощью
# install.packages("devtools") # install if necessary
devtools::install_github("andrewljackson/SIBER@master",
build_vingettes = TRUE)
library(SIBER)
И следите за виньеткой "Эллипс-Перекрытие". Если виньетки не устанавливаются для вас, вы можете загрузить их прямо из репозитория github https://github.com/AndrewLJackson/SIBER/tree/master/vignettes