Генерация идеально нормально распределенной выборки размером n в R
Я хотел бы сгенерировать выборку со средним значением = 0, sd = 1 и размером n = 100, распределение которого является нормальным, насколько это возможно. Использование rnorm само по себе возвращает большую изменчивость.
Единственный способ, который я нашел, - усреднить несколько норм.
rowMeans(replicate(10000, sort(rnorm(100, 0, 1))))
Это возвращает довольно удовлетворительный результат, но я не уверен, что это самый эффективный способ сделать это.
РЕДАКТИРОВАТЬ:
Я не хочу, чтобы среднее значение и sd были строго равны 0 и 1, а скорее, чтобы распределение выглядело как нормальное распределение (при построении кривой плотности).
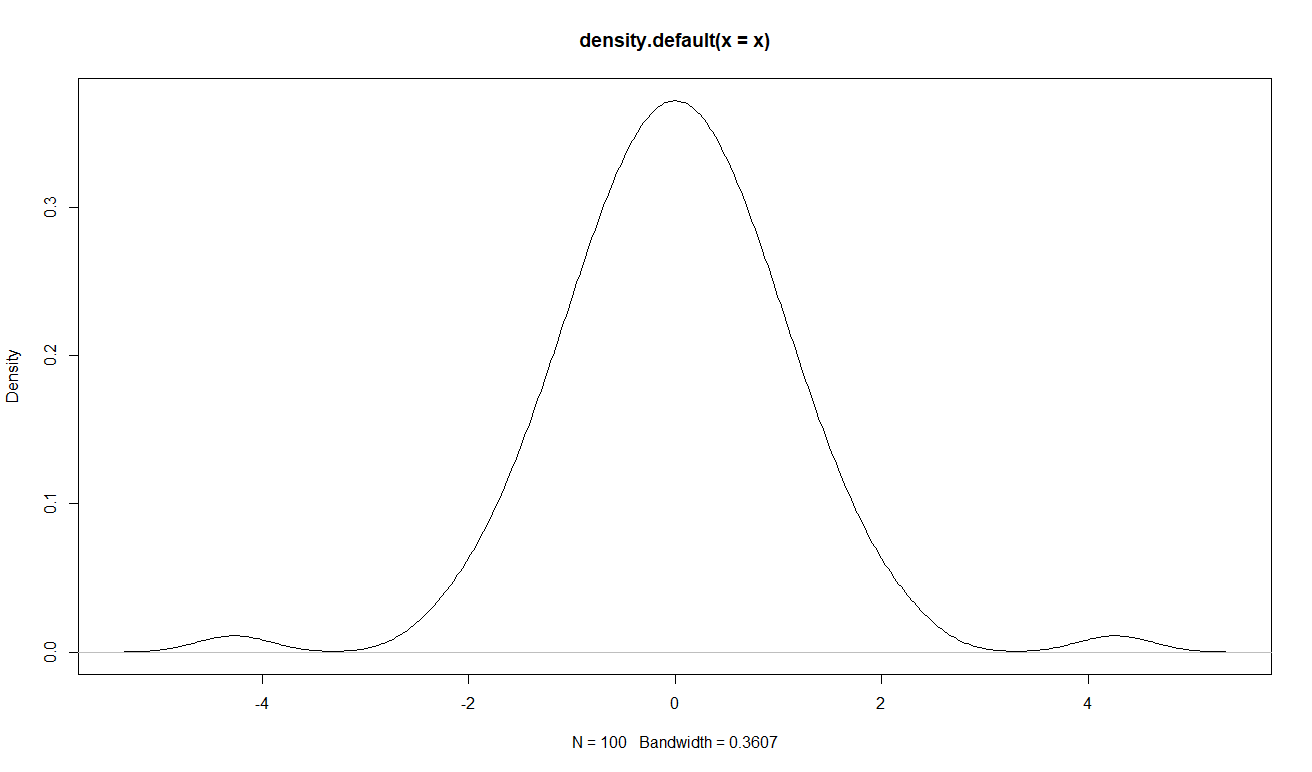
Кажется, что метод qnorm работает хуже, чем "средний" метод:
# qnorm method
x <- qnorm(seq(.00001, .99999, length.out = 100), mean=0, sd=1)
plot(density(x))
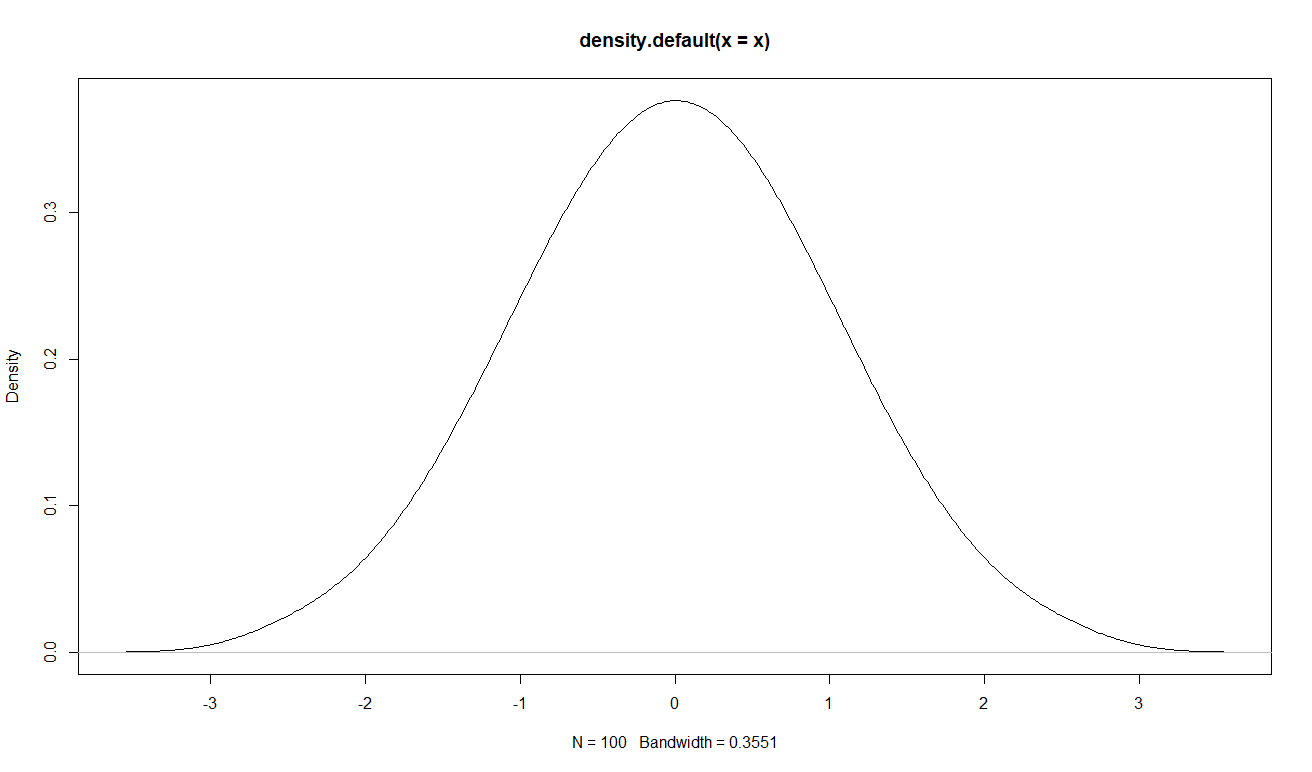
# average method
x <- rowMeans(replicate(10000, sort(rnorm(100, mean=0, sd=1))))
plot(density(x))
Я был бы доволен детерминированным решением, возвращающим результаты, близкие к среднему, более эффективным способом.
РЕДАКТИРОВАТЬ 2: Возможное решение
Основываясь на ответах, кажется, что работает следующее, корректируя границы относительно n:
x <- qnorm(seq(1/n, 1-1/n, length.out = n), mean=0, sd=1)
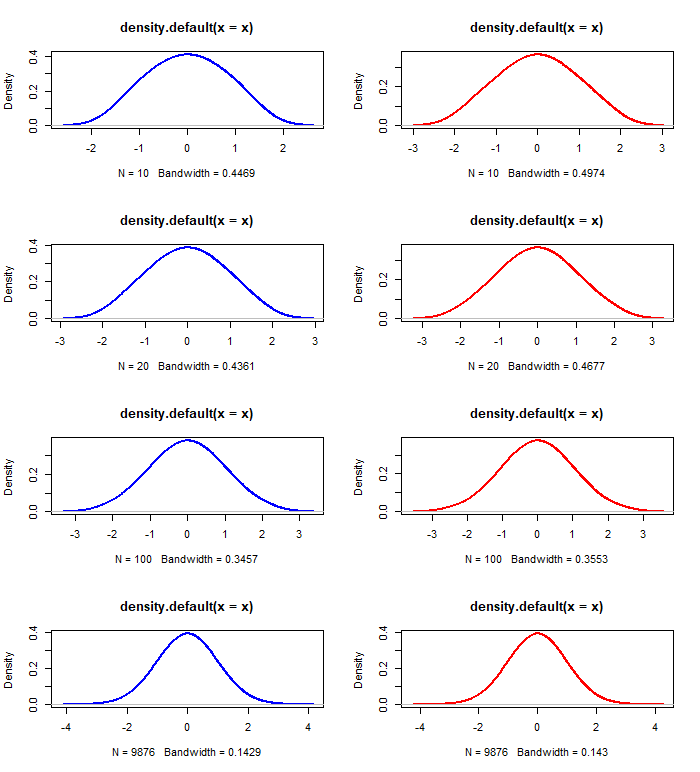
Ниже приведено сравнение методов qnorm и среднего для разных значений n:
par(mfrow=c(6,2))
for(n in c(10, 20, 100, 500, 1000, 9876)){
x <- qnorm(seq(1/n, 1-1/n, length.out = n), mean=0, sd=1)
plot(density(x), col="blue", lwd=2)
x <- rowMeans(replicate(10000, sort(rnorm(n, mean=0, sd=1))))
plot(density(x), col="red", lwd=2)
}
3 ответа
Вы пытаетесь создать 100 чисел с приблизительным нормальным распределением со средним точно нулем и sd ровно одним? Сделай это:
Начните примерно:
> X = rnorm(100)
Сдвинь их:
> X = X-mean(X)
Масштабировать их:
> X = X/sd(X)
Проверь это:
> mean(X)
[1] -7.223497e-18
достаточно близко
> sd(X)
[1] 1
грохнуть
Это то же самое, что scale функция делает:
> X = rnorm(100)
> mean(X)
[1] -0.007667039
> sd(X)
[1] 0.9336842
> sx = scale(X)
> mean(sx)
[1] 1.437056e-17
> sd(sx)
[1] 1
Если вы хотите детерминистическое решение, это должно работать
qnorm(seq(0.01, 0.99, length.out = 100))
Обратите внимание, что qnorm(0) дает $-\infty$ и qnorm(1) $\infty$, поэтому вам нужно найти разумные границы.
За n=100границы 0,01 и 0,99 работают лучше всего. Если вы хотите, чтобы границы для детерминированного решения находились дальше, вам нужно увеличить n,
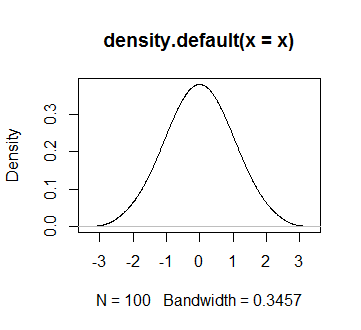
Вы можете использовать пакет bayestestR:
library(bayestestR)
x <- rnorm_perfect(n = 100, mean = 0, sd = 1)
plot(density(x))
Последовательность с низким расхождением? халтон, форе, соболь, хаммерсли: пример:
library(randtoolbox)
sequence <-sobol(n=100, dim = 1, init = TRUE, scrambling = 0, seed = 4711, normal = FALSE)
mean(sequence)
[1] 0.4982031
sd(sequence)
[1] 0.2860574
#trial with prng
set.seed(1)
sequence2 <- runif(100)
mean(sequence2)
[1] 0.5178471
sd(sequence2)
[1] 0.2675848
с таким же количеством точек последовательность с низким расхождением лучше, чем генератор псевдослучайного числа, имейте в виду, что для равномерной случайной выборки истинное среднее значение равно 0,5, sd равно 0,2886751 (sqrt(1/12)), посмотрите на числа.
(mean(sequence) - 0.5)/0.5 # -0.0008984375
(mean(sequence2) - 0.5)/0.5 # -0.008923532
(sd(sequence) - sqrt(1/12))*sqrt(12)
[1] -0.009067992
(sd(sequence2) - sqrt(1/12))*sqrt(12)
[1] -0.07305918
~ В 10 раз лучше, попробуйте с другим семенем, если вы не верите в это
ks.test(sequence,"runif")
One-sample Kolmogorov-Smirnov test
data: sequence
D = 0.96268, p-value < 2.2e-16
alternative hypothesis: two-sided
> ks.test(sequence2,"runif")
One-sample Kolmogorov-Smirnov test
data: sequence2
D = 0.93956, p-value < 2.2e-16
alternative hypothesis: two-sided
Теперь немного балансировки:
sequence <- c(sequence, 1.0 - sequence) #balancing the mean = use antithetics
#or if you want (sequence <- sequence - mean(sequence))
normal_sample <- qnorm(sequence)
normal_sample <- normal_sample/sd(normal_sample)
plot(normal_sample)