График сгруппированных значений кадра данных в R
У меня есть следующий фрейм данных:
stat mTADs DE_genes
5267 -5.452819 chr2:167337500-167447500 chr2:167318145-167341673:+
5268 4.114012 chr6:41532500-41642500 chr6:41555481-41570508:+
5269 9.812369 chr10:18157500-18262500 chr10:18259929-18265882:-
5270 3.371969 chr17:40957500-41062500 chr17:41060000-41071996:-
5271 4.576930 chr17:40957500-41062500 chr17:41012431-41017507:-
5272 2.952151 chr11:72251250-72352500 chr11:72254857-72265270:+
5273 -3.349795 chr1:174307500-174407500 chr1:174405489-174408706:+
5274 -2.685897 chr13:100777500-100877500 chr13:100787949-100874025:-
5275 2.865269 chr13:100777500-100877500 chr13:100718488-100785594:-
5276 6.436959 chr4:150417500-150517500 chr4:150377761-150418774:-
5277 2.622196 chr7:6072500-6162500 chr7:6123828-6142951:+
5278 -5.605531 chr11:48597500-48682500 chr11:48675470-48685185:-
5279 3.554733 chr11:48597500-48682500 chr11:48639642-48665711:+
5280 4.399655 chr11:48597500-48682500 chr11:48638848-48640157:-
Как вы видите, некоторые DE_genes попадают в один и тот же mTAD. Я хотел бы построить для всех DE_genes их значения статов и сгруппировать их по mTAD. Я думал о том, чтобы сделать это как горизонтальную полосу, имеющую на яксисах гены, а на оси х значения статистики и сгруппировать их по TAD, но сначала я не знаю, как это сделать, а во-вторых, я подумал, что тепловая карта может быть лучше вариант. Есть ли способ сделать это в R? Всего у меня 1700 мТД, и я хотел бы посмотреть, есть ли какие-либо закономерности в данных.
Большое спасибо, Димитрис
1 ответ
Вы можете рассмотреть отсортированную точечную диаграмму вместо диаграммы.
> thing
ID stat mTADs DE_genes
1 5267 -5.452819 chr2:167337500-167447500 chr2:167318145-167341673:+
2 5268 4.114012 chr6:41532500-41642500 chr6:41555481-41570508:+
3 5269 9.812369 chr10:18157500-18262500 chr10:18259929-18265882:-
4 5270 3.371969 chr17:40957500-41062500 chr17:41060000-41071996:-
5 5271 4.576930 chr17:40957500-41062500 chr17:41012431-41017507:-
6 5272 2.952151 chr11:72251250-72352500 chr11:72254857-72265270:+
7 5273 -3.349795 chr1:174307500-174407500 chr1:174405489-174408706:+
8 5274 -2.685897 chr13:100777500-100877500 chr13:100787949-100874025:-
9 5275 2.865269 chr13:100777500-100877500 chr13:100718488-100785594:-
10 5276 6.436959 chr4:150417500-150517500 chr4:150377761-150418774:-
11 5277 2.622196 chr7:6072500-6162500 chr7:6123828-6142951:+
12 5278 -5.605531 chr11:48597500-48682500 chr11:48675470-48685185:-
13 5279 3.554733 chr11:48597500-48682500 chr11:48639642-48665711:+
14 5280 4.399655 chr11:48597500-48682500 chr11:48638848-48640157:-
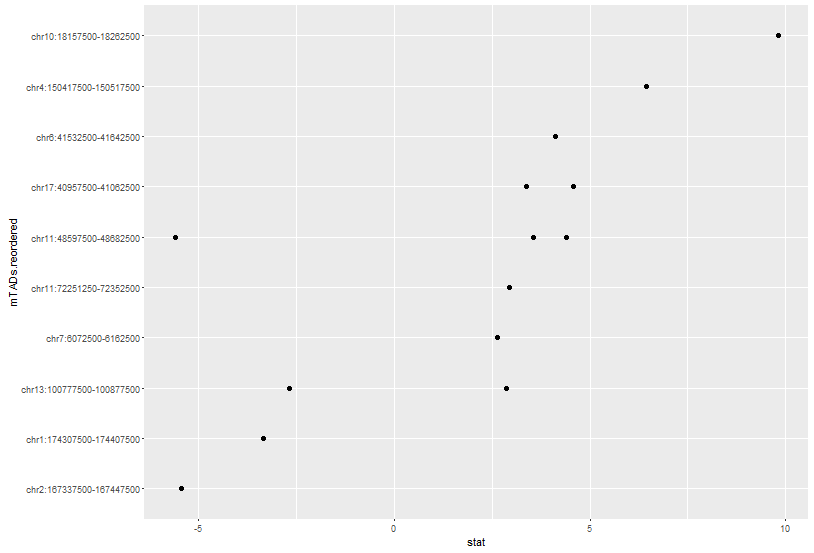
Сначала мы возьмем медианы по mTAD.
medians.of.stat.by.mTADs<-aggregate(stat~mTADs,data=thing,FUN=median)
names(medians.of.stat.by.mTADs)[2]<-"median stat for mTAD"
Теперь объедините эти медианы с исходным фреймом данных и создайте коэффициент, упорядочив отсортированные stat ценности.
thing<-merge(thing,medians.of.stat.by.mTADs,all.x = T,by="mTADs")
thing$mTADs.reordered <-factor(thing$mTADs, levels=thing[order(thing$`median stat for mTAD`), "mTADs"])
Это выдаст предупреждение из-за дублированных уровней, но, похоже, работает.
> thing
mTADs ID stat DE_genes median stat for mTAD mTADs.reordered
1 chr1:174307500-174407500 5273 -3.349795 chr1:174405489-174408706:+ -3.349795 chr1:174307500-174407500
2 chr10:18157500-18262500 5269 9.812369 chr10:18259929-18265882:- 9.812369 chr10:18157500-18262500
3 chr11:48597500-48682500 5278 -5.605531 chr11:48675470-48685185:- 3.554733 chr11:48597500-48682500
4 chr11:48597500-48682500 5279 3.554733 chr11:48639642-48665711:+ 3.554733 chr11:48597500-48682500
5 chr11:48597500-48682500 5280 4.399655 chr11:48638848-48640157:- 3.554733 chr11:48597500-48682500
6 chr11:72251250-72352500 5272 2.952151 chr11:72254857-72265270:+ 2.952151 chr11:72251250-72352500
7 chr13:100777500-100877500 5274 -2.685897 chr13:100787949-100874025:- 0.089686 chr13:100777500-100877500
8 chr13:100777500-100877500 5275 2.865269 chr13:100718488-100785594:- 0.089686 chr13:100777500-100877500
9 chr17:40957500-41062500 5270 3.371969 chr17:41060000-41071996:- 3.974449 chr17:40957500-41062500
10 chr17:40957500-41062500 5271 4.576930 chr17:41012431-41017507:- 3.974449 chr17:40957500-41062500
11 chr2:167337500-167447500 5267 -5.452819 chr2:167318145-167341673:+ -5.452819 chr2:167337500-167447500
12 chr4:150417500-150517500 5276 6.436959 chr4:150377761-150418774:- 6.436959 chr4:150417500-150517500
13 chr6:41532500-41642500 5268 4.114012 chr6:41555481-41570508:+ 4.114012 chr6:41532500-41642500
14 chr7:6072500-6162500 5277 2.622196 chr7:6123828-6142951:+ 2.622196 chr7:6072500-6162500
Теперь сделайте простую точечную диаграмму.
ggplot() + geom_point(data=thing, aes(x=stat,y=mTADs.reordered), shape=20, cex=3.3)