Фильтр Калмана с различными временными шагами
У меня есть некоторые данные, которые представляют местоположение объекта, измеренного двумя разными датчиками. Итак, мне нужно сделать датчик слияния. Более сложная проблема заключается в том, что данные от каждого датчика поступают в основном в случайное время. Я хотел бы использовать pykalman, чтобы объединить и сгладить данные. Как pykalman может обрабатывать переменные метки времени?
Упрощенная выборка данных будет выглядеть так:
import pandas as pd
data={'time':\
['10:00:00.0','10:00:01.0','10:00:05.2','10:00:07.5','10:00:07.5','10:00:12.0','10:00:12.5']\
,'X':[10,10.1,20.2,25.0,25.1,35.1,35.0],'Y':[20,20.2,41,45,47,75.0,77.2],\
'Sensor':[1,2,1,1,2,1,2]}
df=pd.DataFrame(data,columns=['time','X','Y','Sensor'])
df.time=pd.to_datetime(df.time)
df=df.set_index('time')
И это:
df
Out[130]:
X Y Sensor
time
2017-12-01 10:00:00.000 10.0 20.0 1
2017-12-01 10:00:01.000 10.1 20.2 2
2017-12-01 10:00:05.200 20.2 41.0 1
2017-12-01 10:00:07.500 25.0 45.0 1
2017-12-01 10:00:07.500 25.1 47.0 2
2017-12-01 10:00:12.000 35.1 75.0 1
2017-12-01 10:00:12.500 35.0 77.2 2
Что касается вопроса слияния датчиков, я думаю, что я могу просто изменить форму данных, чтобы у меня были позиции X1,Y1,X2,Y2 с кучей пропущенных значений вместо просто X,Y. (Это было связано: http://stackru.com.mevn.net/questions/47386426/2-sensor-readings-fusion-yaw-pitch)
Итак, мои данные могут выглядеть так:
df['X1']=df.X[df.Sensor==1]
df['Y1']=df.Y[df.Sensor==1]
df['X2']=df.X[df.Sensor==2]
df['Y2']=df.Y[df.Sensor==2]
df
Out[132]:
X Y Sensor X1 Y1 X2 Y2
time
2017-12-01 10:00:00.000 10.0 20.0 1 10.0 20.0 NaN NaN
2017-12-01 10:00:01.000 10.1 20.2 2 NaN NaN 10.1 20.2
2017-12-01 10:00:05.200 20.2 41.0 1 20.2 41.0 NaN NaN
2017-12-01 10:00:07.500 25.0 45.0 1 25.0 45.0 25.1 47.0
2017-12-01 10:00:07.500 25.1 47.0 2 25.0 45.0 25.1 47.0
2017-12-01 10:00:12.000 35.1 75.0 1 35.1 75.0 NaN NaN
2017-12-01 10:00:12.500 35.0 77.2 2 NaN NaN 35.0 77.2
Документы для pykalman указывают, что он может обрабатывать недостающие данные, но верно ли это?
Но документы для pykalman не совсем ясно о проблеме переменного времени. Док просто говорит:
"Как Kalman Filter, так и Kalman Smoother могут использовать параметры, которые меняются со временем. Чтобы использовать это, нужно только передать массив длиной n_timesteps вдоль его первой оси:"
>>> transition_offsets = [[-1], [0], [1], [2]]
>>> kf = KalmanFilter(transition_offsets=transition_offsets, n_dim_obs=1)
Я не смог найти каких-либо примеров использования пикальмановского Smoother с переменными временными шагами. Таким образом, любые рекомендации, примеры или даже примеры, использующие мои данные выше, были бы очень полезны. Мне не нужно использовать pykalman, но это кажется полезным инструментом для сглаживания этих данных.
***** Дополнительный код добавлен ниже @Anton Я сделал версию вашего полезного кода, которая использует функцию сглаживания. Странно то, что кажется, что все наблюдения имеют одинаковый вес, и траектория проходит через все. Даже если у меня есть большое различие между значениями дисперсии датчика. Я ожидаю, что около точки 5,4,5,0 отфильтрованная траектория должна приблизиться к точке датчика 1, так как эта точка имеет меньшую дисперсию. Вместо этого траектория идет точно к каждой точке и делает большой поворот, чтобы туда добраться.
from pykalman import KalmanFilter
import numpy as np
import matplotlib.pyplot as plt
# reading data (quick and dirty)
Time=[]
RefX=[]
RefY=[]
Sensor=[]
X=[]
Y=[]
for line in open('data/dataset_01.csv'):
f1, f2, f3, f4, f5, f6 = line.split(';')
Time.append(float(f1))
RefX.append(float(f2))
RefY.append(float(f3))
Sensor.append(float(f4))
X.append(float(f5))
Y.append(float(f6))
# Sensor 1 has a higher precision (max error = 0.1 m)
# Sensor 2 has a lower precision (max error = 0.3 m)
# Variance definition through 3-Sigma rule
Sensor_1_Variance = (0.1/3)**2;
Sensor_2_Variance = (0.3/3)**2;
# Filter Configuration
# time step
dt = Time[2] - Time[1]
# transition_matrix
F = [[1, 0, dt, 0],
[0, 1, 0, dt],
[0, 0, 1, 0],
[0, 0, 0, 1]]
# observation_matrix
H = [[1, 0, 0, 0],
[0, 1, 0, 0]]
# transition_covariance
Q = [[1e-4, 0, 0, 0],
[ 0, 1e-4, 0, 0],
[ 0, 0, 1e-4, 0],
[ 0, 0, 0, 1e-4]]
# observation_covariance
R_1 = [[Sensor_1_Variance, 0],
[0, Sensor_1_Variance]]
R_2 = [[Sensor_2_Variance, 0],
[0, Sensor_2_Variance]]
# initial_state_mean
X0 = [0,
0,
0,
0]
# initial_state_covariance - assumed a bigger uncertainty in initial velocity
P0 = [[ 0, 0, 0, 0],
[ 0, 0, 0, 0],
[ 0, 0, 1, 0],
[ 0, 0, 0, 1]]
n_timesteps = len(Time)
n_dim_state = 4
filtered_state_means = np.zeros((n_timesteps, n_dim_state))
filtered_state_covariances = np.zeros((n_timesteps, n_dim_state, n_dim_state))
import numpy.ma as ma
obs_cov=np.zeros([n_timesteps,2,2])
obs=np.zeros([n_timesteps,2])
for t in range(n_timesteps):
if Sensor[t] == 0:
obs[t]=None
else:
obs[t] = [X[t], Y[t]]
if Sensor[t] == 1:
obs_cov[t] = np.asarray(R_1)
else:
obs_cov[t] = np.asarray(R_2)
ma_obs=ma.masked_invalid(obs)
ma_obs_cov=ma.masked_invalid(obs_cov)
# Kalman-Filter initialization
kf = KalmanFilter(transition_matrices = F,
observation_matrices = H,
transition_covariance = Q,
observation_covariance = ma_obs_cov, # the covariance will be adapted depending on Sensor_ID
initial_state_mean = X0,
initial_state_covariance = P0)
filtered_state_means, filtered_state_covariances=kf.smooth(ma_obs)
# extracting the Sensor update points for the plot
Sensor_1_update_index = [i for i, x in enumerate(Sensor) if x == 1]
Sensor_2_update_index = [i for i, x in enumerate(Sensor) if x == 2]
Sensor_1_update_X = [ X[i] for i in Sensor_1_update_index ]
Sensor_1_update_Y = [ Y[i] for i in Sensor_1_update_index ]
Sensor_2_update_X = [ X[i] for i in Sensor_2_update_index ]
Sensor_2_update_Y = [ Y[i] for i in Sensor_2_update_index ]
# plot of the resulted trajectory
plt.plot(RefX, RefY, "k-", label="Real Trajectory")
plt.plot(Sensor_1_update_X, Sensor_1_update_Y, "ro", label="Sensor 1")
plt.plot(Sensor_2_update_X, Sensor_2_update_Y, "bo", label="Sensor 2")
plt.plot(filtered_state_means[:, 0], filtered_state_means[:, 1], "g.", label="Filtered Trajectory", markersize=1)
plt.grid()
plt.legend(loc="upper left")
plt.show()
1 ответ
Для фильтра Калмана полезно представлять входные данные с постоянным шагом по времени. Ваши датчики отправляют данные случайным образом, так что вы можете определить наименьший значимый временной шаг для вашей системы и дискретизировать временную ось с помощью этого шага.
Например, один из ваших датчиков отправляет данные примерно каждые 0,2 секунды, а второй каждые 0,5 секунды. Таким образом, наименьший шаг по времени может составлять 0,01 секунды (здесь вам нужно найти компромисс между временем вычислений и желаемой точностью).
Ваши данные будут выглядеть так:
Time Sensor X Y
0,52 0 0 0
0,53 1 0,3417 0,2988
0,54 0 0 0
0,56 0 0 0
0,57 0 0 0
0,55 0 0 0
0,58 0 0 0
0,59 2 0,4247 0,3779
0,60 0 0 0
0,61 0 0 0
0,62 0 0 0
Теперь вам нужно вызвать функцию Pykalman filter_update в зависимости от ваших наблюдений. Если наблюдения отсутствуют, фильтр прогнозирует следующее состояние на основе предыдущего. Если есть наблюдение, оно исправляет состояние системы.
Вероятно, ваши датчики имеют разную точность. Таким образом, вы можете указать ковариацию наблюдения в зависимости от дисперсии датчика.
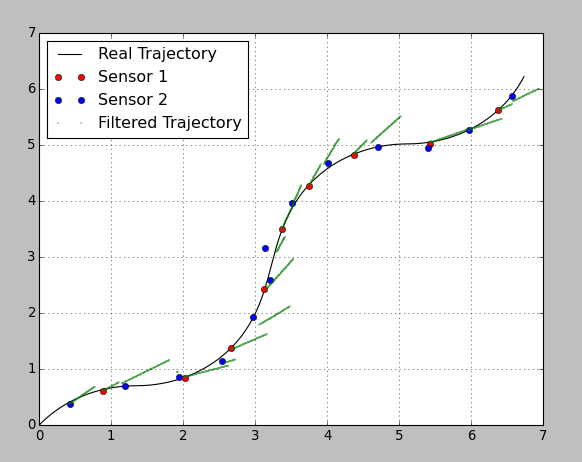
Чтобы продемонстрировать идею, я сгенерировал 2D-траекторию и случайно поместил измерения 2 датчиков с разной точностью.
Sensor1: mean update time = 1.0s; max error = 0.1m;
Sensor2: mean update time = 0.7s; max error = 0.3m;
Вот результат:
Я специально выбрал действительно плохие параметры, чтобы можно было увидеть этапы прогнозирования и коррекции. Между обновлениями датчика фильтр прогнозирует траекторию на основе постоянной скорости от предыдущего шага. Как только приходит обновление, фильтр корректирует положение в соответствии с отклонением датчика. Точность второго датчика очень плохая, поэтому он влияет на систему с меньшим весом.
Вот мой код Python:
from pykalman import KalmanFilter
import numpy as np
import matplotlib.pyplot as plt
# reading data (quick and dirty)
Time=[]
RefX=[]
RefY=[]
Sensor=[]
X=[]
Y=[]
for line in open('data/dataset_01.csv'):
f1, f2, f3, f4, f5, f6 = line.split(';')
Time.append(float(f1))
RefX.append(float(f2))
RefY.append(float(f3))
Sensor.append(float(f4))
X.append(float(f5))
Y.append(float(f6))
# Sensor 1 has a higher precision (max error = 0.1 m)
# Sensor 2 has a lower precision (max error = 0.3 m)
# Variance definition through 3-Sigma rule
Sensor_1_Variance = (0.1/3)**2;
Sensor_2_Variance = (0.3/3)**2;
# Filter Configuration
# time step
dt = Time[2] - Time[1]
# transition_matrix
F = [[1, 0, dt, 0],
[0, 1, 0, dt],
[0, 0, 1, 0],
[0, 0, 0, 1]]
# observation_matrix
H = [[1, 0, 0, 0],
[0, 1, 0, 0]]
# transition_covariance
Q = [[1e-4, 0, 0, 0],
[ 0, 1e-4, 0, 0],
[ 0, 0, 1e-4, 0],
[ 0, 0, 0, 1e-4]]
# observation_covariance
R_1 = [[Sensor_1_Variance, 0],
[0, Sensor_1_Variance]]
R_2 = [[Sensor_2_Variance, 0],
[0, Sensor_2_Variance]]
# initial_state_mean
X0 = [0,
0,
0,
0]
# initial_state_covariance - assumed a bigger uncertainty in initial velocity
P0 = [[ 0, 0, 0, 0],
[ 0, 0, 0, 0],
[ 0, 0, 1, 0],
[ 0, 0, 0, 1]]
n_timesteps = len(Time)
n_dim_state = 4
filtered_state_means = np.zeros((n_timesteps, n_dim_state))
filtered_state_covariances = np.zeros((n_timesteps, n_dim_state, n_dim_state))
# Kalman-Filter initialization
kf = KalmanFilter(transition_matrices = F,
observation_matrices = H,
transition_covariance = Q,
observation_covariance = R_1, # the covariance will be adapted depending on Sensor_ID
initial_state_mean = X0,
initial_state_covariance = P0)
# iterative estimation for each new measurement
for t in range(n_timesteps):
if t == 0:
filtered_state_means[t] = X0
filtered_state_covariances[t] = P0
else:
# the observation and its covariance have to be switched depending on Sensor_Id
# Sensor_ID == 0: no observation
# Sensor_ID == 1: Sensor 1
# Sensor_ID == 2: Sensor 2
if Sensor[t] == 0:
obs = None
obs_cov = None
else:
obs = [X[t], Y[t]]
if Sensor[t] == 1:
obs_cov = np.asarray(R_1)
else:
obs_cov = np.asarray(R_2)
filtered_state_means[t], filtered_state_covariances[t] = (
kf.filter_update(
filtered_state_means[t-1],
filtered_state_covariances[t-1],
observation = obs,
observation_covariance = obs_cov)
)
# extracting the Sensor update points for the plot
Sensor_1_update_index = [i for i, x in enumerate(Sensor) if x == 1]
Sensor_2_update_index = [i for i, x in enumerate(Sensor) if x == 2]
Sensor_1_update_X = [ X[i] for i in Sensor_1_update_index ]
Sensor_1_update_Y = [ Y[i] for i in Sensor_1_update_index ]
Sensor_2_update_X = [ X[i] for i in Sensor_2_update_index ]
Sensor_2_update_Y = [ Y[i] for i in Sensor_2_update_index ]
# plot of the resulted trajectory
plt.plot(RefX, RefY, "k-", label="Real Trajectory")
plt.plot(Sensor_1_update_X, Sensor_1_update_Y, "ro", label="Sensor 1")
plt.plot(Sensor_2_update_X, Sensor_2_update_Y, "bo", label="Sensor 2")
plt.plot(filtered_state_means[:, 0], filtered_state_means[:, 1], "g.", label="Filtered Trajectory", markersize=1)
plt.grid()
plt.legend(loc="upper left")
plt.show()
Я поместил CSV-файл здесь, чтобы вы могли выполнить код.
Я надеюсь, что смогу помочь тебе.
ОБНОВИТЬ
Некоторая информация к вашему предложению о переменной матрице перехода. Я бы сказал, что это зависит от доступности ваших датчиков и от требований к результату оценки.
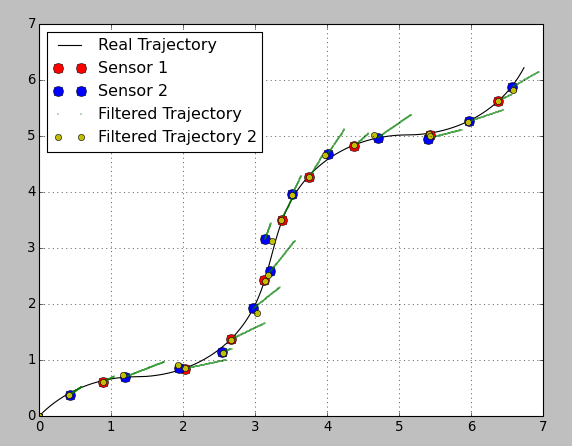
Здесь я построил одну и ту же оценку как с постоянной, так и с переменной матрицей перехода (я изменил ковариационную матрицу перехода, в противном случае оценка была слишком плохой из-за высокой "жесткости" фильтра):
Как видите, примерное положение желтых маркеров довольно хорошее. НО! у вас нет информации между показаниями датчика. Используя переменную матрицу перехода, вы избегаете шага прогнозирования между показаниями и не знаете, что происходит с системой. Это может быть достаточно хорошо, если ваши показания идут с высокой скоростью, но в противном случае это может быть недостатком.
Вот обновленный код:
from pykalman import KalmanFilter
import numpy as np
import matplotlib.pyplot as plt
# reading data (quick and dirty)
Time=[]
RefX=[]
RefY=[]
Sensor=[]
X=[]
Y=[]
for line in open('data/dataset_01.csv'):
f1, f2, f3, f4, f5, f6 = line.split(';')
Time.append(float(f1))
RefX.append(float(f2))
RefY.append(float(f3))
Sensor.append(float(f4))
X.append(float(f5))
Y.append(float(f6))
# Sensor 1 has a higher precision (max error = 0.1 m)
# Sensor 2 has a lower precision (max error = 0.3 m)
# Variance definition through 3-Sigma rule
Sensor_1_Variance = (0.1/3)**2;
Sensor_2_Variance = (0.3/3)**2;
# Filter Configuration
# time step
dt = Time[2] - Time[1]
# transition_matrix
F = [[1, 0, dt, 0],
[0, 1, 0, dt],
[0, 0, 1, 0],
[0, 0, 0, 1]]
# observation_matrix
H = [[1, 0, 0, 0],
[0, 1, 0, 0]]
# transition_covariance
Q = [[1e-2, 0, 0, 0],
[ 0, 1e-2, 0, 0],
[ 0, 0, 1e-2, 0],
[ 0, 0, 0, 1e-2]]
# observation_covariance
R_1 = [[Sensor_1_Variance, 0],
[0, Sensor_1_Variance]]
R_2 = [[Sensor_2_Variance, 0],
[0, Sensor_2_Variance]]
# initial_state_mean
X0 = [0,
0,
0,
0]
# initial_state_covariance - assumed a bigger uncertainty in initial velocity
P0 = [[ 0, 0, 0, 0],
[ 0, 0, 0, 0],
[ 0, 0, 1, 0],
[ 0, 0, 0, 1]]
n_timesteps = len(Time)
n_dim_state = 4
filtered_state_means = np.zeros((n_timesteps, n_dim_state))
filtered_state_covariances = np.zeros((n_timesteps, n_dim_state, n_dim_state))
filtered_state_means2 = np.zeros((n_timesteps, n_dim_state))
filtered_state_covariances2 = np.zeros((n_timesteps, n_dim_state, n_dim_state))
# Kalman-Filter initialization
kf = KalmanFilter(transition_matrices = F,
observation_matrices = H,
transition_covariance = Q,
observation_covariance = R_1, # the covariance will be adapted depending on Sensor_ID
initial_state_mean = X0,
initial_state_covariance = P0)
# Kalman-Filter initialization (Different F Matrices depending on DT)
kf2 = KalmanFilter(transition_matrices = F,
observation_matrices = H,
transition_covariance = Q,
observation_covariance = R_1, # the covariance will be adapted depending on Sensor_ID
initial_state_mean = X0,
initial_state_covariance = P0)
# iterative estimation for each new measurement
for t in range(n_timesteps):
if t == 0:
filtered_state_means[t] = X0
filtered_state_covariances[t] = P0
# For second filter
filtered_state_means2[t] = X0
filtered_state_covariances2[t] = P0
timestamp = Time[t]
old_t = t
else:
# the observation and its covariance have to be switched depending on Sensor_Id
# Sensor_ID == 0: no observation
# Sensor_ID == 1: Sensor 1
# Sensor_ID == 2: Sensor 2
if Sensor[t] == 0:
obs = None
obs_cov = None
else:
obs = [X[t], Y[t]]
if Sensor[t] == 1:
obs_cov = np.asarray(R_1)
else:
obs_cov = np.asarray(R_2)
filtered_state_means[t], filtered_state_covariances[t] = (
kf.filter_update(
filtered_state_means[t-1],
filtered_state_covariances[t-1],
observation = obs,
observation_covariance = obs_cov)
)
#For the second filter
if Sensor[t] != 0:
obs2 = [X[t], Y[t]]
if Sensor[t] == 1:
obs_cov2 = np.asarray(R_1)
else:
obs_cov2 = np.asarray(R_2)
dt2 = Time[t] - timestamp
timestamp = Time[t]
# transition_matrix
F2 = [[1, 0, dt2, 0],
[0, 1, 0, dt2],
[0, 0, 1, 0],
[0, 0, 0, 1]]
filtered_state_means2[t], filtered_state_covariances2[t] = (
kf2.filter_update(
filtered_state_means2[old_t],
filtered_state_covariances2[old_t],
observation = obs2,
observation_covariance = obs_cov2,
transition_matrix = np.asarray(F2))
)
old_t = t
# extracting the Sensor update points for the plot
Sensor_1_update_index = [i for i, x in enumerate(Sensor) if x == 1]
Sensor_2_update_index = [i for i, x in enumerate(Sensor) if x == 2]
Sensor_1_update_X = [ X[i] for i in Sensor_1_update_index ]
Sensor_1_update_Y = [ Y[i] for i in Sensor_1_update_index ]
Sensor_2_update_X = [ X[i] for i in Sensor_2_update_index ]
Sensor_2_update_Y = [ Y[i] for i in Sensor_2_update_index ]
# plot of the resulted trajectory
plt.plot(RefX, RefY, "k-", label="Real Trajectory")
plt.plot(Sensor_1_update_X, Sensor_1_update_Y, "ro", label="Sensor 1", markersize=9)
plt.plot(Sensor_2_update_X, Sensor_2_update_Y, "bo", label="Sensor 2", markersize=9)
plt.plot(filtered_state_means[:, 0], filtered_state_means[:, 1], "g.", label="Filtered Trajectory", markersize=1)
plt.plot(filtered_state_means2[:, 0], filtered_state_means2[:, 1], "yo", label="Filtered Trajectory 2", markersize=6)
plt.grid()
plt.legend(loc="upper left")
plt.show()
Еще один важный момент, который я не реализовал в этом коде: при использовании переменной матрицы перехода необходимо также изменять ковариационную матрицу перехода (в зависимости от текущего значения dt).
Это интересная тема. Дайте мне знать, какая оценка лучше всего подходит для вашей задачи.