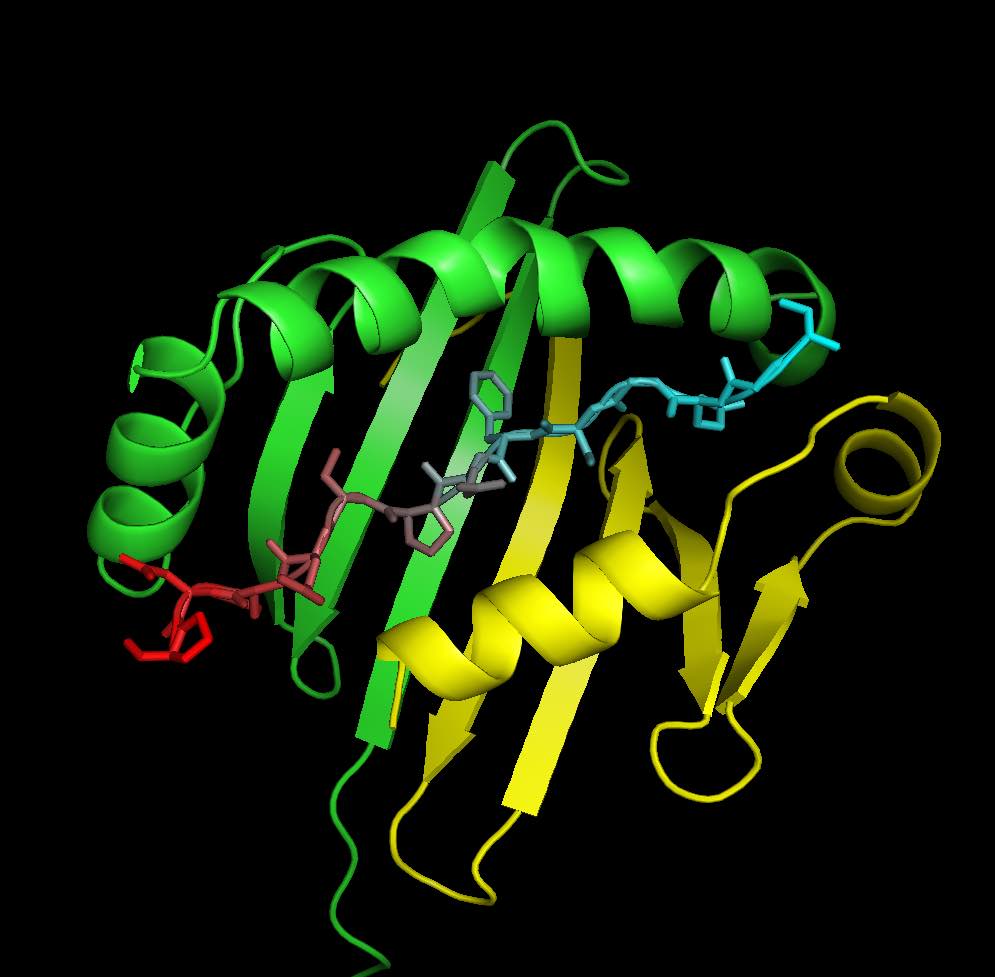
Как показать расстояние между остатками в Пимоле
С этим файлом PDB и следующим кодом PyMOL:
cd /Users/foo/Desktop/
reinitialize
load pdp_4gg6CD1_I.pdb
as cartoon
select chainI, chain I
select chainC, chain C
select chainD, chain D
show sticks, chainI
spectrum count, cyan_red, chainI
color yellow, chain C
Я могу сделать это изображение:
То, что я хочу сделать, это показать расстояние между выбранными остатками в chainC (желтый) с помощью chainI (stick).
Выбранные остатки цепи C, которые я хочу, это:
[9, 23, 25, 44, 53, 54, 55, 59, 62, 63, 66]
Y Y H W R R F F T N V
Как я могу это сделать?
1 ответ
Решение
- Давайте использовать
cmd.iterateчтобы получить все атомы в выборке (цепочка C и цепочка I) и записать координаты и имя атома в словари для дальнейшего использования. - Затем мы можем рассчитать все расстояния между всеми атомами и записать ближайшие остатки и имена атомов в два списка (
min_cа такжеmin_i) - На последнем шаге нам нужно только нарисовать объекты расстояния между ближайшими атомами, и все готово:)
from math import sqrt
def closestAtoms(list1=[9, 23, 25, 44, 53, 54, 55, 59, 62, 63, 66], list2=[5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17]):
atoms = {}
atoms2 = {}
for r in list1:
command = "chain C and resi %s" % (r)
coordinates = {'atoms': []}
cmd.iterate_state(-1, command, 'atoms.append([x, y, z, name])', space=coordinates)
atoms[r] = coordinates['atoms']
for i in list2:
command = "chain I and resi %s" % (i)
coordinates = {'atoms': []}
cmd.iterate_state(-1, command, 'atoms.append([x, y, z, name])', space=coordinates)
atoms2[i] = coordinates['atoms']
for i in list2:
min_dist = 10**3
for c in list1:
for cc in atoms[c]:
for ii in atoms2[i]:
dist = sqrt((cc[0] - ii[0])**2 + (cc[1] - ii[1])**2 + (cc[2] - ii[2])**2)
if dist < min_dist:
min_dist = dist
min_c = [c, cc[3]]
min_i = [i, ii[3]]
cmd.distance('dist_%s_%s' % (min_c[0], min_i[0]), 'chain C and resi %s and name %s' % (min_c[0], min_c[1]), 'chain I and resi %s and name %s' % (min_i[0], min_i[1]))
cmd.extend("closestAtoms", closestAtoms)