mgcv: как извлечь узлы, базис, коэффициенты и прогнозы для P-сплайнов в адаптивном сглаживании?
Я использую пакет mgcv в R, чтобы подогнать некоторые полиномиальные сплайны к некоторым данным через:
x.gam <- gam(cts ~ s(time, bs = "ad"), data = x.dd,
family = poisson(link = "log"))
Я пытаюсь извлечь функциональную форму подгонки. x.gam это gamObjectи я читал документацию, но не нашел достаточно информации для того, чтобы вручную восстановить подобранную функцию.
x.gam$smoothсодержит информацию о том, были ли размещены узлы;x.gam$coefficientsдает коэффициенты сплайна, но я не знаю, в каком порядке используются полиномиальные сплайны, и поиск в коде ничего не выявил.
Есть ли аккуратный способ извлечения используемых узлов, коэффициентов и базиса, чтобы можно было вручную восстановить подгонку?
1 ответ
У меня нет ваших данных, поэтому я беру следующий пример из ?adaptive.smooth чтобы показать вам, где вы можете найти информацию, которую вы хотите. Обратите внимание, что хотя этот пример предназначен для данных Гаусса, а не для данных Пуассона, отличается только функция связи; все остальные просто стандартные.
x <- 1:1000/1000 # data between [0, 1]
mu <- exp(-400*(x-.6)^2)+5*exp(-500*(x-.75)^2)/3+2*exp(-500*(x-.9)^2)
y <- mu+0.5*rnorm(1000)
b <- gam(y~s(x,bs="ad",k=40,m=5))
Теперь вся информация о плавном построении хранится в b$smoothвынимаем это:
smooth <- b$smooth[[1]] ## extract smooth object for first smooth term
узлы:
smooth$knots дает вам местоположение узлов.
> smooth$knots
[1] -0.081161 -0.054107 -0.027053 0.000001 0.027055 0.054109 0.081163
[8] 0.108217 0.135271 0.162325 0.189379 0.216433 0.243487 0.270541
[15] 0.297595 0.324649 0.351703 0.378757 0.405811 0.432865 0.459919
[22] 0.486973 0.514027 0.541081 0.568135 0.595189 0.622243 0.649297
[29] 0.676351 0.703405 0.730459 0.757513 0.784567 0.811621 0.838675
[36] 0.865729 0.892783 0.919837 0.946891 0.973945 1.000999 1.028053
[43] 1.055107 1.082161
Обратите внимание, три внешних узла расположены за каждой стороной [0, 1] построить сплайн-основу.
базовый класс
attr(smooth, "class") говорит вам тип сплайна. Как вы можете прочитать из ?adaptive.smooth, за bs = ad, mgcv используйте P-сплайны, поэтому вы получите "pspline.smooth".
mgcv используйте pspline 2-го порядка, вы можете проверить это, проверив матрицу разностей smooth$D, Ниже приведен снимок:
> smooth$D[1:6,1:6]
[,1] [,2] [,3] [,4] [,5] [,6]
[1,] 1 -2 1 0 0 0
[2,] 0 1 -2 1 0 0
[3,] 0 0 1 -2 1 0
[4,] 0 0 0 1 -2 1
[5,] 0 0 0 0 1 -2
[6,] 0 0 0 0 0 1
коэффициенты
Вы уже знали, что b$coefficients содержать коэффициенты модели:
beta <- b$coefficients
Обратите внимание, что это именованный вектор:
> beta
(Intercept) s(x).1 s(x).2 s(x).3 s(x).4 s(x).5
0.37792619 -0.33500685 -0.30943814 -0.30908847 -0.31141148 -0.31373448
s(x).6 s(x).7 s(x).8 s(x).9 s(x).10 s(x).11
-0.31605749 -0.31838050 -0.32070350 -0.32302651 -0.32534952 -0.32767252
s(x).12 s(x).13 s(x).14 s(x).15 s(x).16 s(x).17
-0.32999553 -0.33231853 -0.33464154 -0.33696455 -0.33928755 -0.34161055
s(x).18 s(x).19 s(x).20 s(x).21 s(x).22 s(x).23
-0.34393354 -0.34625650 -0.34857906 -0.05057041 0.48319491 0.77251118
s(x).24 s(x).25 s(x).26 s(x).27 s(x).28 s(x).29
0.49825345 0.09540020 -0.18950763 0.16117012 1.10141701 1.31089436
s(x).30 s(x).31 s(x).32 s(x).33 s(x).34 s(x).35
0.62742937 -0.23435309 -0.19127140 0.79615752 1.85600016 1.55794576
s(x).36 s(x).37 s(x).38 s(x).39
0.40890236 -0.20731309 -0.47246357 -0.44855437
матрица базиса / матрица модели / матрица линейного предиктора (lpmatrix)
Вы можете получить матрицу модели из:
mat <- predict.gam(b, type = "lpmatrix")
Это n-by-p матрица, где n количество наблюдений, и p это число коэффициентов. Эта матрица имеет имя столбца:
> head(mat[,1:5])
(Intercept) s(x).1 s(x).2 s(x).3 s(x).4
1 1 0.6465774 0.1490613 -0.03843899 -0.03844738
2 1 0.6437580 0.1715691 -0.03612433 -0.03619157
3 1 0.6384074 0.1949416 -0.03391686 -0.03414389
4 1 0.6306815 0.2190356 -0.03175713 -0.03229541
5 1 0.6207361 0.2437083 -0.02958570 -0.03063719
6 1 0.6087272 0.2688168 -0.02734314 -0.02916029
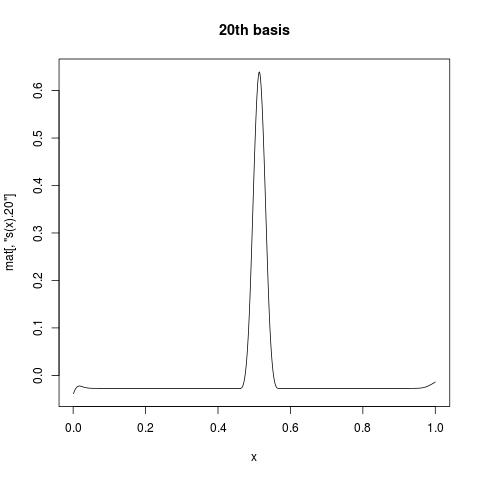
Первый столбец - все 1, дающий перехват. В то время как s(x).1 предлагает первую базисную функцию для s(x), Если вы хотите посмотреть, как выглядит отдельная базовая функция, вы можете построить столбец mat против вашей переменной. Например:
plot(x, mat[, "s(x).20"], type = "l", main = "20th basis")
линейный предиктор
Если вы хотите вручную построить подгонку, вы можете сделать:
pred.linear <- mat %*% beta
Обратите внимание, что это именно то, что вы можете получить от b$linear.predictors или же
predict.gam(b, type = "link")
ответ / установленные значения
Для негауссовых данных, если вы хотите получить переменную ответа, вы можете применить функцию обратной связи к линейному предиктору, чтобы отобразить обратно в исходный масштаб.
Семейная информация хранится в gamObject$family, а также gamObject$family$linkinv является функцией обратной ссылки. Приведенный выше пример наверняка даст вам идентификационную ссылку, но для вашего подобранного объекта x.gam, ты можешь сделать:
x.gam$family$linkinv(x.gam$linear.predictors)
Обратите внимание, это то же самое x.gam$fitted, или же
predict.gam(x.gam, type = "response").
Другие ссылки
Я только что понял, что раньше было много подобных вопросов.
- Этот ответ Гэвина Симпсона великолепен для
predict.gam( , type = 'lpmatrix'), - Этот ответ о
predict.gam(, type = 'terms'),
Но в любом случае, лучшая ссылка всегда ?predict.gam, который включает в себя обширные примеры.