Я получаю не значимые различия в значениях ANOVA, но есть некоторые действительно важные данные. Что не так с моими сценариями?
Я получаю не значимые различия в значениях ANOVA, но есть некоторые действительно важные данные. Что не так с моими сценариями?
df <- structure(list(value = c(1.693086732, 0.25167691, 1.100272527, 1.60428654, 0.908237338, 1.449864567, 1.06604818, 0.596785144, 0.652925021, 0.453697295, 0.544252785, 1.464221767, 1.043720641, 0.735035158, 0.938875327, 0.712832947, 1.701854524, 1.021094251, 0.564349482, 2.326316679, 1.10170484, 1.075217638, 1.397442796, 0.501086703, 0.675502908, 0.846651623, 1.578086856, 1.857360967, 1.194232629, 1.875837087, 1.106882408, 1.112407609, 1.30479321, 0.637491754, 1.281566883, 1.103115742, 1.895286629, 1.623933836, 0.941989812, 1.30636425, 0.69977606, 1.937055334, 0.666069131, 0.829396619, 0.892844633, 0.573255443, 1.27370148, 0.531593222, 2.782899244, 0.972928201, 0.729463812, 1.121965821, 2.55117084, 0.999302442, 1.138902544, 1.656807007, 0.545349299, 0.550315908, 2.346074577, 0.637551271), gene = c("WT", "WT", "WT", "WT", "WT", "WT", "WT", "WT", "WT", "WT", "WT", "WT", "aox2", "aox2", "aox2", "aox2", "aox2", "aox2", "aox2", "aox2", "aox2", "aox2", "aox2", "aox2", "aox5", "aox5", "aox5", "aox5", "aox5", "aox5", "aox5", "aox5", "aox5", "aox5", "aox5", "aox5", "aox7", "aox7", "aox7", "aox7", "aox7", "aox7", "aox7", "aox7", "aox7", "aox7", "aox7", "aox7", "aox9", "aox9", "aox9", "aox9", "aox9", "aox9", "aox9", "aox9", "aox9", "aox9", "aox9", "aox9"), time = c("0t", "0t", "0t", "1t", "1t", "1t", "3t", "3t", "3t", "5t", "5t", "5t", "0t", "0t", "0t", "1t", "1t", "1t", "3t", "3t", "3t", "5t", "5t", "5t", "0t", "0t", "0t", "1t", "1t", "1t", "3t", "3t", "3t", "5t", "5t", "5t", "0t", "0t", "0t", "1t", "1t", "1t", "3t", "3t", "3t", "5t", "5t", "5t", "0t", "0t", "0t", "1t", "1t", "1t", "3t", "3t", "3t", "5t", "5t", "5t")), row.names = c(NA, -60L), class = c("data.table", "data.frame"))
Скрипты, которые я использовал:
library(emmeans)
fit <- aov(value ~ gene*time, df)
summary(fit)
em <- emmeans(fit, ~ gene | time)
pairs(em)
pairs(em, adjust=NULL)
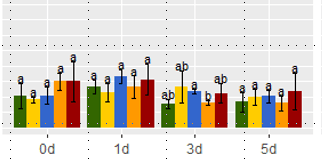
Когда я строю данные в виде гистограммы с помощью Std Err, я вижу, что в некоторых примерах это действительно важно, особенно на 3-й день. Я уже проверил это в парном t-тесте, и эти образцы значительно отличаются при p 0,05.
Это с парным тестом, даже Excel дает результаты в момент времени 3.
Не могли бы вы попытаться исправить мой ANOVA и ls средства.
1 ответ
Не уверен, почему вы ожидаете, что парное сравнение Tukey HSD будет значительным; ни один из главных эффектов в Anova не является, и нет ничего очевидного в сюжете.
Для полноты картины вы можете иметь значимость HSD для Tukey, даже если основной эффект отсутствует, но это редко, и важно заранее решить, какой тест больше подходит для вашего вопроса, а не выбирать тот, который дает "лучшие" результаты.
summary(fit)
## Df Sum Sq Mean Sq F value Pr(>F)
## gene 4 0.873 0.2184 0.625 0.648
## time 3 1.670 0.5568 1.593 0.206
## gene:time 12 1.577 0.1314 0.376 0.965
## Residuals 40 13.984 0.3496
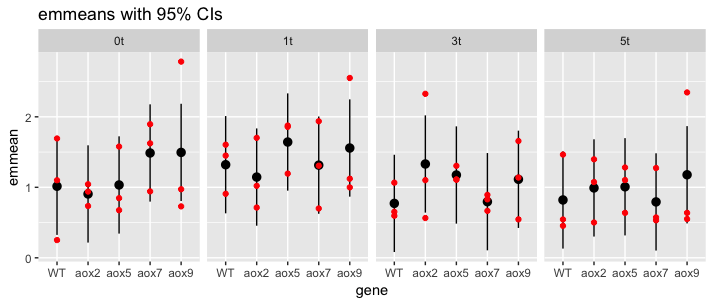
library(ggplot2)
sem <- summary(em)
sem$gene <- relevel(factor(sem$gene), 'WT')
ggplot(sem) + aes(gene, emmean, ymin=lower.CL, ymax=upper.CL) + geom_pointrange() +
facet_wrap(~time, nrow=1) + ggtitle("emmeans with 95% CIs")
График показывает необработанные данные красным цветом (это будет важно).
Кстати, ни один из парных t-тестов также не имеет значения на уровне.05, даже без коррекции для множественных сравнений. Как и в вашем коде, я тестирую попарно для генов каждый раз.
library(broom)
library(tidyr)
library(dplyr)
options(digits=2)
lapply(split(df, df$time), function(x) {
data.frame(time=x$time[1], tidy(pairwise.t.test(x$value, x$gene, p.adjust.method="none")))
}) %>% bind_rows() %>% spread(time, p.value)
## group1 group2 0t 1t 3t 5t
## 1 aox5 aox2 0.82 0.32 0.71 0.97
## 2 aox7 aox2 0.32 0.73 0.21 0.70
## 3 aox7 aox5 0.43 0.50 0.37 0.67
## 4 aox9 aox2 0.31 0.40 0.60 0.71
## 5 aox9 aox5 0.42 0.86 0.88 0.74
## 6 aox9 aox7 0.99 0.62 0.45 0.45
## 7 WT aox2 0.85 0.72 0.20 0.74
## 8 WT aox5 0.97 0.51 0.34 0.71
## 9 WT aox7 0.41 0.99 0.95 0.96
## 10 WT aox9 0.40 0.63 0.42 0.49
Однако, если вы запустите это без объединения дисперсии (или снова, без исправления для множественных сравнений), вы получите ожидаемый результат со значением p для aox5 и aox7 при 3t = 0,016.
Тем не менее, это НЕПРАВИЛЬНАЯ вещь, чтобы сделать по этим двум причинам; Во-первых, вам действительно следует исправить несколько сравнений, а во-вторых, у вас недостаточно данных для достаточно точной оценки дисперсии, поэтому объединение важно. Вы можете видеть это в необработанных данных; у этих двоих более достоверные данные, чем у других, но это почти наверняка связано со случайным шансом.
## group1 group2 0t 1t 3t 5t
## 1 aox5 aox2 0.70 0.25 0.794 0.96
## 2 aox7 aox2 0.17 0.73 0.413 0.61
## 3 aox7 aox5 0.32 0.49 0.016 0.53
## 4 aox9 aox2 0.46 0.52 0.744 0.79
## 5 aox9 aox5 0.56 0.89 0.868 0.80
## 6 aox9 aox7 0.99 0.71 0.428 0.59
## 7 WT aox2 0.82 0.65 0.398 0.70
## 8 WT aox5 0.97 0.36 0.096 0.65
## 9 WT aox7 0.41 0.99 0.892 0.95
## 10 WT aox9 0.57 0.69 0.409 0.63
Таким образом, краткий ответ в основном состоит в том, что ANOVA с парными тестами не совпадает с вашими парными t-тестами, потому что ANOVA объединяет SD, а ваши t-тесты - нет, но больший ответ заключается в том, что вы должны и объединить, и исправить для несколько сравнений, поэтому код, который вы имели в начале, а именно:
pairs(em)
это правильная вещь.