Сохранение начальных нулей в файле сопоставления.txt с разделителями табуляции в Phyloseq
Я делаю анализ данных микробиома в R, используя пакет phyloseq. Первым шагом этого анализа является импорт двух файлов, один из которых представляет собой файл.BIOM (таксономическая информация), а другой - файл метаданных (разделенный табуляцией.txt).
Оба файла содержат 147 сэмплов, которые перечислены в первом столбце (#SampleID), например, 001, 002, 003…….010, 011, …….147
Я могу успешно импортировать файл BIOM с помощью следующей команды -
biom_file = "otu_table.biom"
biomot = import_biom(biom_file, parseFunction = parse_taxonomy_greengenes)
Но когда я пытаюсь импортировать файл метаданных.txt с помощью этой формулы,
map_file = "map2.txt"
bmsd = import_qiime_sample_data(map_file)
Он удаляет все начальные нули из имен образцов в столбце #SampleID. Поэтому я не могу объединить эти два файла на последующих этапах анализа. Может кто-нибудь, пожалуйста, помогите мне, как я могу сохранить эти первые ноль в имени образца в столбце #SampleID.
Спасибо за помощь.
Структура данных во входном файле.txt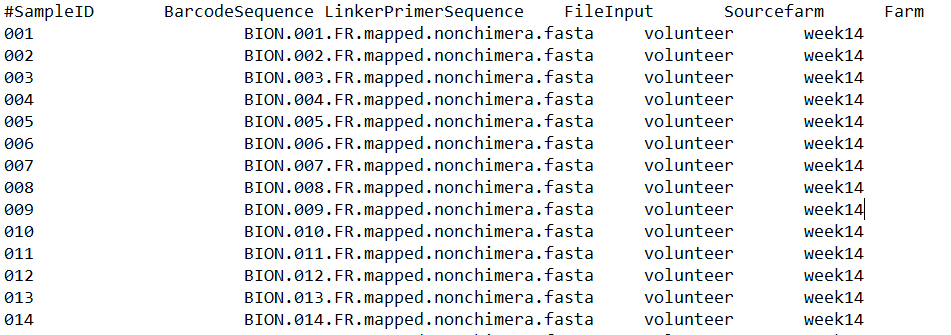
1 ответ
import_qiime_sample_dat определяется как:
import_qiime_sample_dat <- function (mapfilename)
{
QiimeMap <- read.table(file = mapfilename, header = TRUE,
sep = "\t", comment.char = "")
rownames(QiimeMap) <- as.character(QiimeMap[, 1])
return(sample_data(QiimeMap))
}
и, как вы можете видеть, использует read.table, который автоматически преобразует столбцы, содержащие числа, в целые / числовые значения, тем самым удаляя начальные нули.
Чтобы избежать этого, вы можете указать нужные классы столбцов, которые будут использоваться в txt -> data.frame конверсия, но к сожалению import_qiime_sample_dat не позволяет этого.
Следовательно, вы должны импортировать файл вручную:
tmpDF <- read.table(file = mapfilename, header = TRUE, sep = "\t",
comment.char = "", colClasses = 'character')
row.names(tmpDF) <- as.character(tmpDF[[1]])
bmsd <- sample_data(tmpDF)