Интерполяция между двумя изображениями с разным размером пикселей
Для моего приложения я хочу интерполировать между двумя изображениями (от CT до PET). Поэтому я сопоставляю их между собой так:
[X,Y,Z] = ndgrid(linspace(1,size(imagedata_ct,1),size_pet(1)),...
linspace(1,size(imagedata_ct,2),size_pet(2)),...
linspace(1,size(imagedata_ct,3),size_pet(3)));
new_imageData_CT=interp3(imagedata_ct,X,Y,Z,'nearest',-1024);
Размер моего нового изображения new_imageData_CT похож на изображение ПЭТ. Проблема в том, что данные моего нового изображения неправильно масштабированы. Так что это сжато. Я думаю, что причина этого в том, что размер пикселей между двумя изображениями различен и не связан с интерполяцией. Так, например:
- Размер изображения CT: 512x512x1027
- Размер вокселя КТ [мм]: 1,5x1,5x0,6
- Размер изображения ПЭТ: 192x126x128
- Размер вокселя ПЭТ [мм]: 2,6x2,6x3,12
Так, как я мог заботиться о размере вокселя относительно интерполяции?
3 ответа
Необходимо выполнить сопоставление в системе координат пациента, но нужно учитывать не только разрешение и размер вокселя. Вам нужно синхронизировать положения (и, возможно, ориентации, но это маловероятно) двух томов.
Вы можете найти эту ветку полезной, чтобы узнать, какие теги DICOM описывают объем и как рассчитать матрицы преобразования, чтобы использовать их для преобразования между пациентом (x, y, z в миллиметрах) и объемом (x, y, z в столбце, строке, номер среза).
Вы должны убедиться, что позиции объема сопоставимы, так как позиции срезов в CT и PET не обязательно относятся к одному и тому же источнику. Самый простой способ сделать это - сравнить UID Frame Of Reference атрибута DICOM (0020,0052) срезов CT и PET. Для всех срезов, которые используют один и тот же UID Frame Of Reference, позиция среза в заголовке DICOM относится к одному и тому же источнику. Если наборы данных не содержат этого тега, это будет намного сложнее, если вы просто не введете его в качестве предположения. Существуют методы, позволяющие выводить совпадающие срезы двух разных томов из содержимого данных пикселей, называемых "регистрацией", но это отдельная наука. Смотрите ссылку от Hugues Fontenelle.
Кстати: в вашем примере вы не найдете подходящего вокселя в обоих томах для каждой позиции, поскольку тома имеют разный размер. Например, для направления х:
КТ: 512 * 1,5 = 768 мм
ПЭТ: 192 * 2,6 = 499 мм
Я дам кому-то еще ответить на вопрос, но я думаю, что вы спрашиваете не тот. Конечно, мне не хватает контекста, но на первый взгляд Matlab не подходит для работы.
- Взгляните на ITK (библиотека C++ с оболочками Python) и статью " Мультимодальная регистрация 3D-изображений".
- Попробуйте 3DSlicer (он имеет графический интерфейс для предыдущего инструмента)
- Попробуйте FreeSurfer (похоже, ориентирован на сканирование мозга)
После того, как вы сделали этот шаг регистрации, вы можете экспортировать полученные изображения (теперь с одинаковым размером и интервалом) и продолжить интерполяцию в Matlab, если хотите (или с теми же инструментами).
В слайсере есть набор инструментов под названием PETCTFUSION, который выравнивает сканирование ПЭТ по КТ-изображению. Вы можете установить его в новой версии Slicer. На панели "Дисплей" модуля, показанной ниже, предусмотрены параметры для выбора схемы окраски для набора данных PET: серый цвет обеспечит цветопередачу от белого к черному, а черный - наивысшие значения счетчика. Тепло обеспечит теплую цветовую шкалу с темно-красным наименьшим, а белый - наивысшими значениями. Spectrum обеспечит теплую цветовую гамму, которая становится более холодной (темно-синей) на конце низкого счета и белым на самом высоком. Эта панель также предоставляет средства для настройки окна и уровня громкости ПЭТ и КТ.
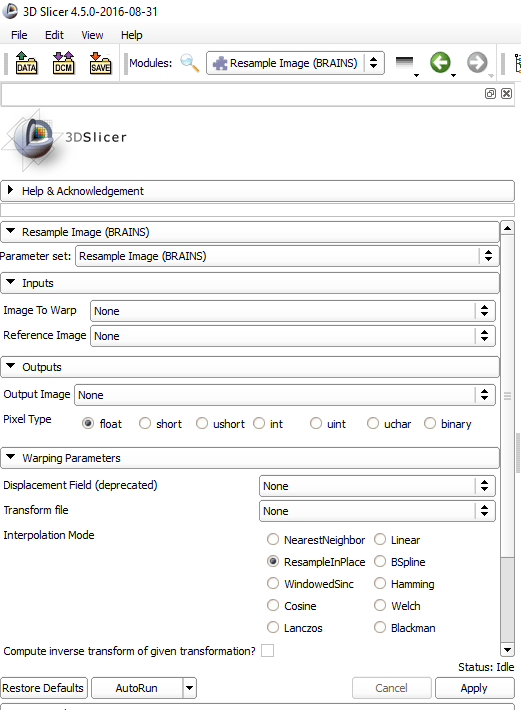
Я обычно использую инструмент resampleinplace после регистрации. Вы можете найти его в пакете: зарегистрируйтесь, а затем повторите образец изображения.
Если вы хотите узнать больше о PETCTFUSION, есть ссылка ниже: https://www.slicer.org/wiki/Modules:PETCTFusion-Documentation-3.6
Поскольку slicer совместим с python, вы также можете использовать его для запуска собственного кода.
И дайте мне знать, если у вас возникнут проблемы