Наложение двух разных форматов qplots в ggplot2
У меня есть два графика рассеяния, основанных на разных, но связанных данных, созданных с помощью qplot() из ggplot2. (Учусь ggplot не было приоритетом, потому что qplot до сих пор было достаточно для моих нужд). То, что я хочу сделать, это наложить / наложить две диаграммы так, чтобы данные x,y для каждой были нанесены в одном и том же пространстве графика. Сложность в том, что я хочу, чтобы каждый сюжет сохранил свое форматирование / эстетику.
Эти данные представляют собой оценки по строкам и столбцам из анализа соответствия - corresp() от MASS - поэтому количество строк данных (то есть выборок или таксонов) отличается между двумя наборами данных. Я могу легко составить два набора партитур. Либо путем объединения двух наборов данных, либо, что еще проще, просто с помощью biplot() функция.
Однако я использовал qplot, чтобы графики выглядели именно так, как мне нужно; с образцами, нанесенными в виде цветных символов и таксонов в качестве меток:
PlotSample <- qplot(DataCorresp$rscore[,1], DataCorresp$rscore[,2],
colour=factor(DataAll$ColourCode)) +
scale_colour_manual(values = c("black","darkgoldenrod2",
"deepskyblue2","deeppink2"))
а также
PlotTaxa <- qplot(DataCorresp$cscore[,1], DataCorresp$cscore[,2],
label=colnames(DataCorresp), size=10, geom=“text”)
Может кто-нибудь предложить способ, по которому либо
- два участка (
PlotSampleа такжеPlotTaxa) могут накладываться друг на друга, - два набора данных (
DataCorresp$rscoreа такжеDataCorresp$cscore) могут быть построены вместе, но отформатированы по-разному, или - другая функция (например,
biplot()) что может быть использовано для достижения моей цели.
Пример рабочего процесса с использованием чрезвычайно упрощенного и готового набора данных:
> require(MASS)
> require(ggplot2)
> alldata<-read.csv("Fake data.csv",header=T,row.name=1)
> selectdata<-alldata[,2:10]
> alldata
Period Species.1 Species.2 Species.3 Species.4 Species.5 Species.6
Sample-1 Early 50 87 97 12 60 49
Sample-2 Early 41 90 36 52 36 27
Sample-3 Early 87 56 82 45 56 13
Sample-4 Early 37 47 78 29 53 34
Sample-5 Early 58 70 34 35 8 21
Sample-6 Early 94 82 48 16 27 26
Sample-7 Early 91 69 50 57 24 13
Sample-8 Early 63 38 86 20 28 11
Sample-9 Middle 4 19 55 99 86 38
Sample-10 Middle 29 25 10 93 37 54
Sample-11 Middle 48 12 59 73 39 92
Sample-12 Middle 31 6 34 81 39 54
Sample-13 Middle 29 40 26 52 34 84
Sample-14 Middle 1 46 15 97 67 41
Sample-15 Late 43 47 30 18 60 23
Sample-16 Late 45 10 49 2 2 45
Sample-17 Late 14 8 51 36 58 51
Sample-18 Late 41 51 32 47 23 43
Sample-19 Late 43 17 6 54 4 12
Sample-20 Late 20 25 1 29 35 2
Species.7 Species.8 Species.9
Sample-1 41 39 57
Sample-2 59 4 45
Sample-3 10 56 5
Sample-4 59 30 39
Sample-5 9 29 57
Sample-6 29 24 35
Sample-7 22 4 42
Sample-8 31 19 40
Sample-9 17 7 57
Sample-10 6 9 29
Sample-11 34 20 0
Sample-12 56 41 59
Sample-13 6 31 13
Sample-14 25 12 28
Sample-15 60 75 84
Sample-16 32 69 34
Sample-17 48 53 56
Sample-18 80 86 46
Sample-19 50 70 82
Sample-20 57 84 70
> biplot(selectca,cex=c(0.6,0.6))
> selectca<-corresp(selectdata,nf=5)
> PlotSample <- qplot(selectca$rscore[,1], selectca$rscore[,2], colour=factor(alldata$Period) )
> PlotTaxa<-qplot(selectca$cscore[,1], selectca$cscore[,2], label=colnames(selectdata), size=10, geom="text")
Биплот создаст этот участок: /r/10wk1a8/5
Образец PlotSample выглядит так: /r/i29cba/5
PlotTaxa выглядит так: /r/245bl9d/5
РЕДАКТИРОВАТЬ, поэтому не хватает представителя, чтобы публиковать фотографии и крошечные ссылки не принимаются (несмотря на https://meta.stackexchange.com/questions/60563/how-to-upload-images-on-stack-overflow). Так что, если вы добавите URL tinypic в начало этих кодов выше, вы попадете туда.
По сути, я хочу создать график биплота, но с цветовой кодировкой сэмплов, как в PlotSample.
1 ответ
Взгляните на ggvegan-пакет Гэвина Симпсона!
require(vegan)
require(ggvegan)
# some data
data(dune)
# CA
mod <- cca(dune)
# plot
autoplot(mod, geom = 'text')
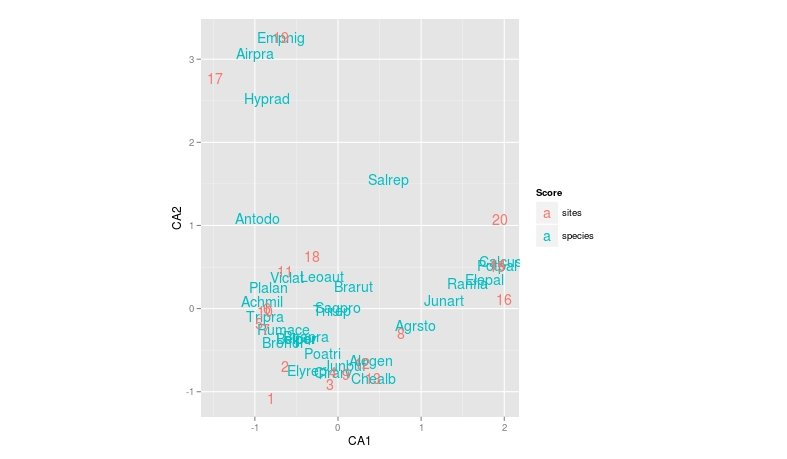
Для более точного контроля (или если вы хотите придерживаться corresp()Вы также можете взглянуть на код двух задействованных функций fortify.cca (который оборачивает данные в объектах cca в удобный для ggplot формат) и autoplot.cca для создания графика.
Если вы хотите сделать это с нуля, вам придется объединить обе оценки (сайты и виды) в один data.frame (см. Как fortify.cca делает это и извлечь соответствующие значения из corresp() объект) и использовать это, чтобы построить сюжет.