Метка оси X не отображается на кластеризованной дендрограмме в ggplot
Я выполнил кластеризацию дендрограммы, следуя предыдущему коду, который нашел в сети, но ось x на графике не отображается. Я хотел бы, чтобы значение отличия отображалось на оси X, но я не добился успеха.
females<-cervidae[cervidae$Sex=="female",]
dstf <- daisy(females[,9:14], metric = "euclidean", stand = FALSE)
hcaf <- hclust(dstf, method = "ave")
k <- 3
clustf <- cutree(hcaf,k=k) # k clusters
dendrf <- dendro_data(hcaf, type="rectangle") # convert for ggplot
clust.dff <- data.frame(label=rownames(females), cluster=factor(clustf),
females$Genus, females$Species)
dendrf[["labels"]] <- merge(dendrf[["labels"]],clust.dff, by="label")
rectf <- aggregate(x~cluster,label(dendrf),range)
rectf <- data.frame(rectf$cluster,rectf$x)
ymax <- mean(hcaf$height[length(hcaf$height)-((k-2):(k-1))])
fem=ggplot() +
geom_segment(data=segment(dendrf), aes(x=x, y=y, xend=xend, yend=yend)) +
geom_text(data=label(dendrf), aes(x, y, label= females.Genus, hjust=0,
color=females.Genus),
size=3) +
geom_rect(data=rectf, aes(xmin=X1-.3, xmax=X2+.3, ymin=0, ymax=ymax),
color="red", fill=NA)+
coord_flip() + scale_y_reverse(expand=c(0.2, 0)) +
theme_dendro() + scale_color_discrete(name="Genus") +
theme(legend.position="none")
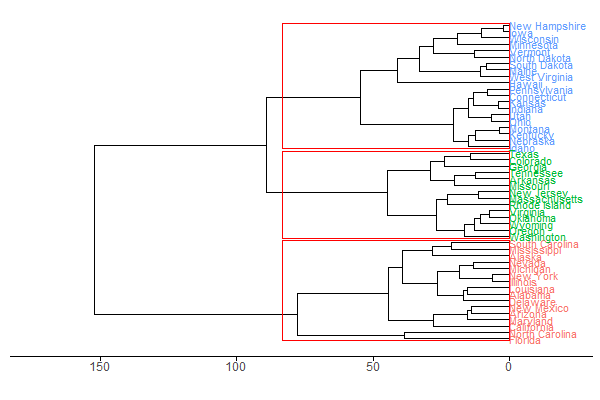
Вот как выглядит моя дендрограмма:
1 ответ
Решение
Ваш код включен theme_dendro(), который описан в файле справки как:
Устанавливает пустыми большинство параметров ggplot, возвращая пустые элементы темы для сетки панели, фона панели, названия оси, текста оси, линии оси и отметок оси.
Вы заставляете линию / текст / галочки оси X быть видимыми в theme():
ggplot() +
geom_segment(data=segment(dendrf), aes(x=x, y=y, xend=xend, yend=yend)) +
geom_text(data=label(dendrf), aes(x, y, label= label, hjust=0,
color=cluster),
size=3) +
geom_rect(data=rectf, aes(xmin=X1-.3, xmax=X2+.3, ymin=0, ymax=ymax),
color="red", fill=NA)+
coord_flip() +
scale_y_reverse(expand=c(0.2, 0)) +
theme_dendro() +
scale_color_discrete(name="Cluster") +
theme(legend.position="none",
axis.text.x = element_text(), # show x-axis labels
axis.ticks.x = element_line(), # show x-axis tick marks
axis.line.x = element_line()) # show x-axis lines
(Эта демонстрация использует встроенный набор данных, так как я не уверен, что такое cervidae. Код, использованный для создания этого, воспроизведен ниже:)
library(cluster); library(ggdendro); library(ggplot2)
hcaf <- hclust(dist(USArrests), "ave")
k <- 3
clustf <- cutree(hcaf,k=k) # k clusters
dendrf <- dendro_data(hcaf, type="rectangle") # convert for ggplot
clust.dff <- data.frame(label=rownames(USArrests),
cluster=factor(clustf))
dendrf[["labels"]] <- merge(dendrf[["labels"]],clust.dff, by="label")
rectf <- aggregate(x~cluster,label(dendrf),range)
rectf <- data.frame(rectf$cluster,rectf$x)
ymax <- mean(hcaf$height[length(hcaf$height)-((k-2):(k-1))])