Как создать граф границ решения для моделей kNN в пакете Caret?
Я хотел бы построить границу решения для модели, созданной пакетом Caret. В идеале, я хотел бы использовать метод общего случая для любой модели классификатора из Caret. Тем не менее, в настоящее время я работаю с методом kNN. Я включил код ниже, который использует набор данных по качеству вина из UCI, с которым я сейчас работаю.
Я нашел этот метод, который работает с общим методом kNN в R, но не могу понять, как сопоставить его с Caret -> https://stats.stackexchange.com/questions/21572/how-to-plot-decision-boundary-of-a-k-nearest-neighbor-classifier-from-elements-o/21602
library(caret)
set.seed(300)
wine.r <- read.csv('https://archive.ics.uci.edu/ml/machine-learning-databases/wine-quality/winequality-red.csv', sep=';')
wine.w <- read.csv('https://archive.ics.uci.edu/ml/machine-learning-databases/wine-quality/winequality-white.csv', sep=';')
wine.r$style <- "red"
wine.w$style <- "white"
wine <- rbind(wine.r, wine.w)
wine$style <- as.factor(wine$style)
formula <- as.formula(quality ~ .)
dummies <- dummyVars(formula, data = wine)
dummied <- data.frame(predict(dummies, newdata = wine))
dummied$quality <- wine$quality
wine <- dummied
numCols <- !colnames(wine) %in% c('quality', 'style.red', 'style.white')
low <- wine$quality <= 6
high <- wine$quality > 6
wine$quality[low] = "low"
wine$quality[high] = "high"
wine$quality <- as.factor(wine$quality)
indxTrain <- createDataPartition(y = wine[, names(wine) == "quality"], p = 0.7, list = F)
train <- wine[indxTrain,]
test <- wine[-indxTrain,]
corrMat <- cor(train[, numCols])
correlated <- findCorrelation(corrMat, cutoff = 0.6)
ctrl <- trainControl(
method="repeatedcv",
repeats=5,
number=10,
classProbs = T
)
t1 <- train[, -correlated]
grid <- expand.grid(.k = c(1:20))
knnModel <- train(formula,
data = t1,
method = 'knn',
trControl = ctrl,
tuneGrid = grid,
preProcess = 'range'
)
t2 <- test[, -correlated]
knnPred <- predict(knnModel, newdata = t2)
# How do I render the decision boundary?
1 ответ
Первый шаг - понять, что делает код, который вы связали! В самом деле, вы можете создать такой граф, не имея ничего общего с KNN.
Например, давайте просто предоставим некоторые образцы данных, где мы просто "раскрасим" нижний квадрант ваших данных.
Шаг 1
Создайте сетку. Принцип работы графика - создать точку для каждой координаты, чтобы мы знали, к какой группе она принадлежит. в R это делается с помощью expand.grid пройти через все возможные точки.
x1 <- 1:200
x2 <- 50:250
cgrid <- expand.grid(x1=x1, x2=x2)
# our "prediction" colours the bottom left quadrant
cgrid$prob <- 1
cgrid[cgrid$x1 < 100 & cgrid$x2 < 170, c("prob")] <- 0
Если бы это было известно, это было бы prob будет прогноз для этой конкретной точки.
Шаг 2
Теперь заговор это относительно просто. Вы должны соответствовать contour функция, поэтому вы сначала создаете матрицу с вероятностями.
matrix_val <- matrix(cgrid$prob,
length(x1),
length(x2))
Шаг 3
Затем вы можете продолжить, как это сделала ссылка:
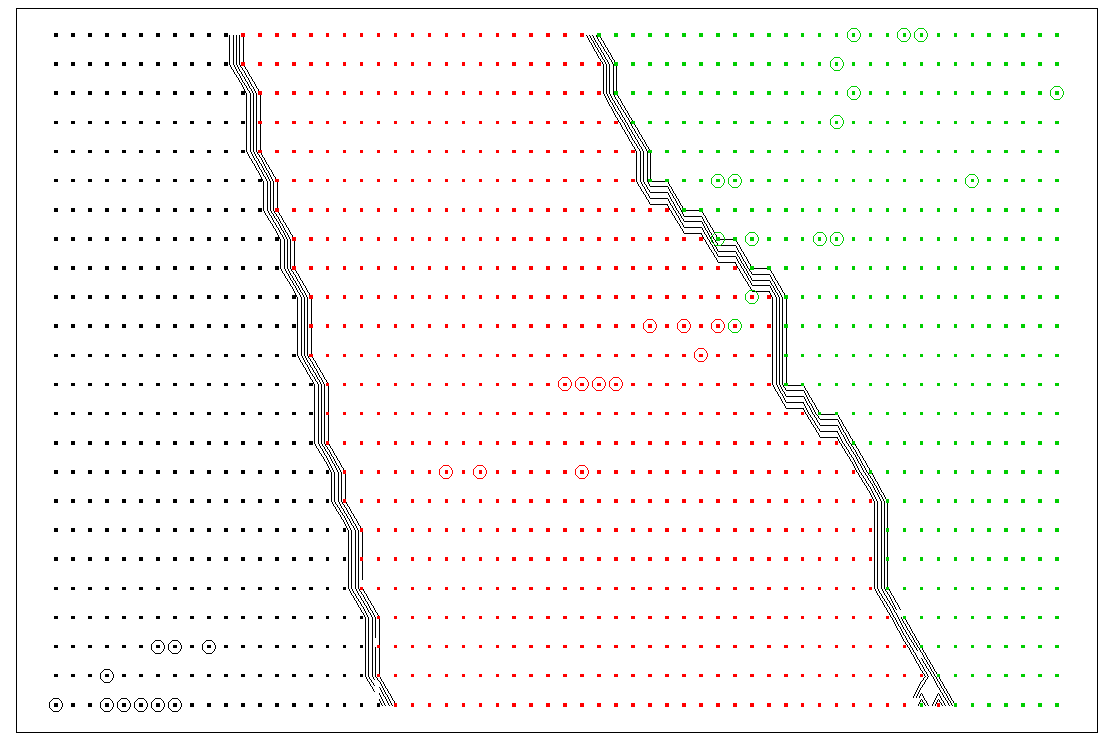
contour(x1, x2, matrix_val, levels=0.5, labels="", xlab="", ylab="", main=
"Some Picture", lwd=2, axes=FALSE)
gd <- expand.grid(x=x1, y=x2)
points(gd, pch=".", cex=1.2, col=ifelse(prob==1, "coral", "cornflowerblue"))
box()
выход:
Итак, вернемся к вашему конкретному примеру. Я собираюсь использовать радужную оболочку, потому что ваши данные не очень интересны для просмотра, но применяется тот же принцип. Для создания сетки вам нужно будет выбрать ось XY и оставить все остальное фиксированным!
knnModel <- train(Species ~.,
data = iris,
method = 'knn')
lgrid <- expand.grid(Petal.Length=seq(1, 5, by=0.1),
Petal.Width=seq(0.1, 1.8, by=0.1),
Sepal.Length = 5.4,
Sepal.Width=3.1)
Далее просто используйте функцию предсказания, как вы сделали выше.
knnPredGrid <- predict(knnModel, newdata=lgrid)
knnPredGrid = as.numeric(knnPredGrid) # 1 2 3
А затем построить график:
pl = seq(1, 5, by=0.1)
pw = seq(0.1, 1.8, by=0.1)
probs <- matrix(knnPredGrid, length(pl),
length(pw))
contour(pl, pw, probs, labels="", xlab="", ylab="", main=
"X-nearest neighbour", axes=FALSE)
gd <- expand.grid(x=pl, y=pw)
points(gd, pch=".", cex=5, col=probs)
box()
Это должно привести к выводу, как это:
Чтобы добавить результаты испытаний / тренировок из вашей модели, вы можете следить за тем, что я сделал. Разница лишь в том, что вам нужно добавить прогнозируемые точки (это не то же самое, что сетка, которая использовалась для создания границы).
library(caret)
data(iris)
indxTrain <- createDataPartition(y = iris[, names(iris) == "Species"], p = 0.7, list = F)
train <- iris[indxTrain,]
test <- iris[-indxTrain,]
knnModel <- train(Species ~.,
data = train,
method = 'knn')
pl = seq(min(test$Petal.Length), max(test$Petal.Length), by=0.1)
pw = seq(min(test$Petal.Width), max(test$Petal.Width), by=0.1)
# generates the boundaries for your graph
lgrid <- expand.grid(Petal.Length=pl,
Petal.Width=pw,
Sepal.Length = 5.4,
Sepal.Width=3.1)
knnPredGrid <- predict(knnModel, newdata=lgrid)
knnPredGrid = as.numeric(knnPredGrid)
# get the points from the test data...
testPred <- predict(knnModel, newdata=test)
testPred <- as.numeric(testPred)
# this gets the points for the testPred...
test$Pred <- testPred
probs <- matrix(knnPredGrid, length(pl), length(pw))
contour(pl, pw, probs, labels="", xlab="", ylab="", main="X-Nearest Neighbor", axes=F)
gd <- expand.grid(x=pl, y=pw)
points(gd, pch=".", cex=5, col=probs)
# add the test points to the graph
points(test$Petal.Length, test$Petal.Width, col=test$Pred, cex=2)
box()
Выход:
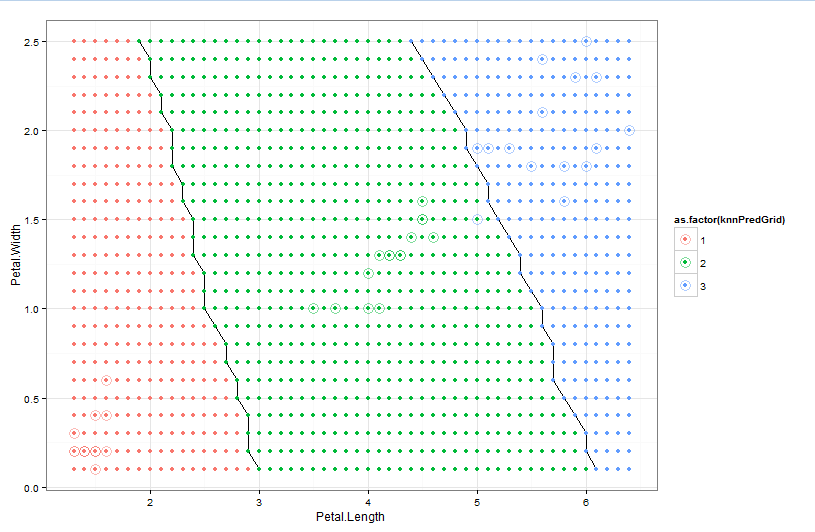
В качестве альтернативы вы можете использовать ggplot сделать графики, которые могут быть проще:
ggplot(data=lgrid) + stat_contour(aes(x=Petal.Length, y=Petal.Width, z=knnPredGrid),
bins=2) +
geom_point(aes(x=Petal.Length, y=Petal.Width, colour=as.factor(knnPredGrid))) +
geom_point(data=test, aes(x=test$Petal.Length, y=test$Petal.Width, colour=as.factor(test$Pred)),
size=5, alpha=0.5, shape=1)+
theme_bw()
Выход: