Создание подграфа с помощью igraph в R
Мне нужно получить подграф начальных узлов (входной список узлов; file.txt) и их первых интеракторов (соседей) из графа (g) с помощью igraph. К сожалению, я заканчиваю только одним узлом в подграфе, а не всеми остальными узлами и ребрами (вершинами), которые их связывают.
g<-read.graph("DATABASE.ncol",format="ncol",directed=FALSE) #load the data
g2<-simplify(g, remove.multiple=TRUE, remove.loops=TRUE) # Remove the self-loops in the data
DAT1 <- readLines("file.txt") #It provides a character vector right away
list_nodes_1 = neighbors(g2, DAT1) #list of nodes to be fetched in subnetwork
list_nodes_1 # 16
g3 <- induced.subgraph(graph=g2,vids=DAT1) #subnetwork construction
g3 # GRAPH UN-- 1 0 --; indicating only one node
plot (g3)
Любые предложения для получения всей подсети (включая узлы и вершины)? или есть какие-либо другие функции для создания подграфов?
DATABASE.ncol:
MAP2K4 FLNC
MYPN ACTN2
ACVR1 FNTA
GATA2 PML
RPA2 STAT3
ARF1 GGA3
ARF3 ARFIP2
ARF3 ARFIP1
XRN1 ALDOA
APP APPBP2
APLP1 DAB1
CITED2 TFAP2A
EP300 TFAP2A
APOB MTTP
ARRB2 RALGDS
CSF1R GRB2
PRRC2A GRB2
LSM1 NARS
SLC4A1 SLC4A1AP
BCL3 BARD1
Это простой текстовый файл с одним ребром в строке. Ребро определяется двумя символическими именами вершин, разделенными табуляцией:
file.txt
ALDOA
APLP1
GRB2
RPA2
FLNC
BCL3
APP
RALGDS
PRRC2A
NARS
LSM1
GGA3
FNTA
2 ответа
Решение
Я не уверен, что полностью понял вашу проблему, поэтому я создал (надеюсь) очевидный пример:
# for example reproducibility
set.seed(123)
# create a fake undirected graph
D <- read.table(
sep=',',
header=T,
text=
'from,to
A,B
A,C
D,E
F,G
H,I')
g1 <- graph.data.frame(D,directed=F)
plot(g1)
# we want a sub-network containing the floowing nodes:
subv <- c('A','B','H')
# first method:
# create a sub-network composed by ONLY the nodes in subv and the edges
# between them
g2 <- induced.subgraph(graph=g1,vids=subv)
plot(g2)
# second method:
# create a sub-network composed by the nodes in subv and, if some of them is
# connected to other nodes (even if not in subv), take also them
# (and of course include all the edges among this bunch of nodes).
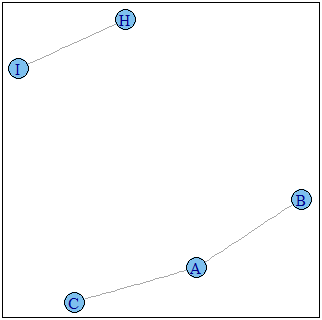
sg1 <- decompose.graph(g1,mode="weak")
neighverts <- unique(unlist(sapply(sg1,FUN=function(s){if(any(V(s)$name %in% subv)) V(s)$name else NULL})))
g3 <- induced.subgraph(graph=g1,vids=neighverts)
plot(g3)
График g1:
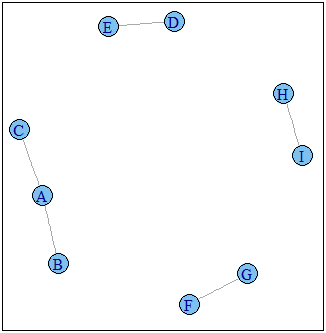
График g2:
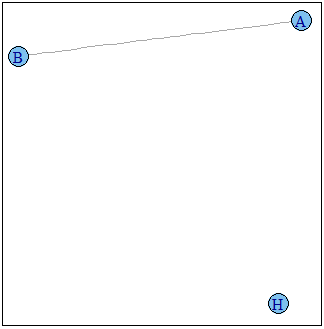
График g3:
Для этого есть встроенная функция igraph. Попробуйте make_ego_graph():
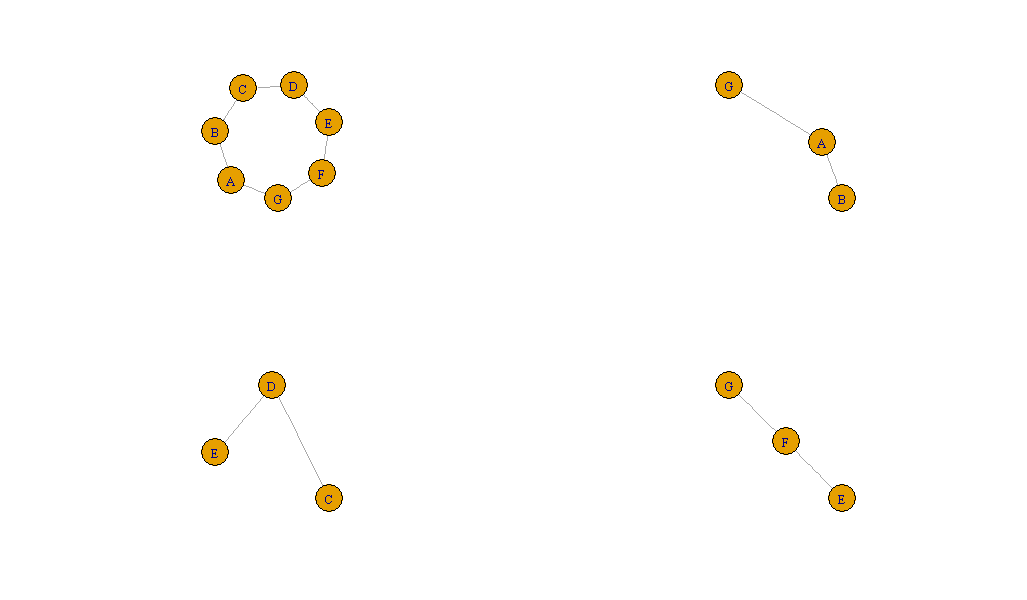
library(igraph)
graph <- make_ring(7)
V(graph)$name <- c("A", "B", "C", "D", "E", "F", "G")
# Get the list of induced subgraphs
subgraph <- make_ego_graph(graph, order=1, c("A", "D", "F"))