PCA prcomp: как получить график с PC1 на PC3
В моем скрипте для PCA (ниже) я всегда получаю график PC1 против PC2.
mydata.pca <- prcomp(mydata.fixed, center = TRUE, scale. = TRUE)
g <- ggbiplot(mydata.pca, obs.scale = 1, var.scale = 1,
groups = mysamples, ellipse = FALSE,
circle = FALSE, var.axes=FALSE)
g <- g + scale_color_discrete(name = '')
g <- g + theme(legend.direction ='horizontal',
legend.position = 'top')
g <- g + theme_bw()
Тем не менее, когда я запускаю что-то вроде:
summary(mydata.pca)
Я вижу всю информацию для ПК1 на ПК4. Как я могу изменить свой скрипт ggbioplot, чтобы получить график ПК1 против ПК3?
1 ответ
Аргумент выбора ggbiplot это то, что вы ищете:
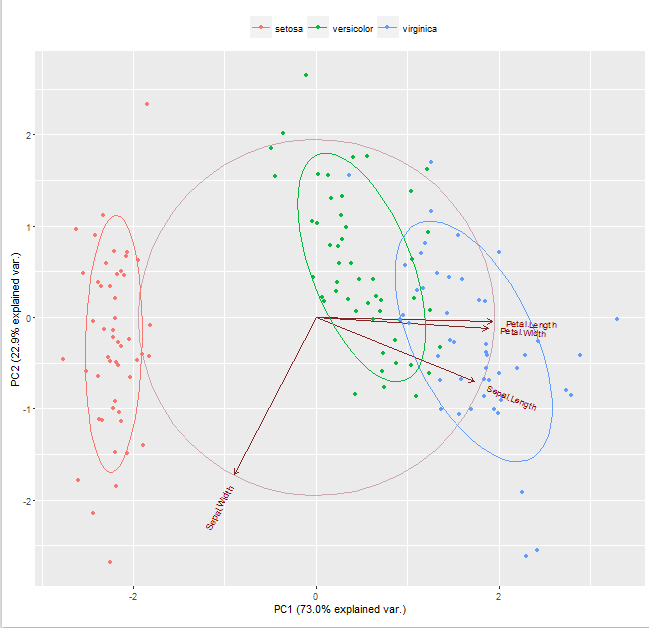
iris_pca <- prcomp(iris[, 1:4], center = TRUE, scale. = T)
# PC1 vs PC 2
ggbiplot(iris_pca, choices = c(1,2), obs.scale = 1, var.scale = 1,
groups = iris$Species, ellipse = TRUE,
circle = TRUE) + scale_color_discrete(name = '') + theme(legend.direction = 'horizontal',
legend.position = 'top')
# PC1 vs PC 3
ggbiplot(iris_pca, choices = c(1,3), obs.scale = 1, var.scale = 1,
groups = iris$Species, ellipse = TRUE,
circle = TRUE) + scale_color_discrete(name = '') + theme(legend.direction = 'horizontal',
legend.position = 'top')