Как использовать настраиваемую функцию кластеризации и метрики для pheatmap
У меня есть следующая функция
library(pheatmap)
plot_kmeans_heatmap <- function (dat=NULL, k=3, nstart=20) {
# Data wrangling ----------------------------------------------------------
set.seed(1)
dat_m <- kmeans(dat, k, nstart = nstart)
m.kmeans <- cbind(dat, dat_m$cluster)
clusterid_col <- dim(m.kmeans)[2]
# Ordering matrix ----------------------------------------------------------------
# order the data row index according the last column (i.e. cluster ID)
ordered_idx <- order(m.kmeans[,clusterid_col])
# order the matrix according to the order of the last column
m.kmeans<- m.kmeans[ordered_idx,]
# Plot it -----------------------------------------------------------------
# Annotate row with class
annot_row <- m.kmeans %>%
transmute(gene_class=paste0("C", `dat_m$cluster`), rn=row.names(.))
rownames(annot_row) <- annot_row$rn
annot_row$rn <- NULL
pheatmap(m.kmeans[,1:clusterid_col-1],
scale="row",
cluster_rows=F,
cluster_cols=F,
legend=T,
annotation_row = annot_row,
border_color = F,
show_rownames=F,
show_colnames=T)
}
Со следующим кодом:
irisn <- iris[sample(nrow(iris), 30), ]
dat <- irisn[,1:4]
plot_kmeans_heatmap(dat=dat, k=6, nstart=1)
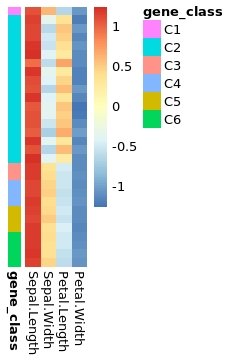
Он производит этот сюжет:
У меня вопрос вместо использования K-средних, я бы хотел использовать собственную функцию:
hclustfun = function(x) hclust(x, method='ward.D2')
distfun = function(x) as.dist(1-cor(x, method='pearson'))
Как я могу включить это в pheatmap?