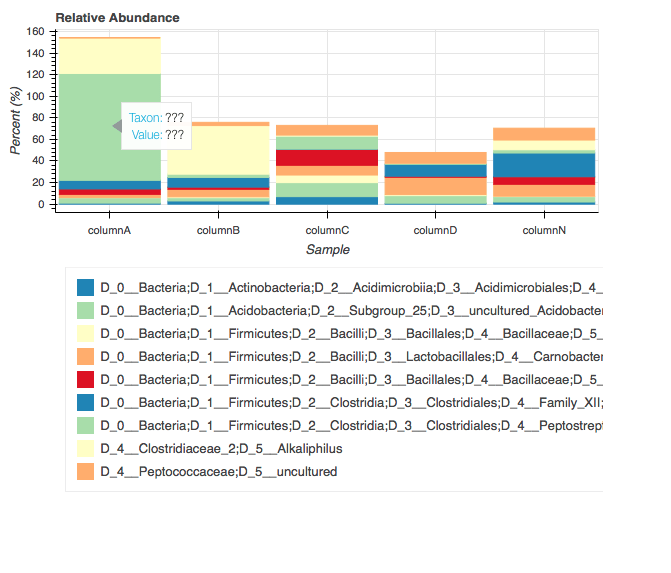
Легенда питона боке вне размера сюжета
Я новичок в Python, и кто-то помог мне с этим кодом, но я хочу изменить какой-то параметр:
Сначала размер легенды выходит из сюжета, иногда легенда слишком велика (пример: D_0__Bacteria;D_1__Firmicutes;D_2__Clostridia;D_3__Clostridiales;D_4__Peptostreptococcaceae;D_5__Acetoanaerobium), а иногда и совсем короткая (Iceto), иногда довольно короткая Легенда автоматически исправляет размер (на рисунке легенда не завершена)!!!.
Во-вторых, метка, которая появляется при наведении указателя на зону панели, показывает имя и значение соответствующих данных (hover.tooltips = [('Taxon','example: Acetoanaerobium'),('Value',' пример соответствующего значения: 99')])
третье: положение сюжета (рисунок), посередине
#!/usr/bin/env python
import pandas as pd
from bokeh.io import show, output_file
from bokeh.models import ColumnDataSource
from bokeh.plotting import figure
from bokeh.core.properties import value
from bokeh.palettes import Spectral
from bokeh.models import HoverTool
#from bokeh.plotting import figure, output_file, show, ColumnDataSource
import itertools
import sys
data_in = sys.argv[1]
data_out = sys.argv[2]
output_file(data_out + ".html")
df = pd.read_csv(data_in, sep='\t')
df.set_index('#OTU_ID', inplace=True)
#print(df)
s_data = df.columns.values # linia de samples !!!
t_data = df.index.values #columna de datos
#print(s_data)
#print(t_data)
# You have two rows with 'uncultured' data. I added these together.
# This may or may not be what you want.
df = df.groupby('#OTU_ID')[s_data].transform('sum')
#grouped = df.groupby(["columnA", "columnB"], as_index=False).count()
#print(grouped)
# create a color iterator
# See https://stackru.com/q/39839409/50065
# choose an appropriate pallete from
# https://bokeh.pydata.org/en/latest/docs/reference/palettes.html
# if you have a large number of organisms
color_iter = itertools.cycle(Spectral[5])
colors = [next(color_iter) for organism in t_data]
# create a ColumnDataSource
data = {'xs': list(s_data)}
for organism in t_data:
data[organism] = list(df.loc[organism])
source = ColumnDataSource(data=data)
#print(organism)
# create our plot
plotX = figure(x_range=s_data, plot_height=500, title="Relative Abundance",
toolbar_location=None, tools="hover")
plotX.vbar_stack(t_data, x='xs', width=0.93, source=source,
legend=[value(x) for x in t_data], color=colors)
plotX.xaxis.axis_label = 'Sample'
plotX.yaxis.axis_label = 'Percent (%)'
plotX.legend.location = "bottom_left"
plotX.legend.orientation = "vertical"
# Position the legend outside the plot area
# https://stackru.com/questions/48240867/how-can-i-make-legend-outside-plot-area-with-stacked-bar
new_legend = plotX.legend[0]
plotX.legend[0].plot = None
plotX.add_layout(new_legend, 'below')
hover = plotX.select(dict(type=HoverTool))
hover.tooltips = [('Taxon','unknow_var'),('Value','unknow_var')]
# I don't know what variable to addd in unknow_var
show(plotX)
в файле является file.txt, файл с разделителями табуляции, как:
#OTU_ID columnA columnB columnC columnD columnN
D_0__Bacteria;D_1__Actinobacteria;D_2__Acidimicrobiia;D_3__Acidimicrobiales;D_4__uncultured;D_5__uncultured_bacterium 1 3 7 0.9 2
D_0__Bacteria;D_1__Acidobacteria;D_2__Subgroup_25;D_3__uncultured_Acidobacteria_bacterium;D_0__Bacteria;D_1__Actinobacteria;D_2__Actinobacteria;D_3__Streptomycetales;D_4__Streptomycetaceae;D_5__Kitasatospora 5 3 13 7 5
D_0__Bacteria;D_1__Firmicutes;D_2__Bacilli;D_3__Bacillales;D_4__Bacillaceae;D_5__Anoxybacillus 0.1 0.8 7 1 0.4
D_0__Bacteria;D_1__Firmicutes;D_2__Bacilli;D_3__Lactobacillales;D_4__Carnobacteriaceae;D_5__Carnobacterium 3 7 9 16 11
D_0__Bacteria;D_1__Firmicutes;D_2__Bacilli;D_3__Bacillales;D_4__Bacillaceae;D_5__Oceanobacillus 5 2 15 1 7
D_0__Bacteria;D_1__Firmicutes;D_2__Clostridia;D_3__Clostridiales;D_4__Family_XII;D_5__Fusibacter 8 9 0 11 22
D_0__Bacteria;D_1__Firmicutes;D_2__Clostridia;D_3__Clostridiales;D_4__Peptostreptococcaceae;D_5__Acetoanaerobium 99 3 12 1 3
D_4__Clostridiaceae_2;D_5__Alkaliphilus 33 45 1 0 9
D_4__Peptococcaceae;D_5__uncultured 0 3 9 10 11
в этом примере значение не в%, как говорят в y-легенде, значения являются лишь примером!!!
Спасибо большое!!!
1 ответ
Легенды Bokeh не имеют автоматического размера (нет возможности сделать это). Вам нужно будет установить ширину легенды, чтобы она была достаточно широкой, чтобы охватить любой ярлык. Кроме того, поскольку они нарисованы на том же холсте, что и график, вам нужно будет сделать график шире, чтобы он соответствовал ширине, установленной в легенде. Если вы не хотите, чтобы центральная площадь участка увеличивалась, вы можете установить различные min_border, min_border_left значения на участке, чтобы освободить место вокруг внутренней области графика.
В качестве альтернативы, вместо изменения размера сюжета и легенды, вы можете уменьшить размер шрифта легенды.
p.legend.label_text_font_size = "8px"