Нечеткая кластеризация на Python с набором данных Iris
Я работаю над нечеткой кластеризацией c-средних в наборе данных радужной оболочки, однако не могу визуализировать из-за некоторых ошибок. Используя этот учебник, я написал следующее для радужной оболочки, однако он показывает ошибку под названием "AttributeError: shape". Это мой код:
from sklearn import datasets
from sklearn.cluster import KMeans
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import sklearn.metrics as sm
import skfuzzy as fuzz
iris = datasets.load_iris()
x = pd.DataFrame(iris.data, columns=['Sepal Length', 'Sepal Width', 'Petal Length', 'Petal Width'])
y = pd.DataFrame(iris.target, columns=['Target'])
plt.figure(figsize=(6, 3))
model =fuzz.cluster.cmeans(iris,3,2,error=0.005,maxiter=1000,init=None,seed=None)
model.fit(x)
plt.show()
Я предположил, что передачи параметра в переменной модели будет достаточно, однако это показывает вышеуказанную ошибку. Если возможно, не могли бы вы показать, где я ошибаюсь? Как это исправить? Я очень ценю вашу помощь!
1 ответ
Сначала я попытался предварительно обработать данные, я создал хороший график, просто следовал инструкциям и выполняю SVD, чтобы уменьшить размерность до двух, а затем начал строить график. Похоже, что для уроков вам нужны только два измерения (х, у). Вам не нужно делать model.fit() Я не нашел такого рода команды в документации, вот код:
import numpy as np, pandas as pd, os
import matplotlib
import matplotlib.pyplot as plt
import itertools
from sklearn.metrics import confusion_matrix
import statsmodels.api as sm
import statsmodels.formula.api as smf
from sklearn.preprocessing import StandardScaler
from sklearn.decomposition import [![TruncatedSVD
from skle][1]][1]arn.preprocessing import Normalizer
import skfuzzy as fuzz
from sklearn import datasets
################################################################################
iris = datasets.load_iris()
x = pd.DataFrame(iris.data, columns=['Sepal Length', 'Sepal Width', 'Petal Length', 'Petal Width'])
y = pd.DataFrame(iris.target, columns=['Target'])
scaler = StandardScaler()
X_std = scaler.fit_transform(x)
lsa = TruncatedSVD(2, algorithm = 'arpack')
dtm_lsa = lsa.fit_transform(X_std)
dtm_lsa = Normalizer(copy=False).fit_transform(dtm_lsa)
a= pd.DataFrame(dtm_lsa, columns = ["component_1","component_2"])
a['targets']=y
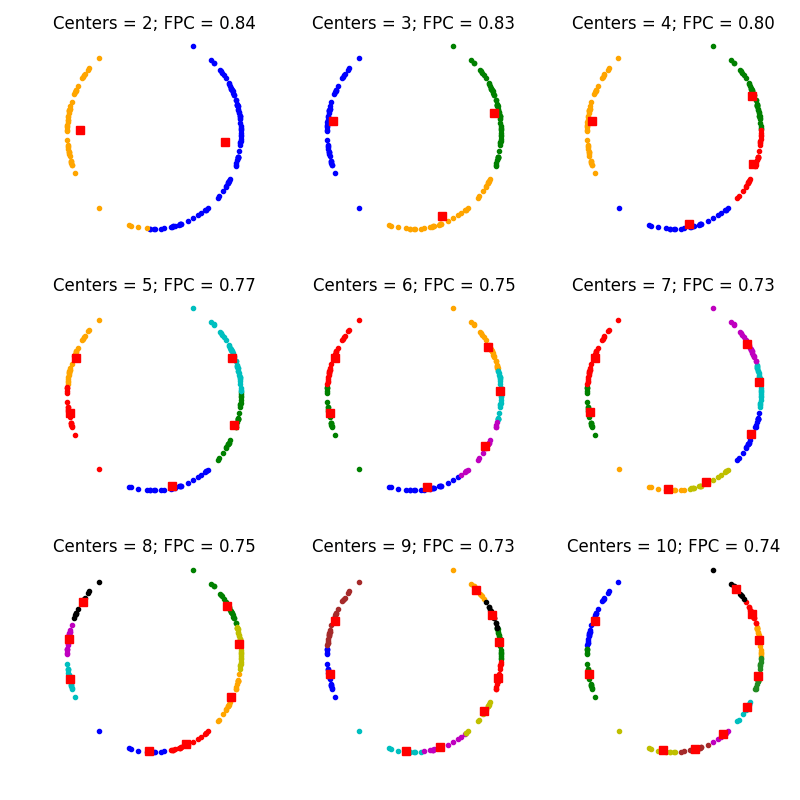
fig1, axes1 = plt.subplots(3, 3, figsize=(8, 8))
alldata = np.vstack((a['component_1'], a['component_2']))
fpcs = []
colors = ['b', 'orange', 'g', 'r', 'c', 'm', 'y', 'k', 'Brown', 'ForestGreen']
for ncenters, ax in enumerate(axes1.reshape(-1), 2):
cntr, u, u0, d, jm, p, fpc = fuzz.cluster.cmeans(
alldata, ncenters, 2, error=0.005, maxiter=1000, init=None)
# Store fpc values for later plots
fpcs.append(fpc)
# Plot assigned clusters, for each data point in training set
cluster_membership = np.argmax(u, axis=0)
for j in range(ncenters):
ax.plot(a['component_1'][cluster_membership == j],
a['component_2'][cluster_membership == j], '.', color=colors[j])
# Mark the center of each fuzzy cluster
for pt in cntr:
ax.plot(pt[0], pt[1], 'rs')
ax.set_title('Centers = {0}; FPC = {1:.2f}'.format(ncenters, fpc))
ax.axis('off')
fig1.tight_layout()
fig1.savefig('iris_dataset.png')