Добавление меток в научной нотации к лесным участкам с использованием пакета metafor
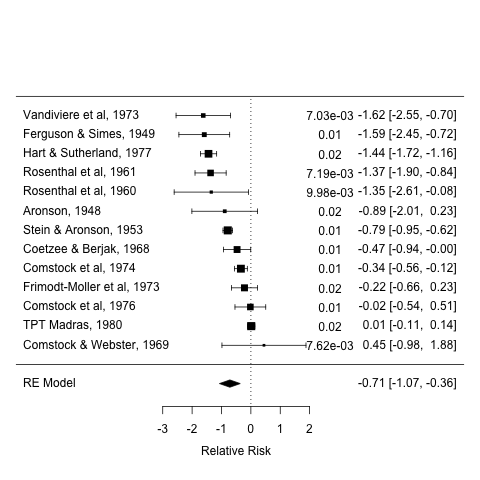
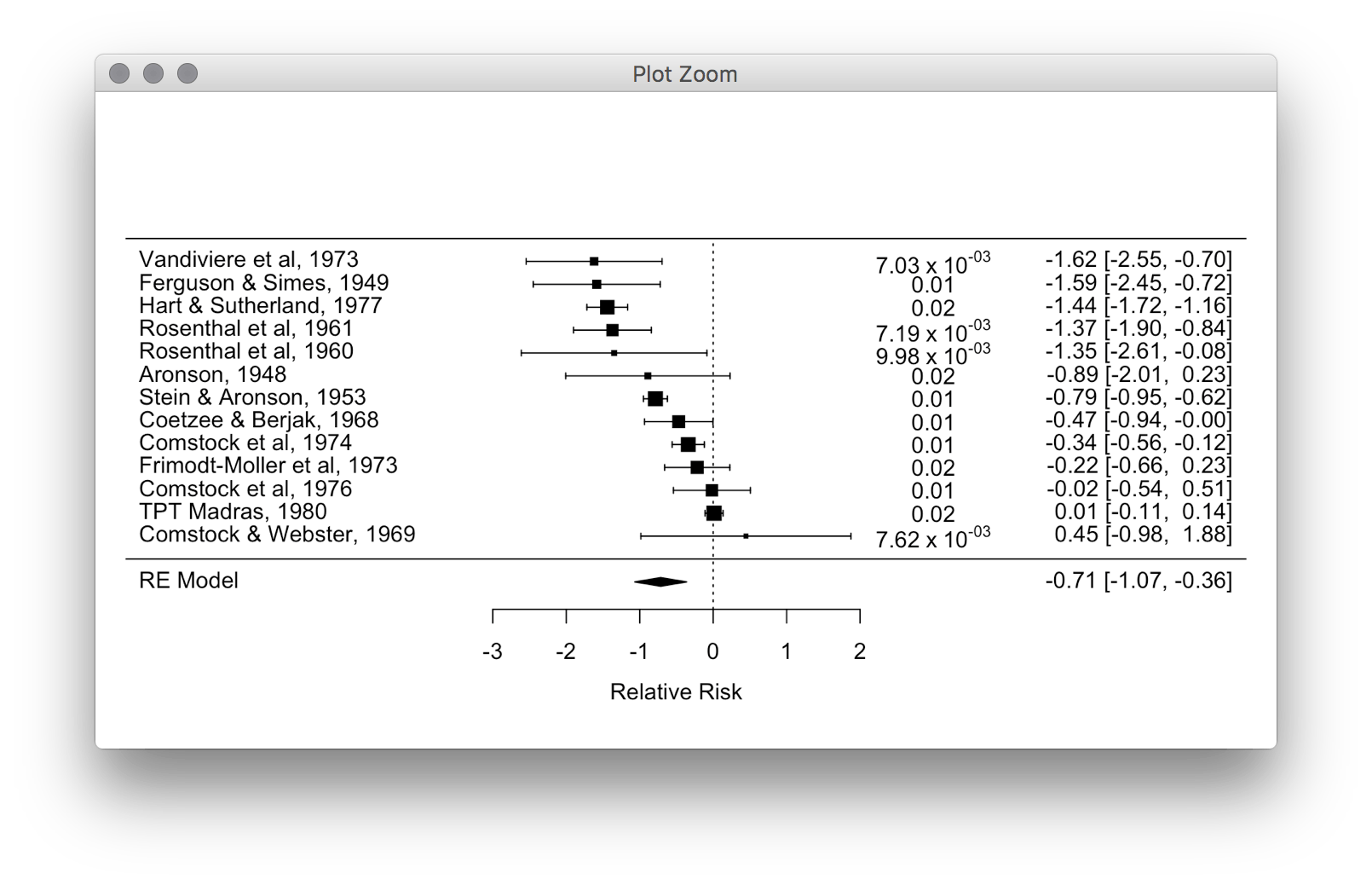
Так что я делаю мета-анализ с использованием meta.for пакет в R, Я готовлю цифры для публикации в научном журнале и хотел бы добавить p-значения к своим лесным графикам, но с научной аннотацией в формате
x10-04e-04
Однако аргумент ilab в forest функция не принимает expression объекты класса, но только векторы
Вот пример:
library(metafor)
data(dat.bcg)
## REM
res <- rma(ai = tpos, bi = tneg, ci = cpos, di = cneg, data = dat.bcg,
measure = "RR",
slab = paste(author, year, sep = ", "), method = "REML")
# MADE UP PVALUES
set.seed(513)
p.vals <- runif(nrow(dat.bcg), 1e-6,0.02)
# Format pvalues so only those bellow 0.01 are scientifically notated
p.vals <- ifelse(p.vals < 0.01,
format(p.vals,digits = 3,scientific = TRUE,trim = TRUE),
format(round(p.vals, 2), nsmall=2, trim=TRUE))
## Forest plot
forest(res, ilab = p.vals, ilab.xpos = 3, order = "obs", xlab = "Relative Risk")
Я хочу, чтобы научное обозначение p-значений было отформатировано как
x10-04expression() но это дает Error in cbind(ilab) : cannot create a matrix from type 'expression' что имеет смысл, потому что файл справки на forest указывает, что ilab аргумент должен быть вектором.Любые идеи о том, как я могу это исправить или обойти это?
1 ответ
Решение
Хакерское решение было бы
forest.rma <- edit(forest.rma)
Перейти к строке 574 и изменить
## line 574
text(ilab.xpos[l], rows, ilab[, l], pos = ilab.pos[l],
в
text(ilab.xpos[l], rows, parse(text = ilab[, l]), pos = ilab.pos[l],
исправить ваши р-значения и сюжет
p.vals <- gsub('e(.*)', '~x~10^{"\\1"}', p.vals)
forest(res, ilab = p.vals, ilab.xpos = 3, order = "obs", xlab = "Relative Risk")