Как нарисовать границы решения в SVM sklearn данных в Python?
Я читаю данные электронной почты из учебного набора и создаю train_matrix, train_labels и test_labels. Теперь, как мне отобразить границу решения, используя matplot в python. Я использую SVM Sklearn. Есть онлайн пример для предварительно заданных наборов данных через радужную оболочку. Но заговор терпит неудачу на пользовательских данных. Вот мой код
Ошибка:
Traceback (most recent call last):
File "classifier-plot.py", line 115, in <module>
Z = Z.reshape(xx.shape)
ValueError: cannot reshape array of size 260 into shape (150,1750)
Код:
import os
import numpy as np
from collections import Counter
from sklearn import svm
import matplotlib
import matplotlib.pyplot as plt
from sklearn.metrics import accuracy_score
def make_Dictionary(root_dir):
all_words = []
emails = [os.path.join(root_dir,f) for f in os.listdir(root_dir)]
for mail in emails:
with open(mail) as m:
for line in m:
words = line.split()
all_words += words
dictionary = Counter(all_words)
list_to_remove = dictionary.keys()
for item in list_to_remove:
if item.isalpha() == False:
del dictionary[item]
elif len(item) == 1:
del dictionary[item]
dictionary = dictionary.most_common(3000)
return dictionary
def extract_features(mail_dir):
files = [os.path.join(mail_dir,fi) for fi in os.listdir(mail_dir)]
features_matrix = np.zeros((len(files),3000))
train_labels = np.zeros(len(files))
count = 0;
docID = 0;
for fil in files:
with open(fil) as fi:
for i,line in enumerate(fi):
if i == 2:
words = line.split()
for word in words:
wordID = 0
for i,d in enumerate(dictionary):
if d[0] == word:
wordID = i
features_matrix[docID,wordID] = words.count(word)
train_labels[docID] = 0;
filepathTokens = fil.split('/')
lastToken = filepathTokens[len(filepathTokens) - 1]
if lastToken.startswith("spmsg"):
train_labels[docID] = 1;
count = count + 1
docID = docID + 1
return features_matrix, train_labels
TRAIN_DIR = "../train-mails"
TEST_DIR = "../test-mails"
dictionary = make_Dictionary(TRAIN_DIR)
print "reading and processing emails from file."
features_matrix, labels = extract_features(TRAIN_DIR)
test_feature_matrix, test_labels = extract_features(TEST_DIR)
model = svm.SVC(kernel="rbf", C=10000)
print "Training model."
features_matrix = features_matrix[:len(features_matrix)/10]
labels = labels[:len(labels)/10]
#train model
model.fit(features_matrix, labels)
predicted_labels = model.predict(test_feature_matrix)
print "FINISHED classifying. accuracy score : "
print accuracy_score(test_labels, predicted_labels)
##----------------
h = .02 # step size in the mesh
# we create an instance of SVM and fit out data. We do not scale our
# data since we want to plot the support vectors
C = 1.0 # SVM regularization parameter
X = features_matrix
y = labels
svc = model.fit(X, y)
#svm.SVC(kernel='linear', C=C).fit(X, y)
# create a mesh to plot in
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = y[:].min() - 1, y[:].max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, h),
np.arange(y_min, y_max, h))
# title for the plots
titles = ['SVC with linear kernel']
Z = predicted_labels#svc.predict(np.c_[xx.ravel(), yy.ravel()])
# Put the result into a color plot
Z = Z.reshape(xx.shape)
plt.contourf(xx, yy, Z, cmap=plt.cm.coolwarm, alpha=0.8)
# Plot also the training points
plt.scatter(X[:, 0], X[:, 1], c=y, cmap=plt.cm.coolwarm)
plt.xlabel('Sepal length')
plt.ylabel('Sepal width')
plt.xlim(xx.min(), xx.max())
plt.ylim(yy.min(), yy.max())
plt.xticks(())
plt.yticks(())
plt.title(titles[0])
plt.show()
1 ответ
В учебнике, который вы читали Z вычисляется путем применения классификатора к набору векторов признаков, сгенерированных для формирования регулярного NxM сетка. Это делает сюжет гладким.
Когда вы заменили
Z = svc.predict(np.c_[xx.ravel(), yy.ravel()])
с
Z = predicted_labels
вы заменили эту регулярную сетку на прогнозы, сделанные в вашем наборе данных. Следующая строка завершилась ошибкой, так как не смогла изменить размер массива len(files) для NxM матрица. Нет причины len(files) = NxM,
Есть причина, по которой вы не могли следовать учебнику напрямую. Ваше измерение данных равно 3000, поэтому границей вашего решения будет 2999-мерная гиперплоскость в 3000-мерном пространстве. Это не легко визуализировать.
В учебнике размер равен 4, а для визуализации он уменьшен до 2. Лучший способ уменьшить размерность ваших данных зависит от данных. В уроке мы просто выбираем первые два компонента 4-мерного вектора.
Другим вариантом, который хорошо работает во многих случаях, является использование анализа основных компонентов, чтобы уменьшить размерность данных.
from sklearn.decomposition import PCA
pca = PCA(n_components = 2)
pca.fit(features_matrix, labels)
reduced_matrix = pca.fit_transform(features_matrix, labels)
model.fit(reduced_matrix, labels)
Такая модель может быть использована для 2D визуализации. Вы можете просто следовать руководству и определить
Z = model.predict(np.c_[xx.ravel(), yy.ravel()])
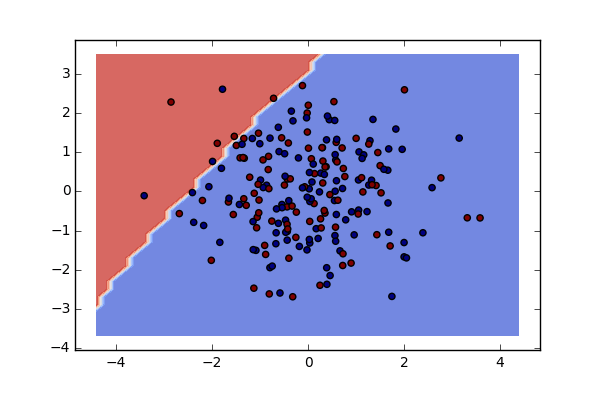
Полный, но не очень впечатляющий пример
У нас нет доступа к вашим данным электронной почты, поэтому для иллюстрации мы могли бы просто использовать случайные данные.
from sklearn import svm
from sklearn.decomposition import PCA
# initialize algorithms and data with random
model = svm.SVC(gamma=0.001,C=100.0)
pca = PCA(n_components = 2)
rng = np.random.RandomState(0)
U = rng.rand(200, 2000)
v = (rng.rand(200)*2).astype('int')
pca.fit(U,v)
U2 = pca.fit_transform(U,v)
model.fit(U2,v)
# generate grid for plotting
h = 0.2
x_min, x_max = U2[:,0].min() - 1, U2[:, 0].max() + 1
y_min, y_max = U2[:,1].min() - 1, U2[:, 1].max() + 1
xx, yy = np.meshgrid(
np.arange(x_min, x_max, h),
np.arange(y_min, y_max, h))
# create decision boundary plot
Z = s.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
contourf(xx,yy,Z,cmap=plt.cm.coolwarm, alpha=0.8)
scatter(U2[:,0],U2[:,1],c=v)
show()
Будет производить решение границы, которая выглядит не очень впечатляюще.
Действительно, первые два основных компонента охватывают только около 1% информации, содержащейся в данных
>>> print(pca.explained_variance_ratio_)
[ 0.00841935 0.00831764]
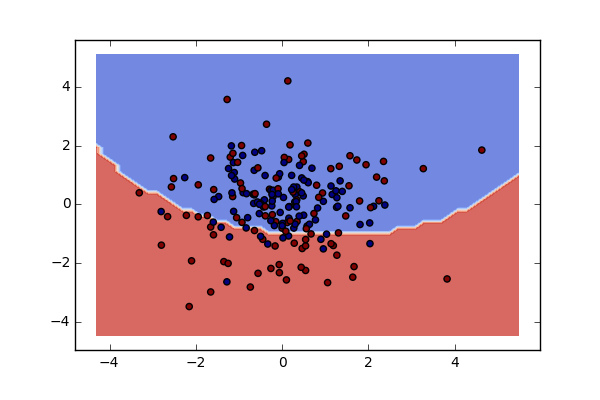
Если сейчас вы введете немного замаскированной асимметрии, вы уже увидите эффект.
Измените данные, чтобы ввести смещения только по одной координате, случайно выбранной для каждого объекта
random_shifts = (rng.rand(2000)*200).astype('int')
for i in range(MM):
if v[i] == 1:
U[i,random_shifts[i]] += 5.0
А применяя PCA, вы получите более информативную картину.
Обратите внимание, что здесь первые два основных компонента уже объясняют около 5% дисперсии, а красная часть изображения содержит гораздо больше красных точек, чем синих.