Создать ROC-кривую в Eclipse(weka)
Я использовал следующий код для обучения / тестирования некоторых классификаторов:
public class WekaTest {
public static BufferedReader readDataFile(String filename)
{
BufferedReader inputReader = null;
try
{
inputReader = new BufferedReader(new FileReader(filename));
}
catch (FileNotFoundException ex)
{
System.err.println("File not found: " + filename);
}
return inputReader;
}
public static Evaluation classify(Classifier model,
Instances trainingSet, Instances testingSet) throws Exception {
Evaluation evaluation = new Evaluation(trainingSet);
model.buildClassifier(trainingSet);
evaluation.evaluateModel(model, testingSet);
return evaluation;
}
public static double calculateAccuracy(FastVector predictions) {
double correct = 0;
for (int i = 0; i < predictions.size(); i++) {
NominalPrediction np = (NominalPrediction) predictions.elementAt(i);
if (np.predicted() == np.actual()) {
correct++;
}
}
return 100 * correct / predictions.size();
}
public static Instances[][] crossValidationSplit(Instances data, int numberOfFolds) {
Instances[][] split = new Instances[2][numberOfFolds];
Random random = new Random();
for (int i = 0; i < numberOfFolds; i++)
{
split[0][i] = data.trainCV(numberOfFolds, i, random);
split[1][i] = data.testCV(numberOfFolds, i);
}
return split;
}
public static void main(String[] args) throws Exception
{
BufferedReader datafile = readDataFile("training_1.arff");
Instances data = new Instances(datafile);
data.setClassIndex(data.numAttributes() - 1);
// Do 10-split cross validation
Instances[][] split = crossValidationSplit(data, 10);
// Separate split into training and testing arrays
Instances[] trainingSplits = split[0];
Instances[] testingSplits = split[1];
// Use a set of classifiers
Classifier[] models = {
// new J48(), // a decision tree
// new PART(),
// new DecisionTable(),//decision table majority classifier
// new DecisionStump(), //one-level decision tree
new NaiveBayes(),
// new AdaBoostM1()
new RandomForest()
// new LMT()
};
// Run for each model
for (int j = 0; j < models.length; j++)
{
// Collect every group of predictions for current model in a FastVector
FastVector predictions = new FastVector();
// For each training-testing split pair, train and test the classifier
for (int i = 0; i < trainingSplits.length; i++)
{
Evaluation validation = classify(models[j], trainingSplits[i], testingSplits[i]);
predictions.appendElements(validation.predictions());
System.out.println(validation.toMatrixString());
// Uncomment to see the summary for each training-testing pair.
// System.out.println(models[j].toString());
// generate curve
ThresholdCurve tc = new ThresholdCurve();
int classIndex = 0;
Instances result = tc.getCurve(validation.predictions(), classIndex);
System.out.println("tPR :"+validation.truePositiveRate(classIndex));
System.out.println("fNR :"+validation.falseNegativeRate(classIndex));
// plot curve
ThresholdVisualizePanel vmc = new ThresholdVisualizePanel();
vmc.setROCString("(Area under ROC = " +
Utils.doubleToString(tc.getROCArea(result), 4) + ")");
vmc.setName(result.relationName());
PlotData2D tempd = new PlotData2D(result);
tempd.setPlotName(result.relationName());
tempd.addInstanceNumberAttribute();
// specify which points are connected
boolean[] cp = new boolean[result.numInstances()];
for (int n = 1; n < cp.length; n++)
cp[n] = true;
tempd.setConnectPoints(cp);
// add plot
vmc.addPlot(tempd);
// display curve
String plotName = vmc.getName();
final javax.swing.JFrame jf =
new javax.swing.JFrame("Weka Classifier Visualize: "+plotName);
jf.setSize(500,400);
jf.getContentPane().setLayout(new BorderLayout());
jf.getContentPane().add(vmc, BorderLayout.CENTER);
jf.addWindowListener(new java.awt.event.WindowAdapter() {
public void windowClosing(java.awt.event.WindowEvent e) {
jf.dispose();
}
});
jf.setVisible(true);
}
// Calculate overall accuracy of current classifier on all splits
double accuracy = calculateAccuracy(predictions);
// Print current classifier's name and accuracy in a complicated,
// but nice-looking way.
System.out.println("Accuracy of " + models[j].getClass().getSimpleName() + ": "
+ String.format("%.2f%%", accuracy)
+ "\n---------------------------------");
}
}
}
Файл arff содержит описание, за которым следуют 20 атрибутов данных, за которыми следует метка класса "Да" или "Нет". При выполнении этого на некоторых данных TPR и FPR точно рассчитываются и отображаются в соответствии с каждой матрицей путаницы; Однако область под кривой ROC отображается как "NaN", а кривая представляет собой прямую вертикальную или горизонтальную линию: 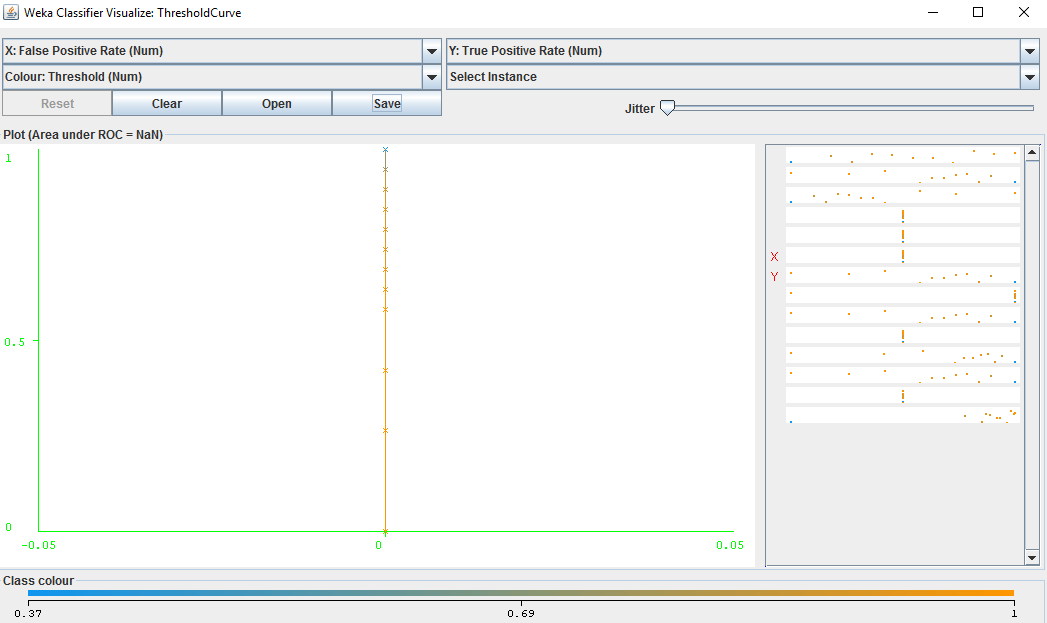
Что я делаю неправильно? Любая помощь приветствуется.
1 ответ
Это был бы комментарий, но я новичок и пока не могу комментировать. Я запустил ваш код из цикла, и с моими данными он работает как шарм. Так что это не проблема с печатью.
Похоже, ваша оценка работает
При выполнении этого на некоторых данных TPR и FPR точно рассчитываются и отображаются в соответствии с каждой матрицей путаницы;
Так вы когда-нибудь пробовали следующую функцию класса Evaluation?
evaluation.areaUnderROC(int classIndex);
Чтобы увидеть, как должна выглядеть кривая ROC?
Ваш класс ярлыков {"yes","no"} или {0,1}? Я не думаю, что это проблема, но не могли бы вы попробовать
Instances result = tc.getCurve(validation.predictions());вместо
Instances result = tc.getCurve(validation.predictions(), classIndex);
Можете ли вы опубликовать некоторые значения матрицы путаницы и TPR/FPR.
ура