Реализация модели нейрона Ижикевича
Я пытаюсь реализовать шипящий нейрон модели Ижикевича. Формула для этого типа нейрона действительно проста:
v [n + 1] = 0,04 * v [n] ^ 2 + 5 * v [n] + 140 - u [n] + I
u [n + 1] = a * (b * v [n] - u [n])
где v - потенциал мембраны, а u - переменная восстановления.
Если значение v превышает 30, оно сбрасывается на c, а u сбрасывается на u + d.
Учитывая такое простое уравнение, я не ожидал бы никаких проблем. Но пока график должен выглядеть 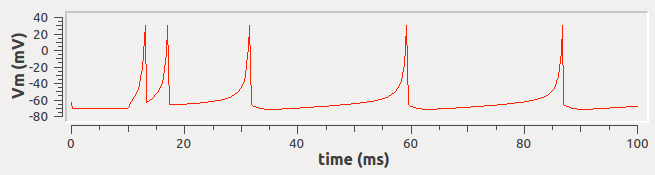 все, что я получаю, это:
все, что я получаю, это: 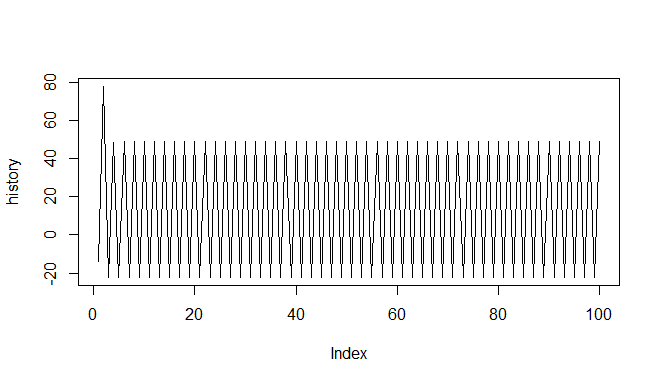
Я в полном недоумении от того, что делаю неправильно, потому что так мало плохого. Я искал другие реализации, но код, который я ищу, всегда где-то спрятан в DLL. Однако я почти уверен, что делаю именно то, что делает код Matlab автора (2). Вот мой полный код R:
v = -70
u = 0
a = 0.02
b = 0.2
c = -65
d = 6
history <- c()
for (i in 1:100) {
if (v >= 30) {
v = c
u = u + d
}
v = 0.04*v^2 + 5*v + 140 - u + 0
u=a*(b*v-u);
history <- c(history, v)
}
plot(history, type = "l")
Всем, кто когда-либо реализовывал модель Ижикевича, что мне не хватает?
Полезные ссылки: (1) http://www.opensourcebrain.org/projects/izhikevichmodel/wiki(2) http://www.izhikevich.org/publications/spikes.pdf
Ответ
Вот и получается, что я неправильно прочитал формулу. Очевидно, v 'означает новый v = v + 0,04*v^2 + 5*v + 140 - u + I. Мои учителя написали бы это как v' = 0,04 * v ^ 2 + 6* v + 140 - u + I Я очень благодарен за вашу помощь, указав мне на это.
1 ответ
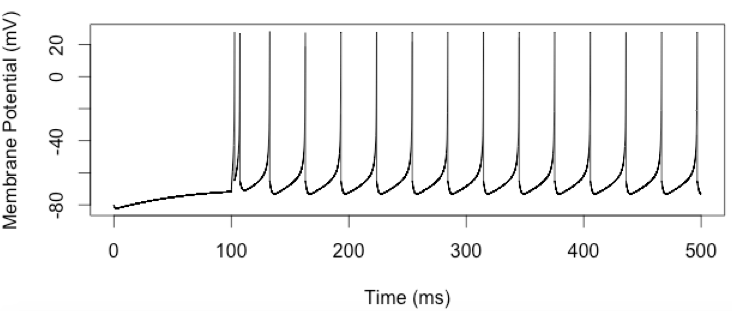
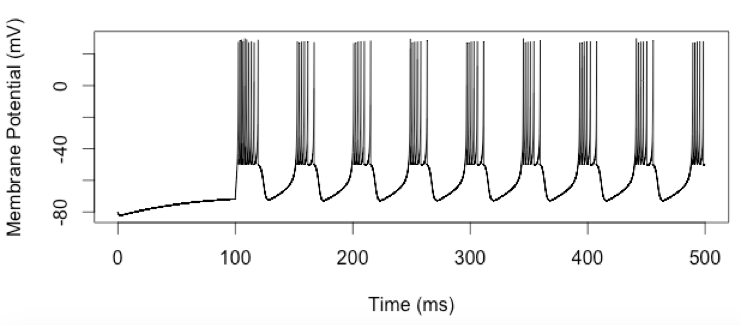
Посмотрите на код, который реализует модель Ижикевича в R ниже. Это приводит к следующим R участкам:
Обычная шипящая клетка:
И код R:
# Simulation parameters
dt = 0.01 # ms
simtime = 500 # ms
t = 0
# Injection current
I = 15
delay = 100 # ms
# Model parameters (RS)
a = 0.02
b = 0.2
c = -65
d = 8
# Params for chattering cell (CH)
# c = -50
# d = 2
# Initial conditions
v = -80 # mv
u = 0
# Input current equation
current = function()
{
if(t >= delay)
{
return(I)
}
return (0)
}
# Model state equations
deltaV = function()
{
return (0.04*v*v+5*v+140-u+current())
}
deltaU = function()
{
return (a*(b*v-u))
}
updateState = function()
{
v <<- v + deltaV()*dt
u <<- u + deltaU()*dt
if(v >= 30)
{
v <<- c
u <<- u + d
}
}
# Simulation code
runsim = function()
{
steps = simtime / dt
resultT = rep(NA, steps)
resultV = rep(NA, steps)
for (i in seq(steps))
{
updateState()
t <<- dt*(i-1)
resultT[i] = t
resultV[i] = v
}
plot(resultT, resultV,
type="l", xlab = "Time (ms)", ylab = "Membrane Potential (mV)")
}
runsim()
Некоторые заметки:
- Я выбрал параметры для ячейки "Regular Spiking (RS)" с сайта Ижикевича. Вы можете выбрать другие параметры из двух верхних правых графиков на этой странице. Раскомментируйте параметры CH, чтобы получить график для ячейки типа "Chattering".
- Как предположили комментаторы, первые два уравнения в вопросе являются неправильно реализованными дифференциальными уравнениями. Правильный способ реализации первого варианта будет выглядеть примерно так: "v[n+1] = v[n] + (0,04*v[n]^2 + 5*v[n] + 140 - u[n] + Я) * дт ". Посмотрите код выше для примера. dt относится к указанной пользователем переменной интеграции временного шага и обычно dt << 1 мс.
- В цикле for в вопросе переменные состояния u и v должны сначала обновляться, а затем проверяться условие.
- Как отмечали другие, источник тока необходим для обоих типов клеток. Я использовал 15 (я считаю, что это пико усилители) с этой страницы на сайте автора (нижнее значение для меня на скриншоте). Я также реализовал задержку для текущего начала (параметр 100 мс).
- Код симуляции должен реализовывать какое-то временное отслеживание, чтобы было легче узнать, когда в результирующем графике возникают пики. Приведенный выше код реализует это и выполняет моделирование в течение 500 мс.