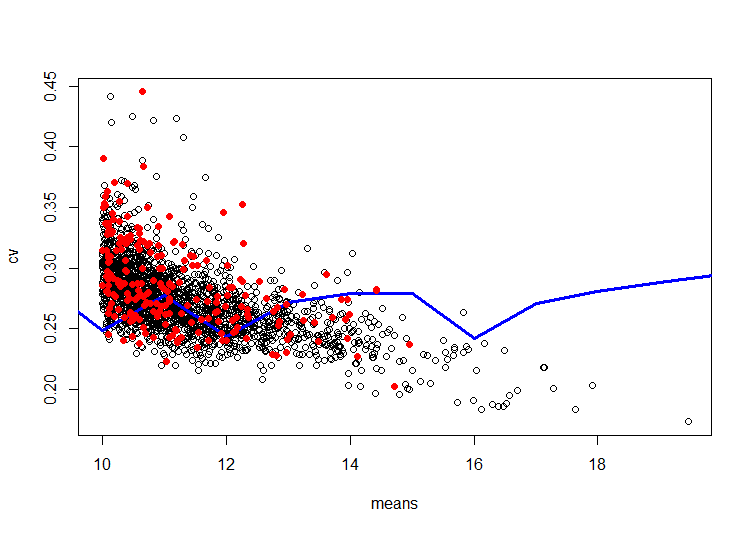
R - кривая лёсса, неправильно выровненная через точки
1 ответ
Решение
Главным образом, мы должны проверить, что predict функция возвращает:
head(predict(afit))
[1] 0.8548271 0.8797704 0.8584954 0.8031563 0.9012096 0.8955874
Это вектор, поэтому, когда мы передаем его lines, R говорит: "Хорошо, вы не указали значение x, поэтому я просто буду использовать индекс для значений x" (попробуйте plot(2:10) чтобы понять, что я имею в виду).
Итак, нам нужно указать матрицу из 2 столбцов, которую нужно передать linesвместо:
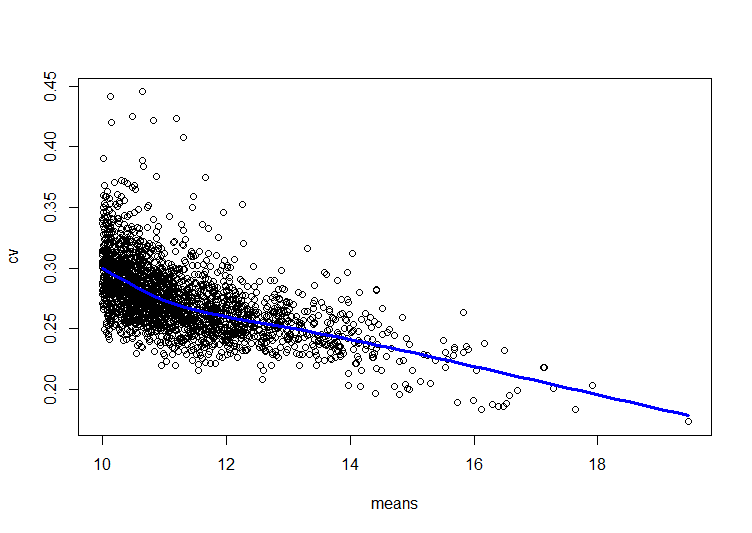
cbind(sort(means), predict(afit, newdata = sort(means)))
должен сделать свое дело. Ваша функция может быть записана как:
FilterByVariance<-function(dat, threshold = 0.90, span = 0.75){
means <- apply(dat,1,mean)
sds <- apply(dat,1,sd)
cv <- sqrt(sds/means)
afit<-loess(cv~means, span = span)
resids<-afit$residuals
# good<-which(resids >= quantile(resids, probs = threshold))
# points above the curve will have a residual > 0
good <- which(resids > 0)
#plots
plot(cv~means)
lines(cbind(sort(means), predict(afit, newdata = sort(means))),
col="blue",lwd=3)
points(means[good],cv[good],col="red",pch=19)
}