Построение апостериорных оценок параметров от нескольких моделей с байесплотом
Я использую отличную библиотеку черчения bayesplot визуализировать апостериорные вероятностные интервалы из моделей, которые я оцениваю с помощью rstanarm, Я хочу графически сравнить рисунки из разных моделей, получив апостериорные интервалы для коэффициентов на одном графике.
Представьте себе, например, что у меня есть 1000 ничьих сзади для трех параметров beta1, beta2, beta3 для двух разных моделей:
# load the plotting library
library(bayesplot)
#> This is bayesplot version 1.6.0
#> - Online documentation and vignettes at mc-stan.org/bayesplot
#> - bayesplot theme set to bayesplot::theme_default()
#> * Does _not_ affect other ggplot2 plots
#> * See ?bayesplot_theme_set for details on theme setting
library(ggplot2)
# generate fake posterior draws from model1
fdata <- matrix(rnorm(1000 * 3), ncol = 3)
colnames(fdata) <- c('beta1', 'beta2', 'beta3')
# fake posterior draws from model 2
fdata2 <- matrix(rnorm(1000 * 3, 1, 2), ncol = 3)
colnames(fdata2) <- c('beta1', 'beta2', 'beta3')
Bayesplot делает фантастические визуализации для отдельных моделей, и это ggplot2 "под капотом", поэтому я могу настроить, как мне будет угодно:
# a nice plot of 1
color_scheme_set("orange")
mcmc_intervals(fdata) + theme_minimal() + ggtitle("Model 1")

# a nice plot of 2
color_scheme_set("blue")
mcmc_intervals(fdata2) + ggtitle("Model 2")

Но чего я хотел бы добиться, так это построить эти две модели вместе на одном графике, так чтобы для каждого коэффициента у меня было два интервала и я мог различить, какой интервал есть какой, сопоставляя цвет с моделью. Однако я не могу понять, как это сделать. Некоторые вещи, которые не работают:
# doesnt work
mcmc_intervals(fdata) + mcmc_intervals(fdata2)
#> Error: Don't know how to add mcmc_intervals(fdata2) to a plot
# appears to pool
mcmc_intervals(list(fdata, fdata2))

Любые идеи о том, как я мог бы сделать это? Или как это сделать вручную, учитывая матрицы задних розыгрышей?
Создано в 2018-10-18 пакетом представлением (v0.2.1)
3 ответа
Точно так же, как ответ также размещен здесь, я расширил код по ссылке от @Manny T (https://github.com/stan-dev/bayesplot/issues/232)
# simulate having posteriors for two different models each with parameters beta[1],..., beta[4]
posterior_1 <- matrix(rnorm(4000), 1000, 4)
posterior_2 <- matrix(rnorm(4000), 1000, 4)
colnames(posterior_1) <- colnames(posterior_2) <- paste0("beta[", 1:4, "]")
# use bayesplot::mcmc_intervals_data() function to get intervals data in format easy to pass to ggplot
library(bayesplot)
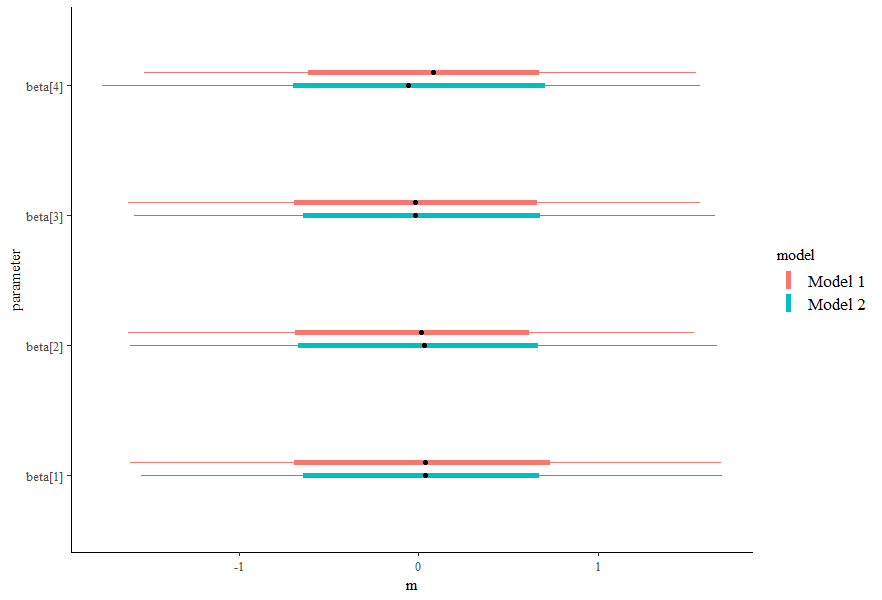
combined <- rbind(mcmc_intervals_data(posterior_1), mcmc_intervals_data(posterior_2))
combined$model <- rep(c("Model 1", "Model 2"), each = ncol(posterior_1))
# make the plot using ggplot
library(ggplot2)
theme_set(bayesplot::theme_default())
pos <- position_nudge(y = ifelse(combined$model == "Model 2", 0, 0.1))
ggplot(combined, aes(x = m, y = parameter, color = model)) +
geom_linerange(aes(xmin = l, xmax = h), position = pos, size=2)+
geom_linerange(aes(xmin = ll, xmax = hh), position = pos)+
geom_point(position = pos, color="black")
Если вы похожи на меня, вам понадобятся 80% и 90% достоверных интервалов (вместо 50%, являющихся внутренними интервалами) и, возможно, вы захотите перевернуть координаты, и давайте добавим пунктирную линию на 0 (модель оценивает без изменений). Вы можете сделать это следующим образом:
# use bayesplot::mcmc_intervals_data() function to get intervals data in format easy to pass to ggplot
library(bayesplot)
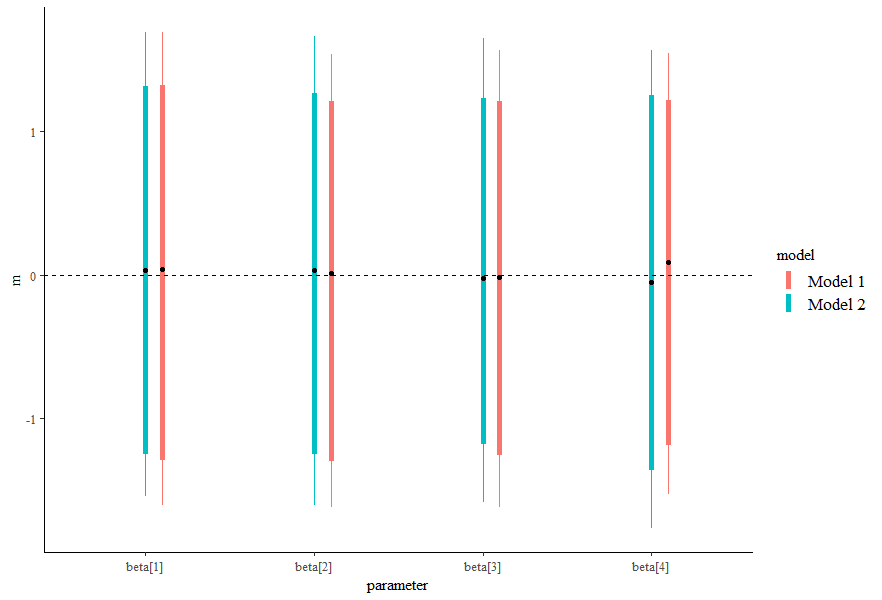
combined <- rbind(mcmc_intervals_data(posterior_1,prob=0.8,prob_outer = 0.9), mcmc_intervals_data(posterior_2,prob=0.8,prob_outer = 0.9))
combined$model <- rep(c("Model 1", "Model 2"), each = ncol(posterior_1))
# make the plot using ggplot
library(ggplot2)
theme_set(bayesplot::theme_default())
pos <- position_nudge(y = ifelse(combined$model == "Model 2", 0, 0.1))
ggplot(combined, aes(x = m, y = parameter, color = model)) +
geom_linerange(aes(xmin = l, xmax = h), position = pos, size=2)+
geom_linerange(aes(xmin = ll, xmax = hh), position = pos)+
geom_point(position = pos, color="black")+
coord_flip()+
geom_vline(xintercept=0,linetype="dashed")
Несколько замечаний по этому последнему. я добавил
prob_outer = 0.9даже если это значение по умолчанию, просто чтобы показать, как вы можете изменить внешние достоверные интервалы. Пунктирная линия создана с помощью
geom_vlineа также
xintercept = здесь вместо
geom_hlineа также
yintercept = из-за
coord_flip(все наоборот). Поэтому, если вы не переворачиваете оси, вам нужно будет сделать наоборот.
Я задал этот вопрос на bayesplotна GitHub и получил ответ (проблема № 232).
Я потратил больше времени, чем хотел бы признать, написав это, так что мог бы также опубликовать это здесь. Вот функция, которая включает в себя предложения выше, которые (на данный момент) работают для объектов модели rstanarm и brms.
compare_posteriors <- function(..., dodge_width = 0.5) {
dots <- rlang::dots_list(..., .named = TRUE)
draws <- lapply(dots, function(x) {
if (class(x)[1] == "stanreg") {
posterior::subset_draws(posterior::as_draws(x$stanfit),
variable = names(fixef(x))
)
} else if (class(x)[1] == "brmsfit") {
brm_draws <- posterior::subset_draws(posterior::as_draws(x$fit),
variable = paste0("b_", rownames(fixef(x)))
)
posterior::variables(brm_draws) <- stringr::str_split(posterior::variables(brm_draws), "_", simplify = T)[, 2]
posterior::rename_variables(brm_draws, `(Intercept)` = Intercept)
} else {
stop(paste0(class(x)[1], " objects not supported."))
}
})
intervals <- lapply(draws, bayesplot::mcmc_intervals_data)
combined <- dplyr::bind_rows(intervals, .id = "model")
ggplot(combined, aes(x = m, y = parameter, color = model, group = model)) +
geom_linerange(aes(xmin = l, xmax = h), size = 2, position = position_dodge(dodge_width)) +
geom_linerange(aes(xmin = ll, xmax = hh), position = position_dodge(dodge_width)) +
geom_point(color = "black", position = position_dodge(dodge_width)) +
geom_vline(xintercept = 0, linetype = "dashed")
}
Применение:
compare_posteriors(mod1, mod2, mod3)