Как избежать перекрытия меток на графике NMDS?
Я попытался избежать наложения меток на графике NMDS с помощью ggrepel пакет. Сначала мой код был таким:
result <- adonis(spiders~Wald, data = env, permutations=1000)
result1 <- metaMDS(spiders, distance = "bray", k = 2)
fit <- envfit(result1, env, perm = 1000)
fig<-plot(result1, type = "none")
points(fig, "sites", pch = as.numeric(env$Wald))
text(fig, "species", font=c(2), cex=c(0.75))
plot(fit, p.max = 0.05, col = "darkgrey", font=c(2), cex=c(0.75))
legend("topright", legend = c("Bestand A", "Bestand B", "Bestand C"),cex =
c(0.75), pch = as.numeric(env$Wald))
И я получил этот сюжет
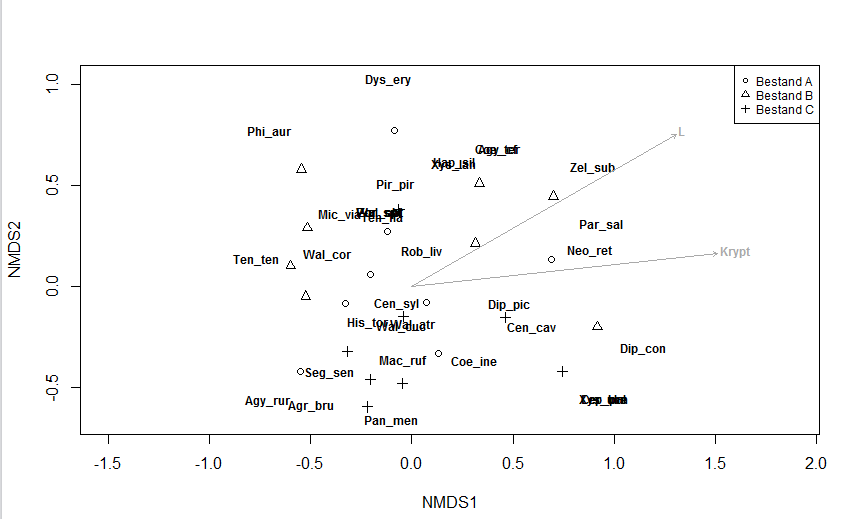
поэтому я немного изменил свой код
fig<-plot(result1, type = "none")
points(fig, "sites", pch = as.numeric(env$Wald))
geom_text_repel(fig, "species", font=c(2), cex=c(0.75))
plot(fit, p.max = 0.05, col = "darkgrey", font=c(2), cex=c(0.75))
но чем я это получил
Ошибка: ggplot2 не знает, как обращаться с данными класса символов.
Я хотел бы предоставить свои данные, чтобы было проще ответить на мой вопрос, но я не знаю, как