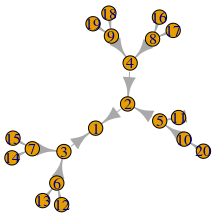
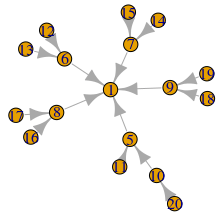
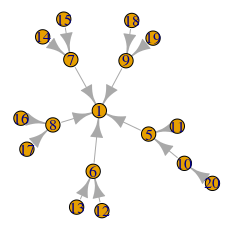
Упростить удаление деревьев
У меня есть нециклический граф, который можно считать деревом. Вот упрощенный пример:
library(tidygraph)
create_tree(20,2, directed = TRUE, mode="in") %>% plot
Пример из реальной жизни может быть немного сложнее, поскольку у меня может быть несколько путей от листьев до корня (все они направлены нециклично).
Я хочу упростить график, удалив промежуточные узлы следующим образом:
К =0
В самом крайнем случае (давайте назовем это "упрощением k=0"), я бы перечислил все листья, заверил, что они связаны с корнем посредством поиска в глубину, а затем удалил все промежуточные соединения, эффективно связывая каждый лист с корнем.
К =-1
Упрощение следующего уровня (скажем, "k=-1") Я хочу начать с узлов, у которых есть хотя бы один дочерний лист, и повторить ту же процедуру. После упрощения все промежуточные узлы будут удалены:
data.frame(from=c(5,6,7,8,9,10,11,12,13,14,15,16,17,18,19,20),
to = c(1,1,1,1,1, 5, 5, 6, 6, 7, 7, 8, 8, 9, 9,10)) %>%
as_tbl_graph() %>% plot
К =-2
Следующий шаг упрощения не будет иметь смысла для этого графа, потому что никакие ребра не будут изменены и никакие узлы не будут удалены.
Как мне написать код, используя igraph/tidygraph в R?
1 ответ
Наиболее важной частью решения является возможность перечисления узлов от листьев до корня, эффективно измеряя "расстояние до ближайшего листа"
Используя тот же пример, что и выше, давайте добавим имена узлов (create_tree() не делает узлов, по какой-то странной причине):
library(tidygraph)
graph <- create_tree(20,2, directed = TRUE, mode="in") %>%
activate(nodes) %>% mutate(name=1:n())
Нам понадобится вспомогательная функция, которая сможет измерять "расстояние до листа":
make_levels <- function(grdf){
i <- 0
repeate <- TRUE
# create helper column
grdf <- grdf %>%
mutate(leaf = node_is_leaf(),
level=ifelse(leaf, i, NA))
while(repeate){
i <- i + 1
index <- grdf %>% activate(edges) %>%
mutate(from_leaf=.N()$leaf[from]) %>%
as_tibble() %>% filter(from_leaf) %>% pull(to)
grdf <- grdf %>% activate(nodes) %>%
mutate(leaf = 1:n() %in% index,
level=ifelse(leaf & is.na(level), i, level))
repeate <- grdf %>% activate(nodes) %>%
as_tibble() %>% pull(level) %>% is.na() %>% any()
}
# remove helper column
grdf %>% activate(nodes) %>% select(-leaf)
}
После этого решение (для k=-1 выше) должно быть легко:
graph %>% make_levels() %>% activate(edges) %>%
reroute(from = from, to = 1, subset=(to %in% which(.N()$level==2))) %>%
activate(nodes) %>% filter(level!=2) %>% plot()
Который производит: