Растрируйте изображения ggplot в R для tikzdevice
Я использую R для анализа данных, ggplot для создания графиков, tikzDevice для их печати и, наконец, латекс для создания отчета. Проблема в том, что большие графики с большим количеством точек не работают из-за ограничения памяти латекса. Я нашел здесь https://github.com/yihui/tikzDevice/issues/103 решение, которое растеризует график перед печатью файла tikz, что позволяет печатать точки и текст по отдельности.
require(png)
require(ggplot2)
require(tikzDevice)
## generate data
n=1000000; x=rnorm(n); y=rnorm(n)
## first try primitive
tikz("test.tex",standAlone=TRUE)
plot(x,y)
dev.off()
## fails due to memory
system("pdflatex test.tex")
## rasterise points first
png("inner.png",width=8,height=6,units="in",res=300,bg="transparent")
par(mar=c(0,0,0,0))
plot.new(); plot.window(range(x), range(y))
usr <- par("usr")
points(x,y)
dev.off()
# create tikz file with rasterised points
im <- readPNG("inner.png",native=TRUE)
tikz("test.tex",7,6,standAlone=TRUE)
plot.new()
plot.window(usr[1:2],usr[3:4],xaxs="i",yaxs="i")
rasterImage(im, usr[1],usr[3],usr[2],usr[4])
axis(1); axis(2); box(); title(xlab="x",ylab="y")
dev.off()
## this works
system("pdflatex test.tex")
## now with ggplot
p <- ggplot(data.frame(x=x, y=y), aes(x=x, y=y)) + geom_point()
## what here?
В этом примере первый pdflatex выходит из строя. Второе успешно из-за растеризации.
Как я могу применить это с помощью ggplot?
2 ответа
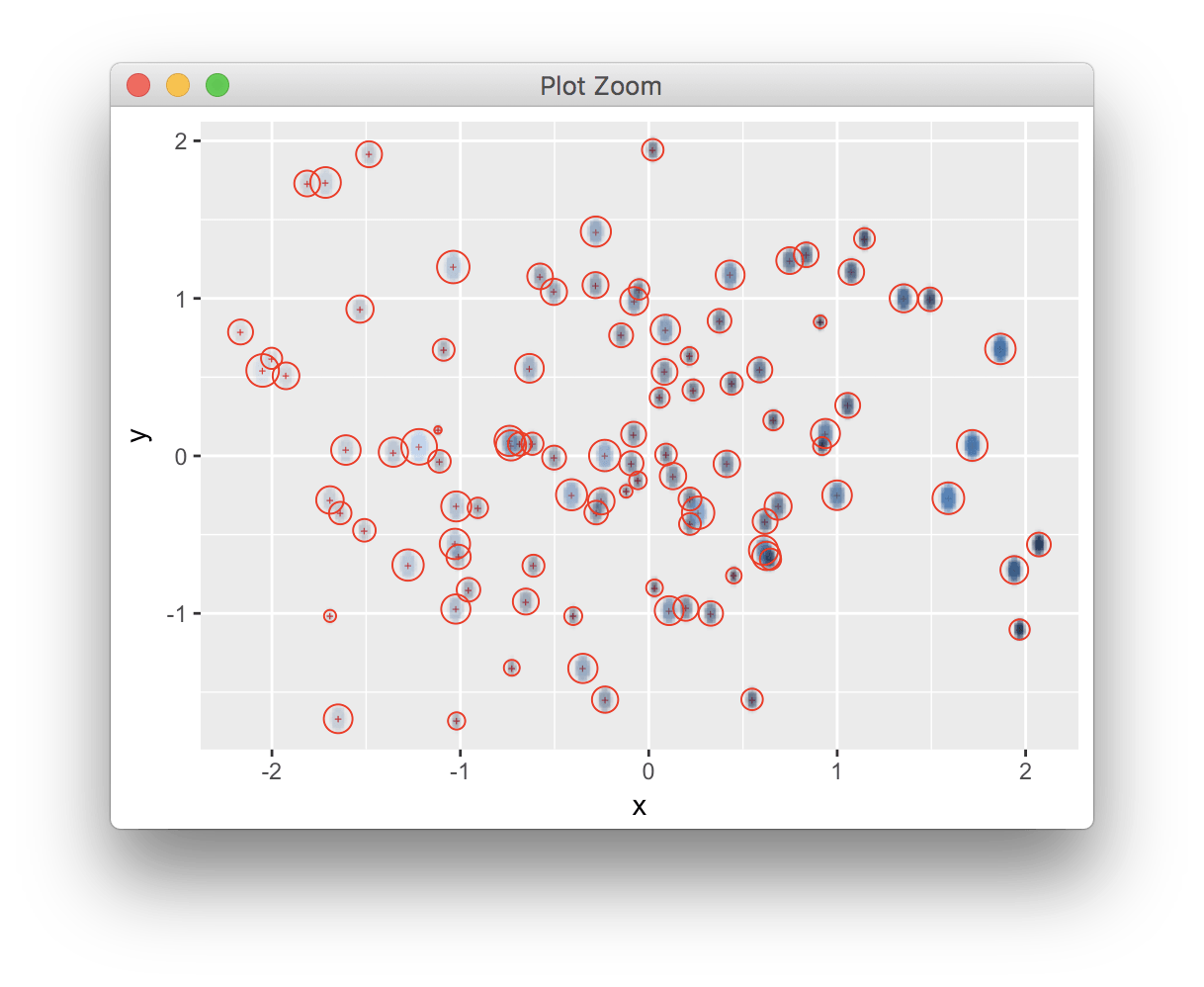
Вот доказательство принципа, чтобы проиллюстрировать шаги, которые будут вовлечены. Как указано в комментариях, это не рекомендуется и не практично, но может стать основой для реализации более низкого уровня.
require(png)
require(ggplot2)
require(tikzDevice)
n=100;
d <- data.frame(x=rnorm(n), y=rnorm(n), z=rnorm(n))
p <- ggplot(d, aes(x=x, y=y, colour=z, size=z, alpha=x)) + geom_point()
## draw the layer by itself on a png file
library(grid)
g <- ggplotGrob(p)
# grid.newpage()
gg <- g$grobs[[6]]$children[[3]]
gg$vp <- viewport() # don't ask me
tmp <- tempfile(fileext = "png")
png(tmp, width=10, height=4, bg = "transparent", res = 30, units = "in")
grid.draw(gg)
dev.off()
## import it as a raster layer
rl <- readPNG(tmp, native = TRUE)
unlink(tmp)
## add it to a plot - note that the positions match,
## but the size can be off unless one ensures that the panel has the same size and aspect ratio
ggplot(d, aes(x=x, y=y)) + geom_point(shape="+", colour="red") +
annotation_custom(rasterGrob(rl, width = unit(1,"npc"), height=unit(1,"npc"))) +
geom_point(aes(size=z), shape=1, colour="red", show.legend = FALSE)
## to illustrate the practical use, we use a blank layer to train the scales
## and set the panel size to match the png file
pf <- ggplot(d, aes(x=x, y=y)) + geom_blank() +
annotation_custom(rasterGrob(rl, width = unit(1,"npc"), height=unit(1,"npc"), interpolate = FALSE))
tikz("test.tex", standAlone=TRUE)
grid.draw(egg::set_panel_size(pf, width=unit(10, "cm"), height=unit(4, "cm")))
dev.off()
system("lualatex test.tex")
system("open test.pdf")
мы можем увеличить масштаб и проверить, что текст является векторным, в то время как слой (здесь с низким разрешением для демонстрации) растровый.
Хорошо, я напишу это здесь, потому что это было слишком большим для поля для комментариев. Вместо добавления растеризованных точек к новому графику с новыми масштабами вы можете фактически заменить исходный гроб растеризованным гробом на g$grobs[[6]]$children[[3]] <- rasterGrob(rl), Проблема в том, что он не масштабируется, поэтому вы должны знать размер конечного изображения раньше. Тогда вы можете подать в суд на это:
rasterise <- function(ggp,
width = 6,
height = 3,
res.raster = 300,
raster.id= c(4,3),
file = ""){
## RASTERISE
require(grid)
require(png)
## draw the layer by itself on a png file
gb <- ggplot_build(ggp)
gt <- ggplot_gtable(gb)
## calculate widths
h <- as.numeric(convertUnit(sum(gt$heights), unitTo="in"))
w <- as.numeric(convertUnit(sum(gt$widths) , unitTo="in"))
w.raster <- width-w
h.raster <- height-h
## print points as png
grid.newpage()
gg <- gt$grobs[[raster.id[1]]]$children[[raster.id[2]]]
gg$vp <- viewport() # don't ask me
tmp <- tempfile(fileext = "png")
png(tmp, width=w.raster, height=h.raster, bg = "transparent", res = res.raster, units = "in")
grid.draw(gg)
dev.off()
## import it as a raster layer
points <- readPNG(tmp, native = TRUE)
points <- rasterGrob(points, width = w.raster, height = h.raster, default.units = "in")
unlink(tmp)
## ADD TO PLOT
gt$grobs[[raster.id[1]]]$children[[raster.id[2]]] <- points
## PLOT TMP
### HERE YOU CAN ONLY PRINT IT IN THIS DIMENSIONS!
pdf(file, width = width, height = height)
grid.draw(gt)
dev.off()
}
А затем использовать его с
data <- data.frame(x = rnorm(1000), y = rnorm(1000))
plot <- ggplot(data, aes(x = x, y = y)) +
geom_point() +
annotate("text", x = 2, y = 2, label = "annotation")
rasterise(ggp = plot,
width = 6,
height = 3,
res.raster = 10,
raster.id = c(4,2),
file = "~/test.pdf")
Проблема остается идентификатором гроба, который вы хотите растеризовать. Я не нашел хороший способ найти правильный автоматически. Это зависит от того, какие слои вы добавляете на график.