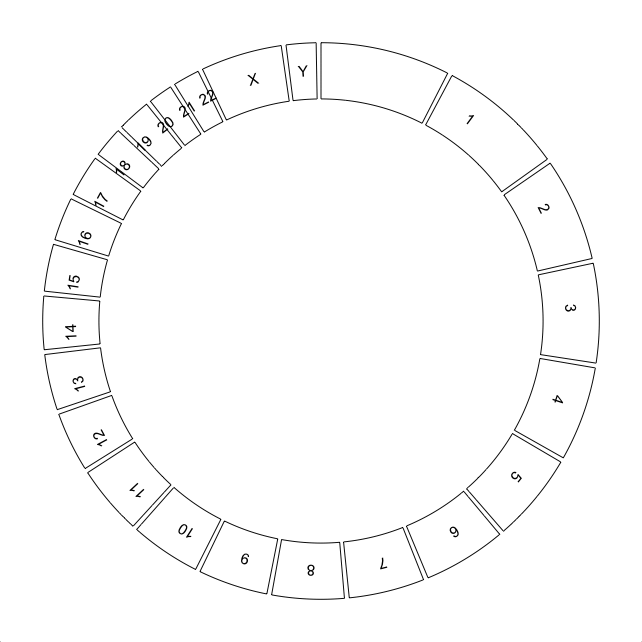
Circlize: текст смещен по сравнению с нижележащими дорожками
Хорошо, я пытаюсь попасть в circlize пакет для некоторых геномных сюжетов, которые я хочу сделать. Однако у меня проблемы с смещенным / смещенным текстом (и другими вещами, которые я пытаюсь добавить в треки). Вот некоторый (надеюсь, воспроизводимый) код:
# Get chromosome starts and ends (GRCh38)
chroms <- read.csv(sep = " ", text = "chr start end
1 0 248956422
2 248956423 491149952
3 491149953 689445512
4 689445513 879660068
5 879660069 1061198328
6 1061198329 1232004308
7 1232004309 1391350282
8 1391350283 1536488919
9 1536488920 1674883637
10 1674883638 1808681060
11 1808681061 1943767683
12 1943767684 2077042993
13 2077042994 2191407322
14 2191407323 2298451041
15 2298451042 2400442231
16 2400442232 2490780577
17 2490780578 2574038019
18 2574038020 2654411305
19 2654411306 2713028922
20 2713028923 2777473090
21 2777473091 2824183074
22 2824183075 2875001543
X 2875001544 3031042439
Y 3031042440 3088269855")
chroms$chr <- factor(chroms$chr, levels = chroms$chr)
# Create a circos plot
library("circlize")
circos.clear()
circos.par("start.degree" = 90)
circos.genomicInitialize(chroms, plotType = NULL)
circos.genomicTrack(chroms, ylim = c(0, 1))
circos.genomicText(chroms[c(2,3)], chroms, y = 0.5, labels.column = "chr")
Когда я запускаю это, я получаю следующий рисунок и предупреждающее сообщение: Note: 23 points are out of plotting region in sector 'Y', track '1'.
Как видите, все текстовые метки не совпадают с основными дорожками. Я тщетно пытался выяснить, с чем связана эта проблема. Я задавался вопросом, были ли мои входные данные как-то неправильно отформатированы, но я также не могу понять это. Например, сама базовая дорожка кажется правильной.
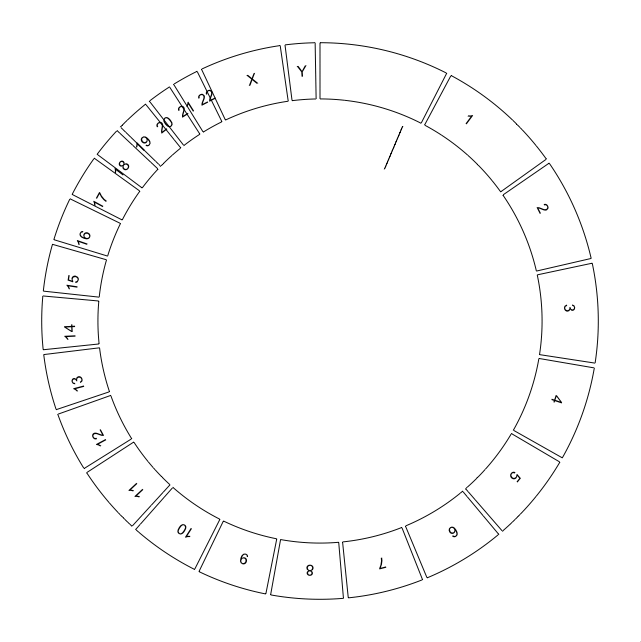
Я также пытался представить конкретные геномные позиции, которые меня интересуют, но у меня возникают похожие проблемы. Например, этот единственный нуклеотидный вариант:
# Add a single genomic position as a line in a new track
regions <- data.frame(start = 1, end = 1)
values <- data.frame(value = 1)
circos.track(ylim = c(0, 1), bg.border = NA)
circos.genomicRect(regions, values)
С предупреждением о Note: 5 points are out of plotting region in sector 'Y', track '2'., Мало того, что SNV неправильно позиционируется в связи с проблемным текстом, который я сгенерировал выше, но он также ошибочно позиционируется относительно основного трека: он должен присутствовать в позиции 1т.е. в самом начале всего сюжета у хромосомы 1, но это не так. Он также неправильно расположен относительно текста, вставленного выше.
Я действительно не знаю, что здесь не так, я не понимаю предупреждающих сообщений (поиск в Google не помог), и я чувствую, что, должно быть, я пропустил довольно большую основную теорию о том, как circlize работает, но я попытался следовать "circlize_book", и я все еще получаю эти ошибки. Я также попытался вручную ввести области генома, используя функции не-генома *(), но получил те же результаты. Мои входные данные могут быть как-то искажены, но я не вижу как. У кого-нибудь есть идеи, что я сделал не так?