Подгонка связанных логнормальных данных в Python
У меня есть диапазон данных о распределении частиц по размерам, упорядоченный по доле объемной доли, например:;
size %
6.68 0.05
9.92 1.15
etc.
Мне нужно подогнать эти данные к логнормальному дистрибутиву, что я планировал сделать с помощью Python stats.lognorm.fit функция, но это, кажется, ожидает ввода в виде массива переменных, а не двоичных данных, судя по тому, что я прочитал.
Я планировал использовать цикл для перебора данных и .extend каждая запись размера в массив-заполнитель требуется количество раз, необходимое для создания массива со списком переменных, соответствующих объединенным данным.
Это кажется действительно уродливым и неэффективным, и такие вещи, вероятно, есть простой способ сделать. Есть ли способ ввода двоичных данных в stats.lognorm.fit функционировать?
1 ответ
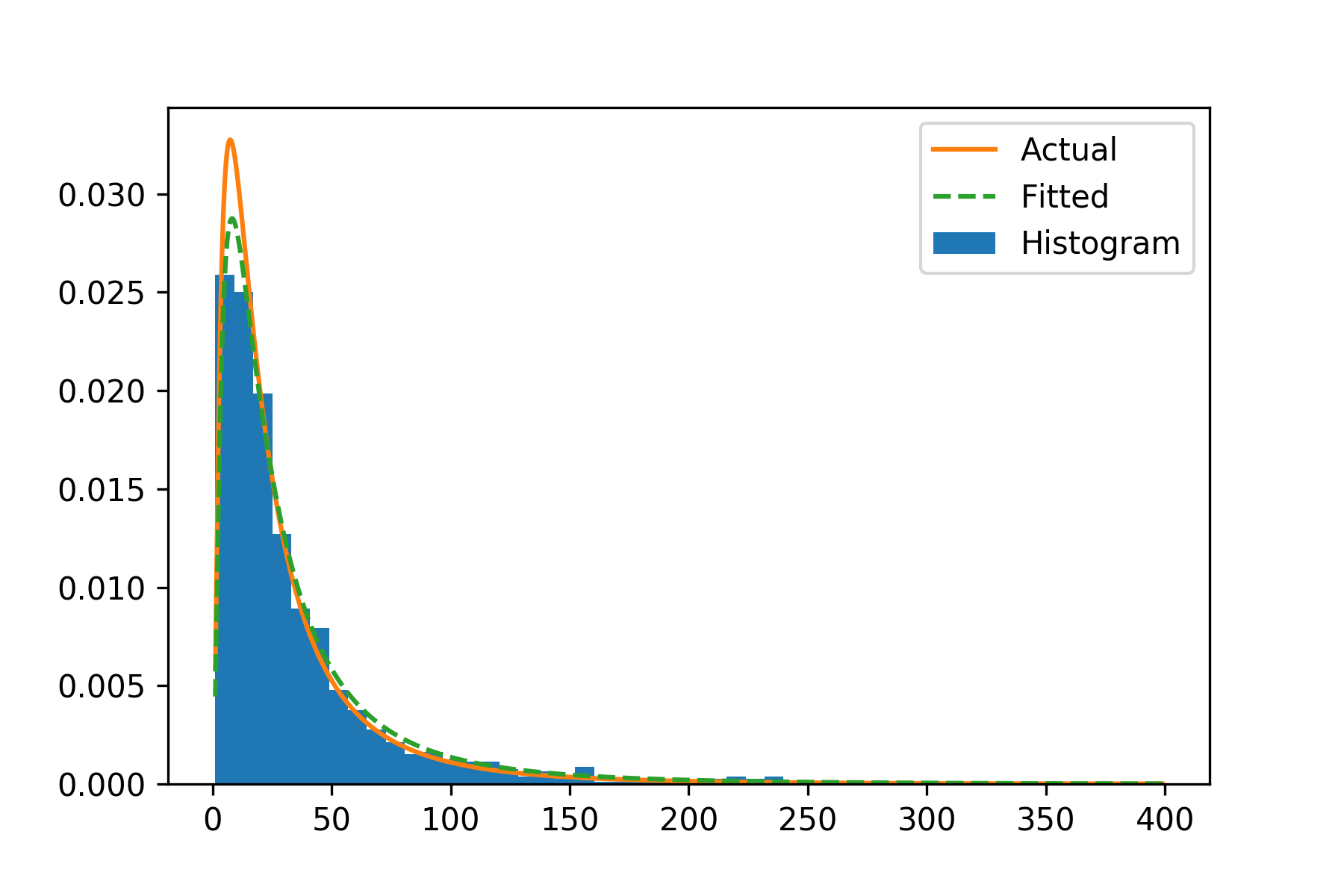
Я предполагаю, что один из возможных обходных путей - это вручную подогнать pdf к данным вашего бина, предполагая, что значения x - это середина каждого интервала, а значения y - соответствующая частота бина. А затем подгоните кривую на основе значений х и у, используя scipy.optimize.curve_fit, Я думаю, что точность результатов будет зависеть от количества бинов у вас есть. Пример показан ниже:
import matplotlib.pyplot as plt
from scipy.optimize import curve_fit
import numpy as np
def pdf(x, mu, sigma):
"""pdf of lognormal distribution"""
return (np.exp(-(np.log(x) - mu)**2 / (2 * sigma**2)) / (x * sigma * np.sqrt(2 * np.pi)))
mu, sigma = 3., 1. # actual parameter value
data = np.random.lognormal(mu, sigma, size=1000) # data generation
h = plt.hist(data, bins=30, normed = True)
y = h[0] # frequencies for each bin, this is y value to fit
xs = h[1] # boundaries for each bin
delta = xs[1] - xs[0] # width of bins
x = xs[:-1] + delta / # midpoints of bins, this is x value to fit
popt, pcov = curve_fit(pdf, x, y, p0=[1, 1]) # data fitting, popt contains the fitted parameters
print(popt)
# [ 3.13048122 1.01360758] fitting results
fig, ax = plt.subplots()
ax.hist(data, bins=30, normed=True, align='mid', label='Histogram')
xr = np.linspace(min(xs), max(xs), 10000)
yr = pdf(xr, mu, sigma)
yf = pdf(xr, *popt)
ax.plot(xr, yr, label="Actual")
ax.plot(xr, yf, linestyle = 'dashed', label="Fitted")
ax.legend()