Составить генеалогию в R
d = data.frame(
offspring = c("G2I1", "G2I2", "G2I3", "G3I1", "G3I2", "G3I3", "G3I4", "G4I1", "G4I2", "G4I3", "G4I4", "G5I1", "G5I2", "G5I3" ),
parent1 = c("G1I1", "G1I2", "G1I1", "G2I1", "G2I3", "G2I1", "G2I3", "G3I2", "G3I2", "G3I1", "G3I4", "G4I3", "G4I3", "G4I1" ),
parent2 = c("G1I3", "G1I2", "G1I2", "G2I2", "G2I2", "G2I2", "G2I3", "G3I4", "G3I1", "G3I2", "G3I4", "G4I1", "G4I1", "G4I2" )
)
print(d)
offspring parent1 parent2
1 G2I1 G1I1 G1I3 # generation 2
2 G2I2 G1I2 G1I2 # generation 2
3 G2I3 G1I1 G1I2 # generation 2
4 G3I1 G2I1 G2I2 # generation 3
5 G3I2 G2I3 G2I2 # generation 3
6 G3I3 G2I1 G2I2 # generation 3
7 G3I4 G2I3 G2I3 # generation 3
8 G4I1 G3I2 G3I4 # generation 4
9 G4I2 G3I2 G3I1 # generation 4
10 G4I3 G3I1 G3I2 # generation 4
11 G4I4 G3I4 G3I4 # generation 4
12 G5I1 G4I3 G4I1 # generation 5
13 G5I2 G4I3 G4I1 # generation 5
14 G5I3 G4I1 G4I2 # generation 5
Представление данных
Эти данные представляют генеалогию. Каждая строка указывает на потомство и двух его родителей. Я звоню им parent1 а также parent2 потому что они гермафродиты. Кроме того, они могут клонировать себя! Поколения не пересекаются, а это означает, что все родители потомков поколения n родились в поколении n-1,
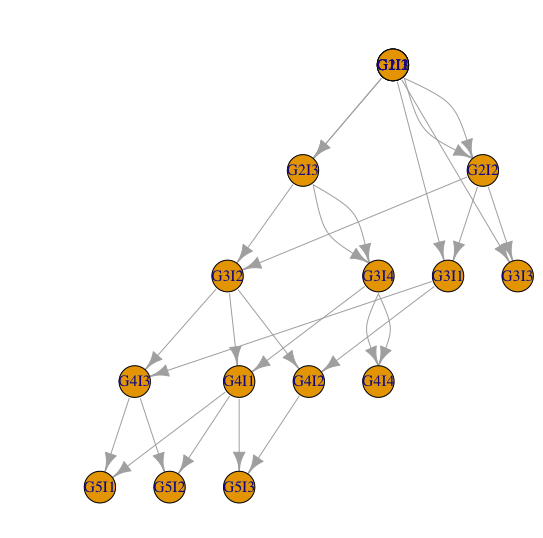
Давайте рассмотрим пример. Индивидуальный G3I4 родился в поколении 3 (G3) и является индивидуальным индексом 4 этого поколения (I4; индекс это просто идентификатор). Этот человек является родителем человека G4I1 и отдельных G4I4, По факту, G3I4 единственный родитель G4I4 как она клонировала себя.
Вопрос
Как я могу построить эту генеалогию в R?
Связанный пост
Пост Как построить семейное древо в R очень связан, но я не смог применить его к своим данным. Первый вопрос использует igraph с которым я не очень знаком. Но я не смог ничего хорошего
d = tibble(
offspring = c("G2I1", "G2I2", "G2I3", "G3I1", "G3I2", "G3I3", "G3I4", "G4I1", "G4I2", "G4I3", "G4I4", "G5I1", "G5I2", "G5I3" ),
parent1 = c("G1I1", "G1I2", "G1I1", "G2I1", "G2I3", "G2I1", "G2I3", "G3I2", "G3I2", "G3I1", "G3I4", "G4I3", "G4I3", "G4I1" ),
parent2 = c("G1I3", "G1I2", "G1I2", "G2I2", "G2I2", "G2I2", "G2I3", "G3I4", "G3I1", "G3I2", "G3I4", "G4I1", "G4I1", "G4I2" )
)
d2 = data.frame(from=c(d$parent1,d$parent2), to=rep(d$offspring,2))
g=graph_from_data_frame(d2)
co=layout.reingold.tilford(g, flip.y=T)
plot(g,layout=co)
но некоторые люди, которые не оставляют потомства, отсутствуют на графике.
Второй ответ использует kinship2, Насколько я понимаю, kinship2 не может иметь дело с бесполым размножением.
1 ответ
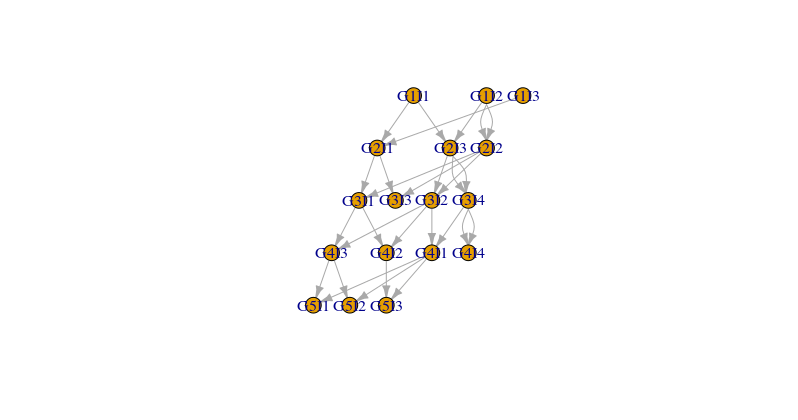
Единственное, что я вижу неправильно - это перекрывающиеся узлы в G1. Имея больше информации, я с удовольствием подправлю вывод по мере необходимости.
library(igraph)
d = data.frame(
offspring = c("G2I1", "G2I2", "G2I3", "G3I1", "G3I2", "G3I3", "G3I4", "G4I1", "G4I2", "G4I3", "G4I4", "G5I1", "G5I2", "G5I3" ),
parent1 = c("G1I1", "G1I2", "G1I1", "G2I1", "G2I3", "G2I1", "G2I3", "G3I2", "G3I2", "G3I1", "G3I4", "G4I3", "G4I3", "G4I1" ),
parent2 = c("G1I3", "G1I2", "G1I2", "G2I2", "G2I2", "G2I2", "G2I3", "G3I4", "G3I1", "G3I2", "G3I4", "G4I1", "G4I1", "G4I2" ),
stringsAsFactors = F
)
d2 = data.frame(from=c(d$parent1,d$parent2), to=rep(d$offspring,2))
g=graph_from_data_frame(d2)
#co=layout.reingold.tilford(g, flip.y=T)
co1 <- layout_as_tree(g, root = which(grepl("G1", V(g)$name)))
#plot(g,layout=co, edge.arrow.size=0.5)
plot(g,layout=co1, edge.arrow.size=0.25)