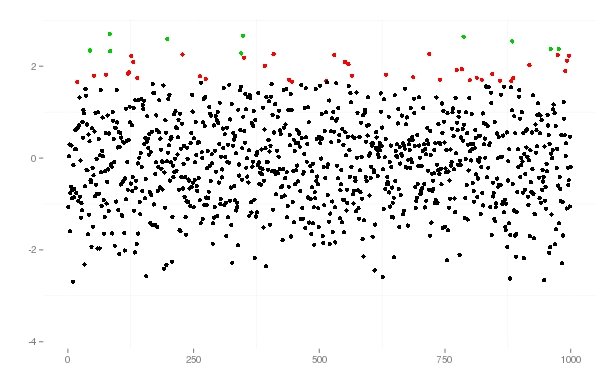
Цветовые кодовые точки на основе процентиля в ggplot
У меня есть несколько очень больших файлов, которые содержат геномную позицию (положение) и соответствующую популяционную генетическую статистику (значение). Я успешно нанес на график эти значения и хотел бы закрасить верхние 5% (синий) и 1% (красный) значений. Мне интересно, есть ли простой способ сделать это в R.
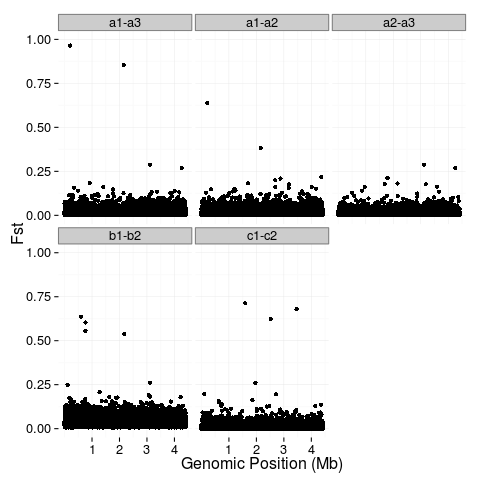
Я исследовал написание функции, которая определяет квантили, однако многие из них оказываются не уникальными и, таким образом, вызывают сбой функции. Я также изучил stat_quantile, но с успехом использовал его для построения линии, обозначающей 95% и 99% (а некоторые линии были диагоналями, которые не имели для меня никакого смысла). (Извините, я новичок в Р.)
Любая помощь приветствуется.
Вот мой код: (файлы очень большие)
########Combine data from multiple files
fst <- rbind(data.frame(key="a1-a3", position=a1.3$V2, value=a1.3$V3), data.frame(key="a1-a2", position=a1.2$V2, value=a1.2$V3), data.frame(key="a2-a3", position=a2.3$V2, value=a2.3$V3), data.frame(key="b1-b2", position=b1.2$V2, value=b1.2$V3), data.frame(key="c1-c2", position=c1.2$V2, value=c1.2$V3))
########the plot
theme_set(theme_bw(base_size = 16))
p1 <- ggplot(fst, aes(x=position, y=value)) +
geom_point() +
facet_wrap(~key) +
ylab("Fst") +
xlab("Genomic Position (Mb)") +
scale_x_continuous(breaks=c(1e+06, 2e+06, 3e+06, 4e+06), labels=c("1", "2", "3", "4")) +
scale_y_continuous(limits=c(0,1)) +
theme(plot.background = element_blank(),
panel.background = element_blank(),
panel.border = element_blank(),
legend.position="none",
legend.title = element_blank()
)
p1
3 ответа
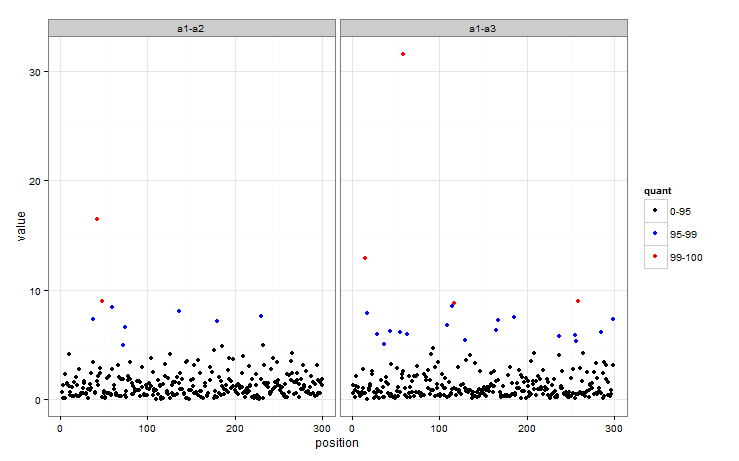
Вот как я бы подошел к этому - в основном создавая фактор, определяющий, в какую группу входит каждое наблюдение, а затем отображал colour к этому фактору.
Во-первых, некоторые данные для работы!
dat <- data.frame(key = c("a1-a3", "a1-a2"), position = 1:100, value = rlnorm(200, 0, 1))
#Get quantiles
quants <- quantile(dat$value, c(0.95, 0.99))
Есть много способов получить фактор, определяющий, к какой группе относится каждое наблюдение, вот один:
dat$quant <- with(dat, factor(ifelse(value < quants[1], 0,
ifelse(value < quants[2], 1, 2))))
Так quant теперь указывает, относится ли наблюдение к группе 95-99 или 99+. Цвет точек на графике может быть легко сопоставлен с quant,
ggplot(dat, aes(position, value)) + geom_point(aes(colour = quant)) + facet_wrap(~key) +
scale_colour_manual(values = c("black", "blue", "red"),
labels = c("0-95", "95-99", "99-100")) + theme_bw()
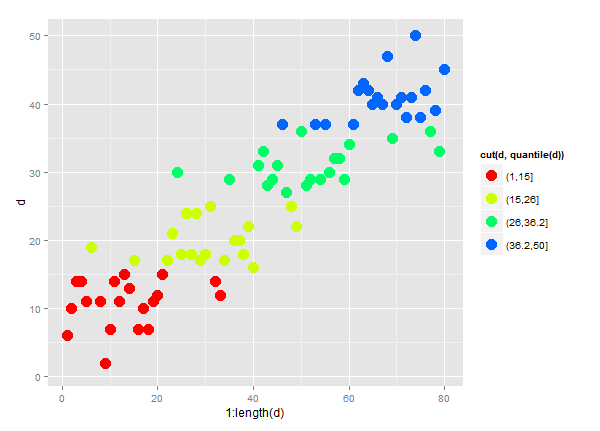
Вы можете достичь этого немного более элегантно, включив quantile а также cut в aes цветовое выражение. Например col=cut(d,quantile(d)) в этом примере:
d = as.vector(round(abs(10 * sapply(1:4, function(n)rnorm(20, mean=n, sd=.6)))))
ggplot(data=NULL, aes(x=1:length(d), y=d, col=cut(d,quantile(d)))) +
geom_point(size=5) + scale_colour_manual(values=rainbow(5))
Я также сделал полезный рабочий процесс для красивых ярлыков с надписями, которые кому-то могут пригодиться.
Я не уверен, что это то, что вы ищете, но, возможно, это поможет:
# a little function which returns factors with three levels, normal, 95% and 99%
qfun <- function(x, qant_1=0.95, qant_2=0.99){
q <- sort(c(quantile(x, qant_1), quantile(x, qant_2)))
factor(cut(x, breaks = c(min(x), q[1], q[2], max(x))))
}
df <- data.frame(samp=rnorm(1000))
ggplot(df, aes(x=1:1000, y=df$samp)) + geom_point(colour=qfun(df$samp))+
xlab("")+ylab("")+
theme(plot.background = element_blank(),
panel.background = element_blank(),
panel.border = element_blank(),
legend.position="none",
legend.title = element_blank())
в результате я получил