seqinr dotplot - изменить ось
У меня есть наборы данных: seq1 и seq2 (последовательности ДНК). Я хотел сделать график данных, сравнивая две последовательности и помещая точку там, где две последовательности совпадают. Я смог сделать это, используя точечную диаграмму seqinr, но я не могу перечислить последовательности на оси, чтобы вы могли увидеть, какие точки совпадают. По сути, я хочу заменить цифры на буквы последовательности.
Есть какой-либо способ сделать это? Может через ggplot2?

Это мои последовательности:
seq1 <- c("G","C","T","A","G","T","C","A","G","A","T","C","T","G","A","C","G","C","T","A")
seq2 <- c("G","A","T","G","G","T","C","A","C","A","T","C","T","G","C","C","G","C")
И вот как я сгенерировал этот график:
dotPlot(seq1, seq2, main = "Dot plot of 2 different sequences
\nwsize = 4, wstep = 1, nmatch = 3", wsize = 4, wstep = 1, nmatch = 3)
2 ответа
Как насчет модифицированной версии (называемой "быстрый и грязный хак") функции dotplot? Сначала скопируйте и вставьте следующий код в R:
dotplot<-function (seq1, seq2, wsize = 1, wstep = 1, nmatch = 1, cols = c("white",
"black"), xlab = deparse(substitute(seq1)), ylab = deparse(substitute(seq2)), type=2,
...) {
cat("This is a modification of the function dotPlot from package seqinr.\n")
require(seqinr)
if (nchar(seq1[1]) > 1)
stop("seq1 should be provided as a vector of single chars")
if (nchar(seq2[1]) > 1)
stop("seq2 should be provided as a vector of single chars")
if (wsize < 1)
stop("non allowed value for wsize")
if (wstep < 1)
stop("non allowed value for wstep")
if (nmatch < 1)
stop("non allowed value for nmatch")
if (nmatch > wsize)
stop("nmatch > wsize is not allowed")
mkwin <- function(seq, wsize, wstep) {
sapply(seq(from = 1, to = length(seq) - wsize + 1, by = wstep),
function(i) c2s(seq[i:(i + wsize - 1)]))
}
wseq1 <- mkwin(seq1, wsize, wstep)
wseq2 <- mkwin(seq2, wsize, wstep)
if (nmatch == wsize) {
xy <- outer(wseq1, wseq2, "==")
}
else {
"%==%" <- function(x, y) colSums(sapply(x, s2c) == sapply(y,
s2c)) >= nmatch
xy <- outer(wseq1, wseq2, "%==%")
}
if(type==1) {
image(x = seq(from = 1, to = length(seq1), length = length(wseq1)),
y = seq(from = 1, to = length(seq2), length = length(wseq2)),
z = xy, col = col, xlab = xlab, ylab = ylab, axes=F, ...)
box()
}
colnames(xy)<-wseq2
rownames(xy)<-wseq1
xy2<-matrix(nrow=length(seq1), ncol=length(seq2), data=FALSE)
rownames(xy2)<-seq1
colnames(xy2)<-seq2
ind<-which(xy, arr.ind=T)
xy2[ind]<-TRUE
ind<-data.frame(ind, row.names=NULL)
res<-data.frame(row=c(), col=c())
for(i in 1:nrow(ind)) {
DF<-data.frame(row=seq(from=ind$row[i], to=ind$row[i]+wsize-1),
col=seq(from=ind$col[i], to=ind$col[i]+wsize-1))
res<-rbind(res, DF)
}
xy2[as.matrix(res)]<-TRUE
if(type==2) {
image(x = seq(from = 1, to = length(seq1)), y = seq(from = 1, to = length(seq2)), z = xy2, col = cols, xlab = xlab, ylab = ylab, axes=F, ...)
box()
axis(side=1, at=1:length(seq1), labels=seq1)
axis(side=2, at=1:length(seq2), labels=seq2, las=1)
}
out<-list(type1=xy, type2=xy2)
return(out)
}
Затем запустим приведенный вами пример:
seq1 <- c("G","C","T","A","G","T","C","A","G","A","T","C","T","G","A","C","G","C","T","A")
seq2 <- c("G","A","T","G","G","T","C","A","C","A","T","C","T","G","C","C","G","C")
xy<-dotplot(seq1, seq2, wsize = 4, wstep = 1, nmatch = 3)
следует изготовить следующий участок:
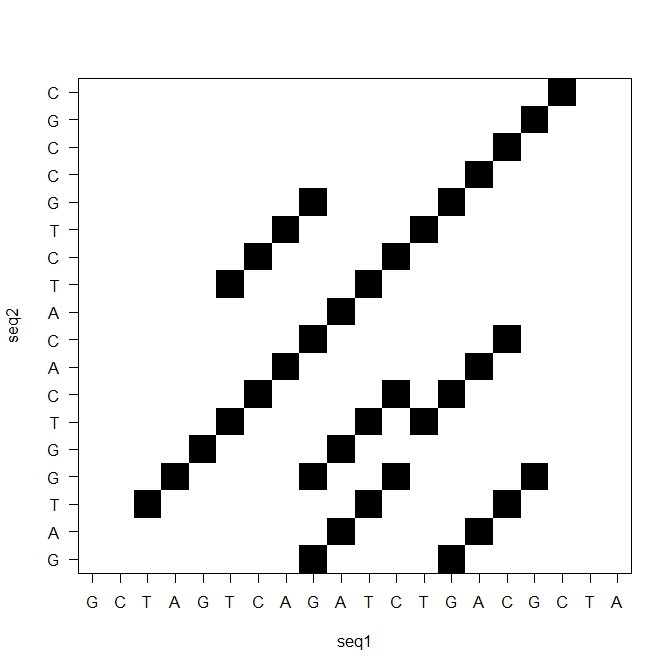
Короче говоря, я немного расширил функцию, чтобы получить полную матрицу между двумя последовательностями. Если вы проверяете выходной объект xyвы увидите исходную (type1) матрицу и расширенную (type2). Для очень длинных последовательностей эта модификация неэффективна, не говоря уже о том, что нуклеотидные / аминокислотные метки на осях будут перекрывать друг друга. Вы можете изменить тип графика между type1 и type2, используя новый аргумент type,
В качестве альтернативы для сравнения двух последовательностей, вы можете рассмотреть seqalign функция TraMineR R пакет.
Вот пример:
library(TraMineR)
seq1 <- c("G","C","T","A","G","T","C","A","G","A","T","C",
"T","G","A","C","G","C","T","A")
## Filling seq2 with "*" to equalize sequence length
seq2m <- c("G","A","T","G","G","T","C","A","C","A","T","C",
"T","G","C","C","G","C","*","*")
## defining sequence object interpreting "*"s as mising states
seq <- seqdef(rbind(seq1,seq2m), missing="*")
## Setting all substitution costs as 2
cost <- matrix(rep(2,16),4,4)
diag(cost) <- 0
cost
## Comparing sequences 1 and 2
sa <- seqalign(seq, 1:2, indel=1, sm=cost)
print(sa)
plot(sa)
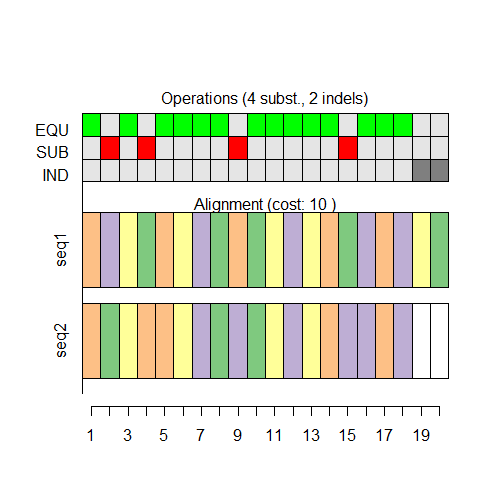
На графике показаны совпадающие элементы (EQU) и минимальные операции редактирования (SUB и IND), которые преобразуют одну последовательность в другую.