График принятия решения с помощью ggplot2?
Как мне построить эквивалент contour (база R) с ggplot2? Ниже приведен пример с анализом линейной дискриминантной функции:
require(MASS)
iris.lda<-lda(Species ~ Sepal.Length + Sepal.Width + Petal.Length + Petal.Width, data = iris)
datPred<-data.frame(Species=predict(iris.lda)$class,predict(iris.lda)$x) #create data.frame
#Base R plot
eqscplot(datPred[,2],datPred[,3],pch=as.double(datPred[,1]),col=as.double(datPred[,1])+1)
#Create decision boundaries
iris.lda2 <- lda(datPred[,2:3], datPred[,1])
x <- seq(min(datPred[,2]), max(datPred[,2]), length.out=30)
y <- seq(min(datPred[,3]), max(datPred[,3]), length.out=30)
Xcon <- matrix(c(rep(x,length(y)),
rep(y, rep(length(x), length(y)))),,2) #Set all possible pairs of x and y on a grid
iris.pr1 <- predict(iris.lda2, Xcon)$post[, c("setosa","versicolor")] %*% c(1,1) #posterior probabilities of a point belonging to each class
contour(x, y, matrix(iris.pr1, length(x), length(y)),
levels=0.5, add=T, lty=3,method="simple") #Plot contour lines in the base R plot
iris.pr2 <- predict(iris.lda2, Xcon)$post[, c("virginica","setosa")] %*% c(1,1)
contour(x, y, matrix(iris.pr2, length(x), length(y)),
levels=0.5, add=T, lty=3,method="simple")
#Eqivalent plot with ggplot2 but without decision boundaries
ggplot(datPred, aes(x=LD1, y=LD2, col=Species) ) +
geom_point(size = 3, aes(pch = Species))
Невозможно использовать матрицу при построении контурных линий с помощью ggplot. Матрица может быть переупорядочена во фрейме данных с использованием melt. В кадре данных ниже значения вероятности из iris.pr1 отображаются в первом столбце вместе с координатами x и y в следующих двух столбцах. Координаты x и y образуют сетку из 30 х 30 точек.
df <- transform(melt(matrix(iris.pr1, length(x), length(y))), x=x[X1], y=y[X2])[,-c(1,2)]
Я хотел бы построить координаты (предпочтительно связанные сглаженной кривой), где апостериорные вероятности равны 0,5 (т.е. границы решения).
2 ответа
Ты можешь использовать geom_contour в ggplot для достижения аналогичного эффекта. Как вы правильно предположили, вы должны преобразовать свои данные. Я в итоге просто делал
pr<-data.frame(x=rep(x, length(y)), y=rep(y, each=length(x)),
z1=as.vector(iris.pr1), z2=as.vector(iris.pr2))
И затем вы можете передать этот data.frame в geom_contour и указать, что вы хотите разрывы на 0,5 с
ggplot(datPred, aes(x=LD1, y=LD2) ) +
geom_point(size = 3, aes(pch = Species, col=Species)) +
geom_contour(data=pr, aes(x=x, y=y, z=z1), breaks=c(0,.5)) +
geom_contour(data=pr, aes(x=x, y=y, z=z2), breaks=c(0,.5))
и это дает
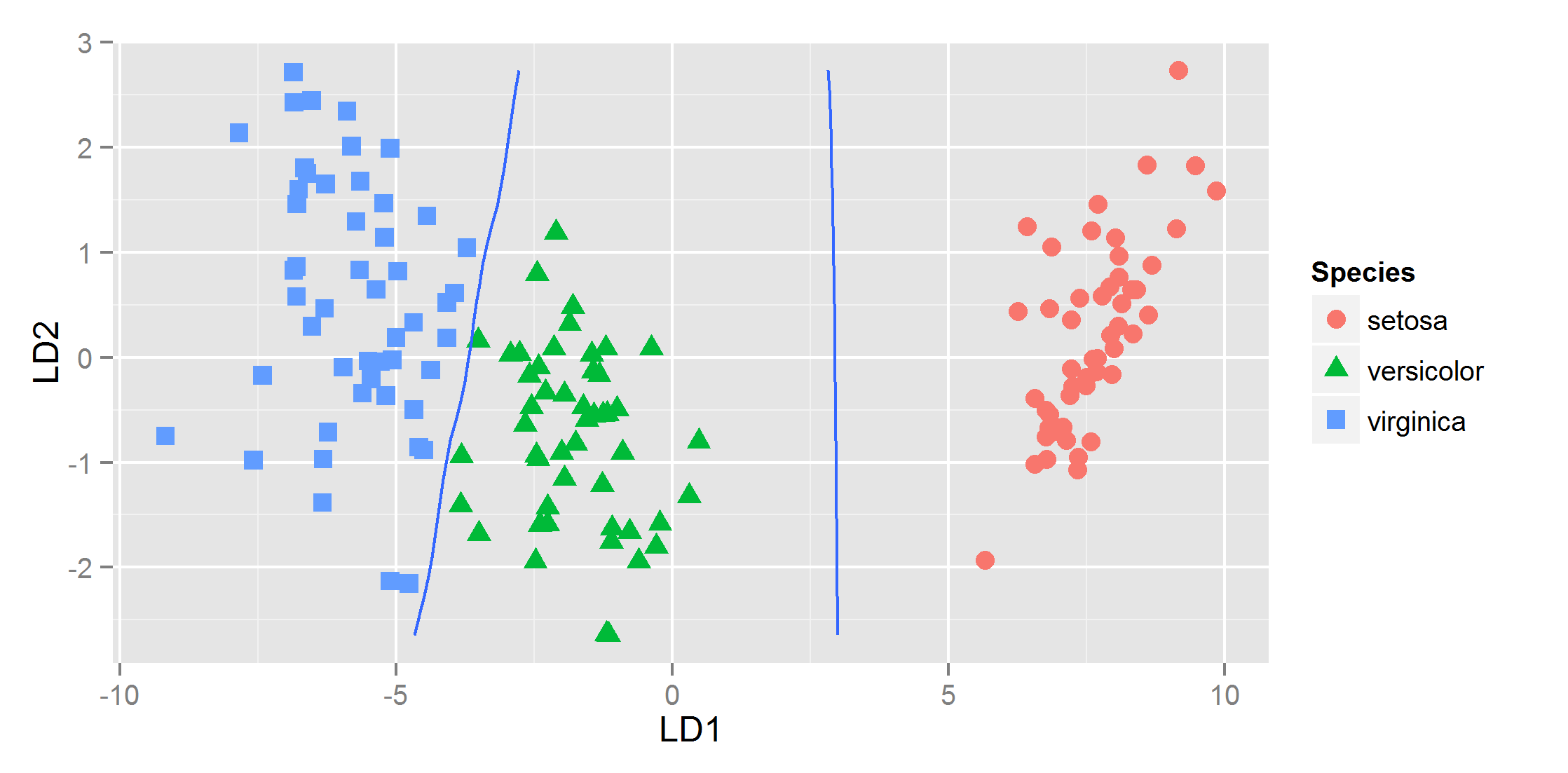
Функция partimat в библиотеке klaR делает то, что вы хотите для наблюдаемых предикторов, но если вы хотите то же самое для проекций LDA, вы можете построить фрейм данных, дополняющий оригинал проекциями LD1...LDk, а затем вызвать partimat с формулой Group~LD1+...+LDk, method='lda' - тогда вы видите "плоскость LD", которую вы намеревались увидеть, приятно разделенную для вас. Мне это показалось легче, по крайней мере, объяснить учащимся, более новым для R, поскольку я просто повторно использую функцию, уже предоставленную таким образом, что она не совсем предназначена.