Tidyverse/ более быстрое решение для условного форматирования с openxlsx в R?
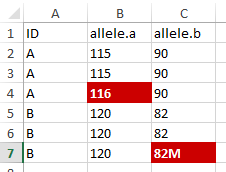
Я работаю с генетическими данными, которые выглядят как эта таблица, но больше:
ID allele.a allele.b
A 115 90
A 115 90
A 116 90
B 120 82
B 120 82
B 120 82M
Моя цель - выделить для каждого идентификатора, какие аллели не соответствуют аллелям, перечисленным в первом ряду каждой группы идентификаторов. Мне нужно экспортировать данные в красиво отформатированный файл Excel.
Вот что я хочу:
Я могу получить его с помощью следующего сценария, но в действительности он включает около 67 идентификаторов, 1000 строк данных и 37 столбцов. Запуск занимает около 5 минут, поэтому я надеюсь найти решение, которое значительно сокращает время обработки. Может быть, решение "сделать" из Tidyverse - не уверен, как это будет выглядеть.
Вот мой скрипт, в том числе тестовый data.frame. Также включает в себя больший тест data.frame для тестирования скорости.
library(xlsx)
library(openxlsx)
library(tidyverse)
# Small data.frame
dframe <- data.frame(ID = c("A", "A", "A", "B", "B", "B"),
allele.a = c("115", "115", "116", "120", "120", "120"),
allele.b = c("90", "90", "90", "82", "82", "82M"),
stringsAsFactors = F)
# Bigger data.frame for speed test
# dframe <- data.frame(ID = rep(letters, each = 30),
# allele.a = rep(as.character(round(rnorm(n = 30, mean = 100, sd = 0.3), 0)), 26),
# allele.b = rep(as.character(round(rnorm(n = 30, mean = 90, sd = 0.3), 0)), 26),
# allele.c = rep(as.character(round(rnorm(n = 30, mean = 80, sd = 0.3), 0)), 26),
# allele.d = rep(as.character(round(rnorm(n = 30, mean = 70, sd = 0.3), 0)), 26),
# allele.e = rep(as.character(round(rnorm(n = 30, mean = 60, sd = 0.3), 0)), 26),
# allele.f = rep(as.character(round(rnorm(n = 30, mean = 50, sd = 0.3), 0)), 26),
# allele.g = rep(as.character(round(rnorm(n = 30, mean = 40, sd = 0.3), 0)), 26),
# allele.h = rep(as.character(round(rnorm(n = 30, mean = 30, sd = 0.3), 0)), 26),
# allele.i = rep(as.character(round(rnorm(n = 30, mean = 20, sd = 0.3), 0)), 26),
# allele.j = rep(as.character(round(rnorm(n = 30, mean = 10, sd = 0.3), 0)), 26),
# stringsAsFactors = F)
# Create a new excel workbook ----
wb <- createWorkbook()
# Add a worksheets
addWorksheet(wb, sheet = 1, gridLines = TRUE)
# add the data to the worksheet
writeData(wb, sheet = 1, dframe, rowNames = FALSE)
# Create a style to show alleles that do not match the first row.
style_Red_NoMatch <- createStyle(fontColour = "#FFFFFF", # white text
bgFill = "#CC0000", # Dark red background
textDecoration = c("BOLD")) # bold text
Groups <- unique(dframe$ID)
start_time <- Sys.time()
# For each unique group,
for(i in 1:length(Groups)){
# Print a message telling us where the script is processing in the file.
print(paste("Formatting unique group ", i, "/", length(Groups), sep = ""))
# What are the allele values of the *first* individual in the group?
Allele.values <- dframe %>%
filter(ID == Groups[i]) %>%
slice(1) %>%
select(2:ncol(dframe)) %>%
as.character()
# for each column that has allele values in it,
for (j in 1:length(Allele.values)){
# format the rest of the rows so that a value that does not match the first value gets red style
conditionalFormatting(wb, sheet = 1,
style_Red_NoMatch,
rows = (which(dframe$ID == Groups[i]) + 1),
cols = 1+j, rule=paste("<>\"", Allele.values[j], "\"", sep = ""))
}
}
end_time <- Sys.time()
end_time - start_time
saveWorkbook(wb, "Example.xlsx", overwrite = TRUE)
1 ответ
Я предполагаю, что один из способов улучшить - применить ко всему столбцу вместо того, чтобы перебирать каждую ячейку.
Вот один способ. Одним из недостатков этого подхода является то, что он создает логический вектор
TRUE и
FALSE который используется для
conditionalFormatting. Эти столбцы можно скрыть с помощью
setColWidths функция.
Данные
library(openxlsx)
dframe <- data.frame(ID = rep(letters, each = 30),
allele.a = rep(as.character(round(rnorm(n = 30, mean = 100, sd = 0.3), 0)), 26),
allele.b = rep(as.character(round(rnorm(n = 30, mean = 90, sd = 0.3), 0)), 26),
allele.c = rep(as.character(round(rnorm(n = 30, mean = 80, sd = 0.3), 0)), 26),
allele.d = rep(as.character(round(rnorm(n = 30, mean = 70, sd = 0.3), 0)), 26),
allele.e = rep(as.character(round(rnorm(n = 30, mean = 60, sd = 0.3), 0)), 26),
allele.f = rep(as.character(round(rnorm(n = 30, mean = 50, sd = 0.3), 0)), 26),
allele.g = rep(as.character(round(rnorm(n = 30, mean = 40, sd = 0.3), 0)), 26),
allele.h = rep(as.character(round(rnorm(n = 30, mean = 30, sd = 0.3), 0)), 26),
allele.i = rep(as.character(round(rnorm(n = 30, mean = 20, sd = 0.3), 0)), 26),
allele.j = rep(as.character(round(rnorm(n = 30, mean = 10, sd = 0.3), 0)), 26),
stringsAsFactors = F)
Первая часть сценария без изменений.
# Create a new excel workbook ----
wb <- createWorkbook()
# Add a worksheets
addWorksheet(wb, sheet = 1, gridLines = TRUE)
# Create a style to show alleles that do not match the first row.
style_Red_NoMatch <- createStyle(fontColour = "#FFFFFF", # white text
bgFill = "#CC0000", # Dark red background
textDecoration = c("BOLD")) # bold text
Затем определите первую строку для каждого идентификатора и объедините с исходным набором данных. Затем проверьте, есть ли какие-либо изменения в каких-либо ячейках (проходит через каждый столбец).
# selects first row for each ID which will be used as benchmark
first_row <- dframe[!duplicated(dframe$ID), ]
# Creating new df with the first_row columns added
dframe_chk <- merge(dframe, first_row, by = "ID", all.x = TRUE, suffixes = c("", "_first"))
# Adding TRUE/FALSE factor for each column to see if it matches or not (-1 to exclude ID column)
for (j in names(dframe)[-1]) {
dframe_chk[, paste0(j, "_chk")] <- dframe_chk[, j] == dframe_chk[, paste0(j, "_first")]
}
# Remove _first columns when exporting into Excel
cols <- names(dframe_chk)[!grepl("_first", names(dframe_chk))]
# add the data to the worksheet
writeData(wb, sheet = 1, dframe_chk[, cols], rowNames = FALSE)
# This is for conditional Formatting
# first_row is header
row_start <- 2
# Need to add 1 to cover full range (as first row is header)
row_end <- nrow(dframe) + 1
# first column is ID
col_start <- 2
# last column as per the original dataset
col_end <- ncol(dframe)
# this is to point to the _chk column.
# Note if you have columns more than A-Z then this needs to be adjusted
rule_col <- LETTERS[col_end + 1]
# Using the _chk column to apply conditional formula
conditionalFormatting(wb, sheet = 1,
style_Red_NoMatch,
rows = row_start:row_end,
cols = col_start:col_end,
rule = paste0(rule_col, "2 = FALSE"))
# Exported file includes _chk columns. Hide these columns.
setColWidths(wb, sheet = 1, cols = (col_end + 1):length(cols), hidden = TRUE)
saveWorkbook(wb, "Example2.xlsx", overwrite = TRUE)