Почему Python вызывает ошибку во время выполнения только с определенными изображениями?
Это третий вопрос, который я задаю в stackru по этому поводу, потому что каждый раз, когда я получаю некоторые изменения в том, как Python вызывает ошибку времени выполнения.
Предыдущие вопросы были: здесь и здесь. В первом вопросе я думаю, что это вопрос памяти, потому что я anylized много изображений, во втором случае ошибки времени выполнения происходят в этой строке
p2 = numpy.percentile(img, 2)
и я думаю, что это была проблема с модульным модулем.
но теперь я во время выполнения ошибки происходит здесь:
imgbnbin = mh.morph.dilate(gray, disk7)
При махотах функция расширяется.
единственные три изображения, которые у меня есть среди 90 изображений, это:



и это 2 примера изображения, где код работает нормально:
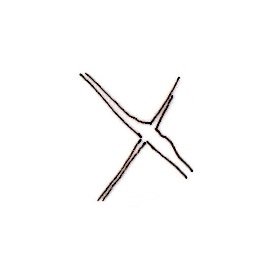
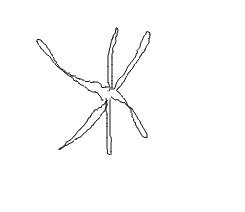
Ниже приведен код моей функции skelfeatures, где я получил ошибку времени выполнения:
import os
import glob
import scipy
import numpy as np
import pymorph as pm
import pylab as plb
import matplotlib
from matplotlib import pyplot as plt
import cv2
import mahotas as mh
from skimage import morphology
from skimage import io
from math import sqrt
from skimage import data, img_as_float
from skimage import exposure
from skimage import color
from skimage import io, filter
from skimage.morphology import erosion, dilation, opening, closing, white_tophat
from skimage.morphology import black_tophat, skeletonize, convex_hull_image
from skimage.morphology import disk
def plot_img_and_hist(img, axes, bins=256):
"""Plot an image along with its histogram and cumulative histogram.
"""
img = img_as_float(img)
ax_img, ax_hist = axes
ax_cdf = ax_hist.twinx()
# Display image
ax_img.imshow(img, cmap=plt.cm.gray)
ax_img.set_axis_off()
# Display histogram
ax_hist.hist(img.ravel(), bins=bins, histtype='step', color='black')
ax_hist.ticklabel_format(axis='y', style='scientific', scilimits=(0, 0))
ax_hist.set_xlabel('Pixel intensity')
ax_hist.set_xlim(0, 1)
ax_hist.set_yticks([])
# Display cumulative distribution
img_cdf, bins = exposure.cumulative_distribution(img, bins)
ax_cdf.plot(bins, img_cdf, 'r')
ax_cdf.set_yticks([])
return ax_img, ax_hist, ax_cdf
import urllib, cStringIO
listarough = list()
def skelfeatures(path):
import copy
if (path[0] == "h"):
#URL
req = urllib.urlopen(path) #cv2.imdecode(arr,0) # load as grayscale
arr = np.asarray(bytearray(req.read()), dtype=np.uint8)
gray = cv2.imdecode(arr,0) # 'load it as it is'
#cv2.imshow('lalala',gray)
#if cv2.waitKey() & 0xff == 27: quit()
else:
#local path
print("prima di cv2imread")
gray = cv2.imread(path,0)
stampac = "no"
originale = copy.copy(gray)
oshape = list(gray.shape)
if (oshape[0] <= 140):
#print(oshape[0]/100)
ow = int ((oshape[0]/100 )*2.5 )
oh = int ((oshape[0]/100 )*2.5 )
#print("ow ",ow)
elif (oshape[0] <= 300):
#print(oshape[0]/100)
ow = int ((oshape[0]/100 )*3.5 )
oh = int ((oshape[0]/100 )*3.5 )
#print("ow ",ow)
else:
ow = int ((oshape[0]/100 )*7 )
oh = int ((oshape[0]/100 )*7 )
owclose = ow * 2
element = cv2.getStructuringElement(cv2.MORPH_CROSS,(ow,oh))
graydilate = cv2.erode(gray, element) #imgbnbin
ret,thresh = cv2.threshold(gray,127,255,cv2.THRESH_BINARY_INV)
gray = thresh
proxMax = int((oshape[0]/100 )*22)
proxMin = int((oshape[0]/100 )*19)
proxGE = 5
proxGEmin = 2
oshape = list(gray.shape)
if (oshape[0] <= 140):
#print(oshape[0]/100)
ow = int ((oshape[0]/100 )*2.5 )
oh = int ((oshape[0]/100 )*2.5 )
#print("ow ",ow)
elif (oshape[0] <= 300):
#print(oshape[0]/100)
ow = int ((oshape[0]/100 )*3.5 )
oh = int ((oshape[0]/100 )*3.5 )
#print("ow ",ow)
else:
ow = int ((oshape[0]/100 )*7 )
oh = int ((oshape[0]/100 )*7 )
#print(ow)
owclose = ow * 2
disko = pm.sedisk(0.2)
imgbnbin7 = gray
############################################################
# OPEN ImAGE PATH
############################################################
print("mahotas")
img = color.rgb2gray(io.imread(path))
print(type(img))
print("FINE ioimread")
from skimage import exposure
#print dir(exposure)
# Contrast stretching
###########################################################################
# THE FIRST TIME WHERE I GOT RUNTIME ERROR, the following line
###########################################################################
p2 = np.percentile(img, 2)
p98 = np.percentile(img, 98)
img_rescale = exposure.rescale_intensity(img, out_range=(0, 1))
# Equalization
img_eq = exposure.equalize_hist(img)
# Adaptive Equalization
img_adapteq = exposure.equalize_adapthist(img, clip_limit=0.03)
# Display results
'''f, axes = plt.subplots(2, 4, figsize=(8, 4))
ax_img, ax_hist, ax_cdf = plot_img_and_hist(img, axes[:, 0])
ax_img.set_title('Low contrast image')
y_min, y_max = ax_hist.get_ylim()
ax_hist.set_ylabel('Number of pixels')
ax_hist.set_yticks(np.linspace(0, y_max, 5))
ax_img, ax_hist, ax_cdf = plot_img_and_hist(img_rescale, axes[:, 1])
ax_img.set_title('Contrast stretching')
ax_img, ax_hist, ax_cdf = plot_img_and_hist(img_eq, axes[:, 2])
ax_img.set_title('Histogram equalization')
ax_img, ax_hist, ax_cdf = plot_img_and_hist(img_adapteq, axes[:, 3])
ax_img.set_title('Adaptive equalization')
ax_cdf.set_ylabel('Fraction of total intensity')
ax_cdf.set_yticks(np.linspace(0, 1, 5))
#prevent overlap of y-axis labels
plt.subplots_adjust(wspace=0.4)
plt.show()'''
if ( stampac =="no" ):
plt.gray()
plt.subplot(121)
plt.title("dopo histo")
plt.imshow(img)
plt.show()
binimg = img_adapteq
if ( stampac =="no" ):
plt.gray()
plt.subplot(121)
plt.title("dopo conversione")
plt.imshow(binimg)
plt.show()
binimgafter = copy.copy(binimg)
threshold = filter.threshold_otsu(img_rescale)
gray =( img_rescale< threshold)
if ( stampac =="si" ):
plt.gray()
plt.subplot(121)
plt.title("dopo otsu")
plt.imshow(gray)
plt.show()
shape = gray.shape
w = 0
if (shape[0] > shape[1]):
shape = shape[0]
else:
shape = shape[1]
if (shape < 100):
w = int((shape/100 )*1.5)
elif(shape > 100 and shape <420):
w = int((shape/100 )*2.5)
else:
w = int((shape/100)*4)
disk7 = pm.sedisk(w)
print("bau2")
imgbnbin = mh.morph.dilate(gray, disk7)
if ( stampac =="no" ): #2
plt.gray()
plt.subplot(121)
plt.title("dopo dilate prima di close")
plt.imshow(imgbnbin)
plt.show()
########################################################
#RUNTIME ERROR HER. After that python does not show bau3
###########################################################
print("bau3")
imgbnbin = mh.morph.close(imgbnbin, disko)
if ( stampac =="no" ): #2
plt.gray()
plt.subplot(121)
plt.title("dopo close prima di skeletonize")
plt.imshow(imgbnbin)
plt.show()
out = morphology.skeletonize(imgbnbin>0)
# the function continue...
path = "https://stackru.com/images/4f76e8bb6e746856c53ccf43b34c82be96306353.jpg"