Как нормализовать оценку плотности ядра с помощью scikit?
Я использую KDE для классификации нескольких классов. Я реализую это с помощью Scikit. Как упоминалось на веб-сайте, KDE для точки x определяется как
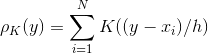
Должен ли я нормализовать результат при сравнении разных оценок плотности ядра для разных классов?
Ссылка для KDE:
http://scikit-learn.org/stable/modules/density.html
1 ответ
Равенство не имеет места, это явно плохой пример документации. Вы можете увидеть в коде, что он нормализован, как здесь
log_density -= np.log(N)
return log_density
так что вы четко делите на N,
Правильная формула с математической точки зрения на самом деле либо
1/N SUM_i K(x_i - x)
или же
1/(hN) SUM_i K((x_i - x)/h)
вы также можете глубже погрузиться в код.c, фактически вычисляющий ядра, и вы обнаружите, что они внутренне нормализованы
case __pyx_e_7sklearn_9neighbors_9ball_tree_GAUSSIAN_KERNEL:
/* "binary_tree.pxi":475
* cdef ITYPE_t k
* if kernel == GAUSSIAN_KERNEL:
* factor = 0.5 * d * LOG_2PI # <<<<<<<<<<<<<<
* elif kernel == TOPHAT_KERNEL:
* factor = logVn(d)
*/
__pyx_v_factor = ((0.5 * __pyx_v_d) * __pyx_v_7sklearn_9neighbors_9ball_tree_LOG_2PI);
break;
Таким образом, каждый K на самом деле интегрируется в 1 и, следовательно, вы просто берете среднее значение, чтобы получить действительную плотность для всего KDE, и это именно то, что происходит внутри.