Как настроить ось Y с помощью seqIplot в R?
Я пытаюсь настроить ось Y графика индекса последовательности, используя R-пакет TraMineRЯ выяснил, как установить глобальный предел для оси Y, что полезно, если вы хотите сравнить количество определенных последовательностей между двумя или более группами, поскольку оно выравнивает масштаб. Но мне не удалось установить галочки по оси Y (как в xtstep). Может быть, вы можете быстро помочь мне, используя этот пример кода:
library(TraMineR)
data(mvad)
mvad.alphabet <- c("employment", "FE", "HE", "joblessness", "school",
"training")
mvad.labels <- c("Employment", "Further Education", "Higher Education",
"Joblessness", "School", "Training")
mvad.scodes <- c("EM", "FE", "HE", "JL", "SC", "TR")
## Define sequence objects
mvad.seq <- seqdef(mvad[, 17:86], alphabet = mvad.alphabet,
states = mvad.scodes, labels = mvad.labels, weights = mvad$weight, xtstep = 6)
## Plots
seqIplot(mvad.seq, group=mvad$gcse5eq, withlegend=TRUE, border=NA, xtstep=3, sortv="from.start") ## Default plot
seqIplot(mvad.seq, group=mvad$gcse5eq, withlegend=TRUE, border=NA, xtstep=3, sortv="from.start", ylim=c(0, 400)) ## Plot with custom ylim to compare the number of sequences between groups
График индекса последовательности по умолчанию выглядит следующим образом и очень затрудняет сравнение двух групп: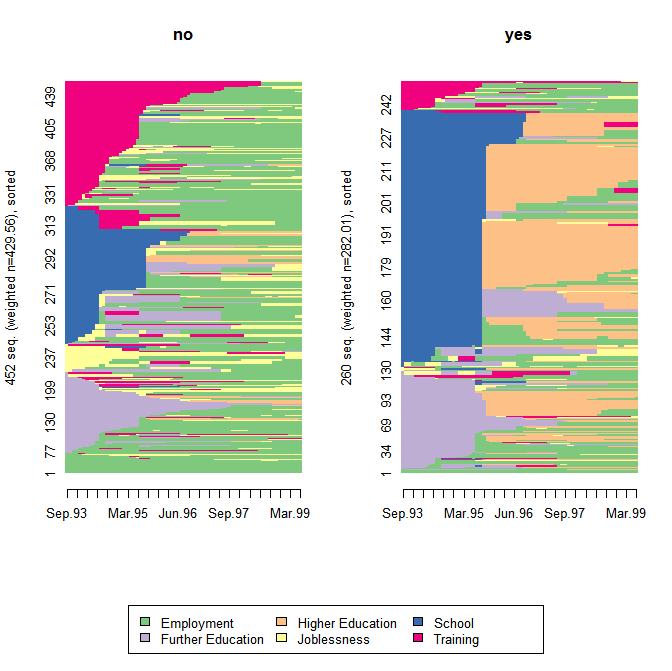
1 ответ
Когда ylim дан в seqIplot, он используется для всех групп. Чтобы сделать высоту графика пропорциональной взвешенному количеству последовательностей в каждой группе, верхний ylim должно быть установлено в качестве значения для наиболее частой группы.
group <- mvad$gcse5eq
(nseq <- xtabs(mvad$weight ~ group))
(nmax <- max(nseq))
seqIplot(mvad.seq, group=group, withlegend=TRUE,
border=NA, xtstep=3, sortv="from.start",
ylim=c(0, nmax) )
Метки галочек на оси Y являются индексами последовательности. Вы можете подавить их, дав yaxis = FALSE в seqIplot, Чтобы отобразить свои собственные ярлыки, вы можете выпустить что-то вроде (см. Помощь axisфункция для деталей)
axis(2, at = c(1, nseq[1]))
но в этом случае вы должны сгенерировать seqIplot отдельно для каждой группы с withlegend=FALSE и организовать графики самостоятельно в одном графике с layout или же par(mfrow=...),