Построение числа функций в EBImage в R
У меня есть вопрос, касающийся функции computeFeatures в EBImage. Я использовал следующий код для вычисления функций.
biocLite("EBImage")
library (EBImage)
Image <- readImage("test.jpg")
Image3<-getFrame(Image,3)
x = thresh(Image3, w=15, h=15, offset=0.05)
x = opening(x, makeBrush(5, shape='disc'))
x = bwlabel(x)
fts = computeFeatures.shape(x)
Это дает мне список вычисленных функций. Тем не менее, это список из 39 функций, и меня интересуют только некоторые из них. Таким образом, я хотел бы знать, какие из computeFeatures соответствуют каждой функции в изображении.
Решение было представлено по адресу http://www.bricol.net/research/leafranks/11-01MS/EBImage-introduction.pdf с использованием следующего кода:
> nmask = thresh(nuc, 10, 10, 0.05)
> nmask = opening(nmask, makeBrush(5, shape='disc'))
> nmask = fillHull(nmask)
> nmask = bwlabel(nmask) Cell bodies are segmented using propagate.
> ctmask = opening(cel>0.1, makeBrush(5, shape='disc'))
> cmask = propagate(cel, nmask, ctmask)
> res = paintObjects(cmask, img, col='#ff00ff')
> res = paintObjects(nmask, res, col='#ffff00')
> xy = lapply(hullFeatures(cmask), function(hf) hf[, c('g.x', 'g.y')])
> labels = lapply(xy, function(z) as.character(1:nrow(z)))
> font = drawfont(weight=600, size=16)
> res = drawtext(res, xy=xy, labels=labels , font=font, col="white")
Которые наносят числа, соответствующие функциям на изображении. Однако функция hullFeatures больше не доступна.
Таким образом, есть ли другой способ нанести на график количество объектов или объектов в изображении?
0 ответов
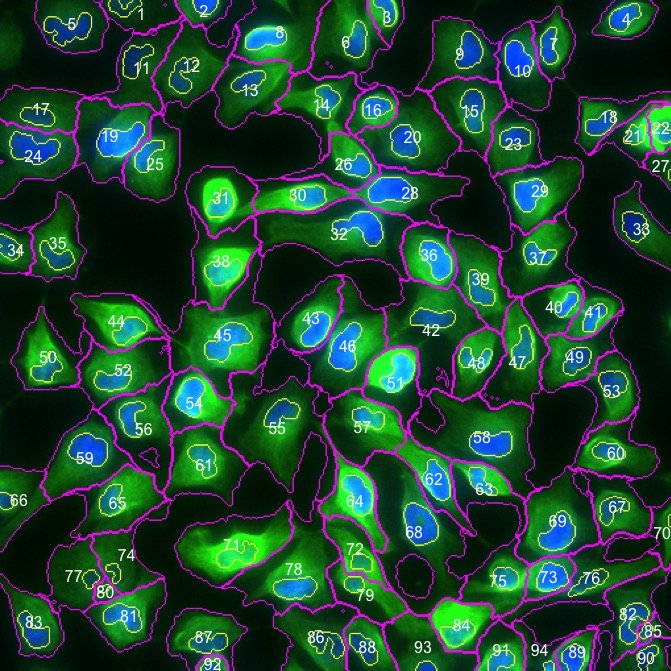
Функции hullFeatures а также drawtext несущественны hullFeatures был заменен computeFeatures.shape, Теперь нужно использовать computeFeatures.moment найти положение каждого объекта. База text теперь функция используется для добавления текста к изображению в растровом режиме.
Отличный обзор EBImage можно найти в виньетке: vignette("EBImage-introduction", package = "EBImage"), Однако в версии 4.22.1 виньетка больше не показывает, как нумеровать сегментированные объекты. Вот воссоздание примера из PDF от 22 апреля 2010 года Пау, Скляра и Хубера.
Чтобы немного уточнить номенклатуру, computeFeatures семейство функций возвращает матрицу размерности n x p где n количество сегментированных объектов (строк) и p количество объектов (столбцов). В этом примере каждый объект является ячейкой. Функции, возвращаемые computeFeatures.moment являются m.cx, m.cy, m.majoraxis, m.eccentricity а также m.theta, Положение определяется первыми двумя признаками.
# Starting from EBImage
if (!require(EBImage)) {
source("http://bioconductor.org/biocLite.R")
biocLite("EBImage")
library(EBImage)
}
# Use example nuclei and cells, make colorized composite
nuc <- readImage(system.file('images', 'nuclei.tif', package='EBImage'))
cel <- readImage(system.file('images', 'cells.tif', package='EBImage'))
img <- rgbImage(green=1.5*cel, blue=nuc)
# Recreate nuclear and cell masks, add border to color composite
nmask <- thresh(nuc, 10, 10, 0.05)
nmask <- opening(nmask, makeBrush(5, shape='disc'))
nmask <- fillHull(nmask)
nmask <- bwlabel(nmask)
ctmask <- opening(cel>0.1, makeBrush(5, shape='disc'))
cmask <- propagate(cel, nmask, ctmask)
res <- paintObjects(cmask, img, col='#ff00ff')
res <- paintObjects(nmask, res, col='#ffff00')
# Determine position (center of mass) of each object in 'cmask'
M <- apply(cmask, 3, computeFeatures.moment) # list of length 4 returned
xy <- lapply(M, function(m) m[,c("m.cx", "m.cy")])
# Create labels and plot in 'raster' mode
labels <- lapply(xy, function(z) as.character(1:nrow(z)))
plot(getFrame(res, 3, type = "render")) # or plot(res[,,,3])
# Add labels by text function
text(xy[[3]], labels[[3]], col = "white")