Как построить байесовскую сеть с созданными узлами с помощью bnlearn и graphviz?
Я пытаюсь построить байесовскую сеть (BN) с созданными узлами, используя библиотеки.
bnlearn а также
Rgraphviz. Мой рабочий процесс выглядит следующим образом:
После создания фрейма данных со случайными данными (данные, которые я на самом деле использую, очевидно, не случайны), я затем дискретизирую данные, изучаю структуру направленного ациклического графа (DAG), помещаю данные в DAG и затем строю DAG. Я также рисую DAG, который показывает апостериорные вероятности каждого из узлов.
#rm(list = ls())
library(bnlearn)
library(Rgraphviz)
# Generating random dataframe
data_clean <- data.frame(a = runif(min = 0, max = 100, n = 1000),
b = runif(min = 0, max = 100, n = 1000),
c = runif(min = 0, max = 100, n = 1000),
d = runif(min = 0, max = 100, n = 1000),
e = runif(min = 0, max = 100, n = 1000))
# Discretising the data into 3 bins
bins <- 3
data_discrete <- discretize(data_clean, breaks = bins)
# Creating factors for each bin in the data
lv <- c("low", "med", "high")
for (i in names(data_discrete)){
levels(data_discrete[, i]) = lv
}
# Structure learning the DAG from the training set
whitelist <- matrix(c("a", "b",
"b", "c",
"c", "e",
"a", "d",
"d", "e"),
ncol = 2, byrow = TRUE, dimnames = list(NULL, c("from", "to")))
bn.hc <- hc(data_discrete, whitelist = whitelist)
# Plotting the DAG
dag.hc <- graphviz.plot(bn.hc,
layout = "dot")
# Fitting the data to the structure
fitted <- bn.fit(bn.hc, data = data_discrete, method = "bayes")
# Plotting the DAG with posteriors
graphviz.chart(fitted, type = "barprob", layout = "dot")
Следующее, что я делаю, это вручную изменяю распределения в объекте, назначенном для
fitted, а затем постройте DAG, который показывает экземпляры узлов и обновленную апостериорную вероятность переменной ответа
e.
# Manually instantiating
fitted_evidence <- fitted
cpt.a = matrix(c(1, 0, 0), ncol = 3, dimnames = list(NULL, lv))
cpt.c = c(1, 0, 0,
0, 1, 0,
0, 0, 1)
dim(cpt.c) <- c(3, 3)
dimnames(cpt.c) <- list("c" = lv, "b" = lv)
cpt.b = c(1, 0, 0,
0, 1, 0,
0, 0, 1)
dim(cpt.b) <- c(3, 3)
dimnames(cpt.b) <- list("b" = lv, "a" = lv)
cpt.d = c(0, 0, 1,
0, 1, 0,
1, 0, 0)
dim(cpt.d) <- c(3, 3)
dimnames(cpt.d) <- list("d" = lv, "a" = lv)
fitted_evidence$a <- cpt.a
fitted_evidence$b <- cpt.b
fitted_evidence$c <- cpt.c
fitted_evidence$d <- cpt.d
# Plotting the DAG with instantiation and posterior for response
graphviz.chart(fitted_evidence, type = "barprob", layout = "dot")
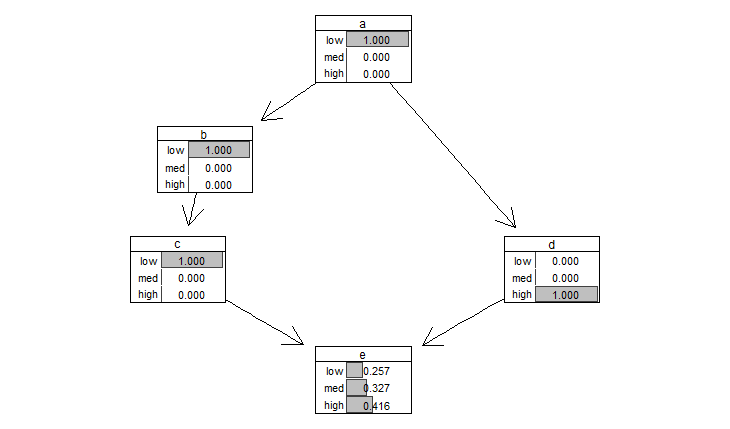
Это результат, который я получаю, но мой фактический BN намного больше, с большим количеством дуг, и было бы непрактично вручную изменять объект.
Я хотел бы узнать, есть ли способ построить DAG с созданием экземпляра без изменения
bn.fitобъект вручную? Есть ли обходной путь или функция, которые мне не хватает?
Я думаю / надеюсь, что внимательно прочитал документацию по bnlearn. Я ценю любые отзывы и буду рад изменить что-либо в вопросе, если я не передал свои мысли достаточно ясно.
Спасибо.
1 ответ
Как насчет использования для извлечения образцов из апостериорной области, учитывая доказательства. Затем вы можете оценить обновленные параметры, используя
bn.fit с помощью
cpdistобразцы. Затем заговорите, как прежде.
Пример:
set.seed(69184390) # for sampling
# Your evidence vector
ev <- list(a = "low", b="low", c="low", d="high")
# draw samples
updated_dat <- cpdist(fitted, nodes=bnlearn::nodes(fitted), evidence=ev, method="lw", n=1e6)
# refit : you'll get warnings over missing levels
updated_fit <- bn.fit(bn.hc, data = updated_dat)
# plot
par(mar=rep(0,4))
graphviz.chart(updated_fit, type = "barprob", layout = "dot")
Обратите внимание, я использовал
bnlearn::nodes в качестве
nodes маскируется зависимостью
Rgraphviz. Я склонен загружать
bnlearn последний.